「RELIONチュートリアル」の版間の差分
(→3.3 CTF estimation(CTF推定)) |
|||
| (7人の利用者による、間の335版が非表示) | |||
| 行1: | 行1: | ||
| + | '''前書き''' | ||
| + | |||
| + | ここに示したものは、Relion2のTutorialをDr. Scheresに許可を得て、日本語訳を行ったものを元にし、それに注釈等を加えたものです。誤訳等がある場合には、ご連絡頂けますと幸いです。少しずつ、良いものにしたいと思います。 | ||
| + | |||
| + | @http://www.yasunaga-lab.bio.kyutech.ac.jp/EosJ/index.php?title=RELIONチュートリアル | ||
| + | |||
== 1 Getting prepared(準備) == | == 1 Getting prepared(準備) == | ||
| − | === 1.1 Recommended reading( | + | |
| − | + | RELIONのインストールに関わる他の情報は下記を参考にして下さい。 | |
| + | |||
| + | [[http://www.yasunaga-lab.bio.kyutech.ac.jp/EosJ/index.php/RELION RELION]] | ||
| + | |||
| + | |||
| + | === 1.1 Recommended reading(参考文献) === | ||
| + | RELIONの精密化処理に関する理論的詳細は、以下の論文で述べられています。 | ||
●S.H.W. Scheres (2012) "RELION: Implementation of a Bayesian approach to cryo-EM structure determination" ''J.Struc. Biol.'', 180, 519-530. | ●S.H.W. Scheres (2012) "RELION: Implementation of a Bayesian approach to cryo-EM structure determination" ''J.Struc. Biol.'', 180, 519-530. | ||
| 行8: | 行20: | ||
| − | + | RELIONの使い方に関する、包括的な概要は以下の論文が参考になります。 | |
●S.H.W. Scheres (2016) "Processing of structurally heterogeneous cryo-EM data in RELION" ''Meth. Enzym.'', in press. | ●S.H.W. Scheres (2016) "Processing of structurally heterogeneous cryo-EM data in RELION" ''Meth. Enzym.'', in press. | ||
| − | === 1.2 Install MPI === | + | === 1.2 Install MPI(MPIのインストール) === |
| − | MPI(massage passing interface) | + | 計算量が多いため、並列処理による高速化のために[[MPI(massage passing interface)]]を利用しています。MPIがインストールされた計算機クラスタ、もしくは、NVIDIA GPU搭載のマルチコアデスクトップマシンが必要になることに注意してください。RELIONをコンパイルするには、mpi-develパッケージが必要です。まだmpi-develがインストールされていない場合は、openMPI([[http://www.open-mpi.org http://www.open-mpi.org]])をインストールすることをお勧めしますが、恐らく、その種類(openMPI、MPICH、LAM-MPIなど)またはバージョンはそれほど重要ではありません。 |
| − | + | ※ MPIは、マルチプロセスの並列処理により高速化を行うための共通プロトコルの一つです。 | |
| − | + | ||
| − | === 1. | + | === 1.3 Install CUDA(CUDAのインストール) === |
| − | + | NVIDIAの比較的新しいGPU(compute capability 3.5+以上)をお持ちの場合、オートピッキング、分類、精密化作業を高速化することができます。GPUアクセラレータをサポートするRELIONをコンパイルするには、CUDAをインストールする必要があります。このチュートリアルの準備の際には、CUDA-7.5を使用しました。 NVIDIAの[https://developer.nvidia.com/cuda-downloads Webサイト]からダウンロードしてください。 | |
| − | + | ※ CUDAは、NVIDIA社が開発した、GPU(グラフィカル・プロセッシング・ユニット)を用いて、マルチスレッドの並列計算を行うためのライブラリです。 | |
| − | + | ||
| − | === 1. | + | === 1.4 Install RELION(RELIONのインストール) === |
| − | + | RELIONはオープンソースのソフトウェアです。[http://www2.mrc-lmb.cam.ac.uk/relion/index.php/Download_%26_install the RELION wiki]から無料でダウンロードして、手順に従ってインストールしてください。 | |
| − | + | もしジョブ投函システム(Sun Grid EngineやPBS/TORQUEなど、ジョブの順次処理(ジョブキューイングシステム)に詳しくない場合は、インストール手順の説明にあるように、qsub.cshスクリプトの設定に関して、システム管理者に助けを求めて下さい。 | |
| − | + | ||
| − | === 1.8 Download the test data === | + | 皆さんは、分散メモリ並列化のためのMPIと、共用メモリの並列化のためのpthreadsの両方を使用する、いわゆるハイブリッド並列計算を実行したいと考えるでしょう。その場合、ジョブ投函キューシステムは、これを可能にするためにいくつかの調整が必要な場合があります。その場合は再度、システム管理者に問い合わせてください。 |
| − | このチュートリアルでは、さまざまなベータガラクトシダーゼの再構成を計算するために使用される顕微鏡写真のサブセットであるテストデータセットを使用しています[13,2,17] | + | |
| + | ※ ジョブキューイングシステム(job submission queueing system) | ||
| + | キュー(待ち行列)とは、実行したいと考えるジョブを投函した後、投函されたものから順番に実行するためのシステムです。一台のホストを大人数で使用する場合などには、一度に利用するとホストのメモリやCPU が足りなくなり、利用できなくなったり、異常に遅くなったりします。そのため、計算機リソースを順番に使うために利用されています。一台を1人で使う場合などには余り考える必要はありません。 | ||
| + | |||
| + | ※プロセスとスレッド | ||
| + | プロセスとスレッドはいずれも並列に動作させる単位を指していますが、その実行方法が異なります。プロセスは仮想計算機と呼ばれ、メモリ空間・CPUの利用等が独立して動く事になります。そのために、複数のプ ロセスを並列して動かすためには、プロセス間で情報の受け渡し(message passing)が必要になります。MPIはその情報の受け渡しのための一つのプロトコル(規格)です。 | ||
| + | それに対してスレッドは | ||
| + | |||
| + | === 1.5 Install motion-correction software(モーション補正ソフトウェアのインストール) === | ||
| + | RELION-2.0は、全フレーム顕微鏡写真ムービーアライメント[[#ref5|[5]]]に使用される、UCSFプログラムMOTIONCORRへのラッパーを提供します。[http://cryoem.ucsf.edu/software/driftcorr.html Yigan Chengのページ]からプログラムをダウンロードし、インストール手順に従ってください。また、Niko GrigorieffのグループからUNBLUR [[#ref3|[3]]]を使用することもできます。 [http://grigoriefflab.janelia.org/unblur Niko's UNBLUR Webサイト]からダウンロードしてください。 | ||
| + | |||
| + | ※ 全フレーム顕微鏡写真ムービーアライメント(Motion Correction([[モーション補正]]))とは、CMOSカメラ等で撮影された連続写真を使って、グリッドのドリフト(移動)などの補正を行う為の操作です。 | ||
| + | |||
| + | ※ ここで述べるラッパーとは、他のプログラム等と結合するためのプログラムのことを示しています。 | ||
| + | |||
| + | === 1.6 Install CTF-estimation software(CTF推定ソフトウェアのインストール) === | ||
| + | CTF推定は、RELIONには含まれていません。代わりに、RELIONは、Niko GrigorieffのCTFFIND3 [[#ref6|[6]]]、またはAlexis Rohou、CTFFIND4 [[#ref7|[7]]]によって書かれたこのプログラムの次世代へのラッパーを提供します。[http://grigoriefflab.janelia.org/ctf Niko's CTFFINDのWebサイト]からダウンロードし、インストール手順に従ってください。また、あなたのマシンにNVIDIAグラフィックスカード(GPU)を持っている場合は、Kai ZhangのGCTF [18]を使用することもできます。これは[http://www.mrc-lmb.cam.ac.uk/kzhang/ Kai's website at LMB]からダウンロードできます。 | ||
| + | |||
| + | #[[CTF]] (Contrast transfer function)とは、電子顕微鏡写真における撮影上の変調を表現した関数(フーリエ空間で定義)です。 | ||
| + | |||
| + | === 1.7 Install RESMAP(RESMAPのインストール) === | ||
| + | 局所分解能推定もRELIONの一部ではありません。 代わりに、RELIONはAlp KucukelbirのRESMAP [[#ref4|[4]]]にラッパーを提供します。 [http://resmap.sourceforge.net/ Alp'sRESMAP website]からダウンロードし、インストール手順に従ってください。 | ||
| + | |||
| + | # RESMAPは、再構成されたプログラムを利用して、場所毎の分解能を計算するためのプログラムです。 | ||
| + | |||
| + | === 1.8 Download the test data(テストデータのダウンロード) === | ||
| + | このチュートリアルでは、さまざまなベータガラクトシダーゼの再構成を計算するために使用される顕微鏡写真のサブセットであるテストデータセットを使用しています[13,2,17]。計算負荷を軽減するために、これらのデータを2倍ダウンサンプリングしたものを利用しています。データは、直接ブラウザから[ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion20_tutorial.tar.gz ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion20_tutorial.tar.gz]ダウンロードするか、次のコマンドを使用してダウンロードして解凍することができます。 | ||
wget ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion20_tutorial.tar.gz | wget ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion20_tutorial.tar.gz | ||
gunzip relion20_tutorial.tar.gz | gunzip relion20_tutorial.tar.gz | ||
tar -xf relion20_tutorial.tar | tar -xf relion20_tutorial.tar | ||
| − | + | ||
| + | ----- | ||
| + | '''より便利な使い方''' | ||
| + | |||
| + | また、日本国内からアクセスしている場合以下のURLの方が高速かもしれません。 | ||
| + | http://www.yasunaga-lab.bio.kyutech.ac.jp/pub/relion20_tutorial.tar.gz | ||
| + | Checksum MD5: c62ff74922283389c5318f3e852365ce | ||
| + | ダウンロードしたファイルを解凍する際には、「pigz」プログラムを使うと更に高速です。「pigz」プログラムは、aptまたはyumで入手できます。それぞれのOSでのインストール方法は、 | ||
| + | Ubuntu: apt install pigz | ||
| + | CentOS: yum install pigz | ||
| + | です。インストールには管理者権限が必要です。また、このファイルの解凍方法は、 | ||
| + | tar xvf relion20_tutorial.tar.gz --use-compress-program=pigz | ||
| + | です。おおよそ、場合によりますがおおよそ1.5-2倍程度高速化されます。 | ||
| + | |||
| + | ----- | ||
| + | |||
| + | === 1.9 キューイングシステムの利用 === | ||
| + | |||
| + | このチュートリアルに従って演習をする際に、独自のコンピュータシステムを使用している場合には注意が必要です。特に、計算機クラスタで実行するには、それぞれ特定のキューイングシステム用に構成されたqsub.cshスクリプトが必要となるでしょう。 正しいスクリプトを持っているかどうかわからない場合は、システム管理者に相談してください。 | ||
== 2 What's new in RELION-2.0? == | == 2 What's new in RELION-2.0? == | ||
| − | === 2.1 The GUI === | + | === 2.1 The GUI(GUIについて) === |
| − | + | [[ファイル:relion.png|500px]] | |
| − | + | ||
| − | + | ==== 2.1.1 A pipeline approach(パイプラインアプローチ) ==== | |
| + | RELION-1.4のGUIは、便利なコマンドラインプログラムを自動生成するだけにすぎませんでしたが、RELION-2.0のGUIは、新しいパイプラインのアプローチにおいて、より中心的な役割を果たすようになりました。 | ||
| + | |||
| + | 以前のバージョンでは、ユーザーがそれぞれの異なるjobから出力ファイルを紐づけする必要がありました。また、以前のプログラムはプロジェクトの履歴を認識できませんでした。 | ||
| + | |||
| + | RELION-2.0では、GUIがすべてのjobの動作経歴と、それぞれのjobがどのような出力をし、それを他のjobに入力したのかを保存します。そのため、パイプラインを形成できます。その結果、ユーザは、出力ファイルがどこに格納されるかを、ユーザ自身が管理しておく必要は無くなりました。各jobタイプはそれぞれ独自の出力ディレクトリがあり、これらのjobタイプのディレクトリ内(例えばClass2D)では、新しいjobは連続する番号(Class2D/job010)が与えられます。これら個々のjobディレクトリ内では、Class2D/job010/runのように出力名が固定されています。各jobに意味のある名前を付ける仕組みを提供するために、ファイルシステム上の個々のjobディレクトリへのシンボリックリンクとして実装されたジョブ "エイリアス"のシステムが使用されています。 | ||
| + | |||
| + | パイプラインに関するすべての情報は、default_pipeline.starというファイルに保存されますが、通常の状況ではユーザーはこのファイルを調べる必要はありません。 | ||
| + | |||
| + | ※ ここで述べるパイプラインとは、一つのプログラムの出力を次のプログラムの入力として順次繋いでいくような情報の受け渡し方法のことを示しています。 | ||
| + | |||
| + | [[ファイル:RELION-desktop.png|500px]] | ||
| + | |||
| + | ==== 2.1.2 The upper half: jobtype-browser and parameter-panel(上半分:jobタイプブラウザとパラメータパネル) ==== | ||
| − | |||
GUIは上半分と下半分に分かれています。上半分はRELION-1.4に似ています。左側にjobタイプ(【2D classification】のようなjobタイプを選ぶ縦の)ブラウザがあります。このリストはRELION-2.0で拡張されました。右側には複数のタブがあるパネルがあり、それぞれのjobタイプへのパラメータを入力できます。GUIの左上には3つの異なるメニューがあり、さまざまな機能が提供されています。RELION1.4の【Run!】ボタンが【Schedule】と【Run now!】の2つの新しいボタンに置き換えられました。前者のものは新しく、今後の実行のためにjobをスケジュールするために使用できます。これは、データが収集される際にリアルタイムで反復的に実行できる完全に自動化された「パイプライン」の準備に特に役立ちます。詳細は以下を参照してください。GUIの左側のjobtype-browserをクリックすると、新しい動作(【Run now!】ボタン付き)が右側のパラメータパネルにロードされます。 | GUIは上半分と下半分に分かれています。上半分はRELION-1.4に似ています。左側にjobタイプ(【2D classification】のようなjobタイプを選ぶ縦の)ブラウザがあります。このリストはRELION-2.0で拡張されました。右側には複数のタブがあるパネルがあり、それぞれのjobタイプへのパラメータを入力できます。GUIの左上には3つの異なるメニューがあり、さまざまな機能が提供されています。RELION1.4の【Run!】ボタンが【Schedule】と【Run now!】の2つの新しいボタンに置き換えられました。前者のものは新しく、今後の実行のためにjobをスケジュールするために使用できます。これは、データが収集される際にリアルタイムで反復的に実行できる完全に自動化された「パイプライン」の準備に特に役立ちます。詳細は以下を参照してください。GUIの左側のjobtype-browserをクリックすると、新しい動作(【Run now!】ボタン付き)が右側のパラメータパネルにロードされます。 | ||
| − | ==== 2.1.3 The lower half: job-lists and stdout/stderr windows ==== | + | ==== 2.1.3 The lower half: job-lists and stdout/stderr windows(下半分:jobリストとstdout/stderrウィンドウ) ==== |
| − | + | GUIの下半分には、まだ実行中のジョブ(【Running jobs】)、すでに完了しているジョブ(【Finished jobs】)、または後で実行するようにスケジュールされたジョブ(【Scheduled jobs】)のリストが含まれています。これらのリスト内の動作をクリックすると、その動作のパラメータがパラメータパネルにロードされ、【Run now!】ボタンの色が変わり、【continue now!】に変わります。それをクリックすると、新しい出力jobディレクトリは作成されませんが、パラメータパネルで指定されたパラメータに従ってjobが続行されます。【2D classification】、【3D classification】、【3D auto-refine】jobでは、_optimiser.starファイルが必要になり、ファイル名には、run_ct23のような継続された繰り返しのファイル名が付きます。他のjobタイプでは、それらが以前に実行されるまで、その時点から継続することができます。【モーション補正】、【CTF推定】、【自動ピッキング】、【粒子抽出】は、これまでに行われていなかった顕微鏡写真でのみ実行されます。【Input to this job】(このjobへの入力)と【Output from this job】(このjobからの出力)は、リンクjobをまとめてリストし、プロジェクト履歴内を前後にブラウズするために使用できます。 | |
| − | + | GUIの下半分の下部には、選択された(実行済みまたは実行中の)jobの標準出力(stdout)と標準エラー出力(stderr)がそれぞれ黒と赤のテキストで表示されます。は理想的には空白でなければなりません。これらのテキスト表示は、jobリスト内のjobをクリックするたびに更新されます。stdoutまたはstderrのディスプレイをダブルクリックすると、より便利なスクロールのためにテキスト全体を含むポップアップウィンドウが開きます。 | |
| − | + | ||
| − | ==== 2.1. | + | ==== 2.1.4 The Display button(ディスプレイボタン) ==== |
| + | RELION-1.4には、GUI上に汎用の【Display!】ボタンがあり、粒子、顕微鏡写真、マップ、マスクなどの表示に使用できました。一般的なファイルブラウザが開いて、これらのファイルを選択することができました。このボタンは、RELION-2.0のGUIからは消えましたが、GUIの左上にある【File】メニューの【Display】オプションを使用してアクセスすることができるように変わっています。 | ||
| − | + | [[ファイル:File-RELION2.0.png|500px]] | |
| − | + | 【便利になった点】RELION-2.0では、汎用の【Display】ボタンの代わりに、【Run】ボタンと【Schedule】ボタンの下に、もっと特別な【Display:】ボタンができました。GUIの下半分にあるjobリストのうちのひとつのjobを選択した後にこのボタン【Display:】をクリックすると、選択されたjobの入力と出力がすべて(例えば、パーティクル、顕微鏡写真、座標、 PDFファイルなど)がポップアップメニューとして表示できます。 これにより、新しい【Display】ボタンはファイルブラウザを起動する必要がなくなりました。 | |
| − | + | ||
| − | + | ||
| − | + | もう1つの違いは、RELION-1.4での【Display】ボタンは、(_model.starを表示することによって)クラスファイルを選択するために使用されていましたが、RELION-2.0では、【Display:】ボタンでこれを行うことができなくなりました。その代わりに、【Subset selection】の新しいjobタイプができました。このjobでは、クラスや顕微鏡写真を選択することができます。このjobタイプには、顕微鏡写真を再グループ化し、質量中心に応じて自動的に2Dクラス平均をセンタリングできる(中心を移動させる)タブもあります。後者は、これらのクラス平均を自動ピッキングのテンプレートとして使用する場合に便利です。 | |
| − | subset | + | ==== 2.1.5 The Job action button(jobアクションボタン) ==== |
| + | 【Job action】ボタンは、選択した(実行中、完了後、またはスケジュールされた)ジョブの設定を含む小さなメニューを開きます。ここで、note.txtというファイルにアクセスすることができます。このファイルは個々のジョブディレクトリに保存されており、ユーザーのコメントを格納したり、jobのエイリアスを変更したり、jobを終えたものをマークすることができたり、job履歴のフローチャートを作ったり(LATEXとTikZパッケージがシステムにインストールされている場合は、第10章を参照してください)、ディスク容量を節約するためにjobを削除またはクリーンアップ(下記参照)したりすることができます。 | ||
| + | |||
| + | ==== 2.1.6 Clean-up to save disk space(ディスク領域を節約するためのクリーンアップ) ==== | ||
| + | jobを削除すると、jobディレクトリ全体がProjectディレクトリからTrash/というディレクトリに移動します。RELIONの左上にある【File】メニューの【Trash】フォルダを空にすると、完全に削除され空き領域が増えます。これを行うまでは、左上の[job]メニューから対応する設定を使用して、jobを復元することができます。 | ||
| + | |||
| + | ディスク容量を節約するため、jobを「clean」にすると、refine jobのすべての中間的な繰り返しに対して書き出されたファイルのような中間ファイルをごみ箱フォルダに移動します。2つのクリーニングオプションがあり、片方は【gentle clean】穏やかなクリーニング方法です。この方法では、他のジョブへの入力として使用できるすべてのファイルをそのまま残します。もう一方は【harsh clean】完全なクリーニング方法です。この方法では全てのファイルを消しますので、より多くの容量を確保でき、【Motion correction】、【Particle extraction】、【Movie refinement】、【Particle polishing】のjobタイプから選択できる粒子スタックまたは顕微鏡写真を含むディレクトリに関しては特に、大きく容量を確保できます。 | ||
| + | |||
| + | また、RELIONの左上にある【job】メニューの対応するオプション(【Gently clean all jobs】あるいは、【Harshly clean all jobs】)を使用して、プロジェクト内のすべてのディレクトリをワンクリックで消去することもできます。 | ||
| + | その際、特定のディレクトリを削除対象から除外したい場合には、NO_HARSH_CLEANというファイルをその中に置いてください。 | ||
| + | |||
| + | 例えば、保護したい粒子データがjob098にある場合、次のコマンドを実行してください。 | ||
| + | $ touch Polish/job098/NO_HARSH_CLEAN | ||
| + | |||
| + | ※ touchコマンドは、指定したファイルが存在しない場合には空のファイルを作成するプログラムです。存在している場合には、ファイルの修正時刻を変更します。 | ||
| + | |||
| + | === 2.2 Optimise computations for your setup(それぞれの環境における計算を最適化) === | ||
| + | ==== 2.2.1 GPU-acceleration(GPUによる高速化) ==== | ||
| + | Erik Lindahl(ストックホルム)のグループのDari KimaniusとBjoern Forsbergは、RELIONの計算コストの高い部分を移植して、GPUを使うようにしました。彼らはNVIDIAのcuda-librariesを使ってこの作業を行ったため、RELION内のGPUアクセラレータはnvidiaカードでしか動作しません。これらは、NVIDIAのGPUが、compute campacity 3.5以上である必要があります。単精度と倍精度の両方のカードが動作するため、高価な倍精度カードに限らず安価なゲーミングカードを使用することもできます。 | ||
| + | |||
| + | 2つの異なるRELIONプログラムがGPUにより高速化されています。それは、relion_autopick(自動ピッキング)とrelion_refine(2次元分類、3次元分類、3次元自動精密化job)です。これらのプログラムのシーケンシャル版とMPI版の両方が加速されました。 | ||
| + | |||
| + | ==== 2.2.2 Disk access(ディスクへのアクセス) ==== | ||
| + | GPUアクセラレータによって画像処理の速度がはるかに向上したために、ハードディスクへのアクセスがますますネックになります。RELION-2.0のGUIには、データセットとコンピュータセットアップのディスクアクセスを最適化するためのいくつかのオプションが用意されています。2次元分類、3次元分類、3次元自動精密化のために、パラレルディスクI/Oの使用【'''use parallel disc I/O'''】を選択できます。Yesに設定すると、すべてのMPIプロセスがハードディスクから同時に粒子を読み込みます。それ以外の場合は、マスターだけがイメージを読み取り、ネットワークを介してスレーブに送信します。fhgfsのglusterのような並列ファイルシステムは、並列ディスクI/Oで優れています。NFSは、多くのスレーブが並行して読み込みを行うと中断することがあります。 | ||
| + | |||
| + | メモリ中にプールされた粒子の数【'''number of pooled particles'''】を設定することもできます。粒子はMPIスレーブによって個々のバッチで処理されます。各バッチ中に、粒子画像のスタックは、一度だけ開閉され、ディスクアクセス時間を改善します。1つのバッチのすべての粒子画像は、一度にメモリに読み込まれます。これらのバッチのサイズは、使用されるスレッド当たり少なくとも粒子1つ分です。このパラメータは、スレッドごとにバッチ内で一緒に読み込まれる粒子の数を制御します。例えば、3に設定し、1つにつき8つのスレッドを使用する場合、3x8 = 24個の粒子のバッチが一緒に読み込まれます。これにより、ディスクアクセス、特にディスクアクセスのメタデータ処理が問題となるシステムでのパフォーマンスが向上する可能性があります。それは、控えめなRAM使用を与えます。 | ||
| + | |||
| + | 比較的小さなデータセット(および/または大量のRAMを持つコンピュータ)を使用する場合は、計算の開始時にすべての粒子をRAMに事前に読み込むことができます【'''pre-reqd all particles into RAM'''】。これにより、ディスクアクセスが比較的遅いシステムでの計算が大幅に高速化されます。ただし、使用可能なRAMの量には注意が必要です。粒子はフロート精度で読み込まれるため、N個の粒子をRAMに読み込むには(N×boxsize×boxsize×4)/(1024×1024×1024)GBが必要です。200ピクセルのボックスサイズを持つ100,000個の粒子は15Gbになり、400ピクセルのボックスサイズでは同じ数の粒子に対して60Gbになります。 | ||
| + | |||
| + | データセットが大きすぎてRAMに先読みできないが、各計算ノードに同じ名前でマウントされたローカルの高速ディスク(ソリッドステートドライブなど)(スクラッチディスクと呼ぶ)がある場合、各MPIスレーブはすべての粒子を 計算を開始する前にローカルディスクを使用することができます。これは、粒子をスクラッチディレクトリにコピーすることによって行われます【'''Copy particles to scratch directory.'''】。複数のスレーブが同じノード上で実行される場合、最初のスレーブだけが粒子をコピーします。ローカルディスクが小さすぎてデータセット全体を保持できない場合、もはやスクラッチディスクに置くことができない粒子は元の場所から読み込まれます。'''relion_volatile'''というサブディレクトリが、指定されたディレクトリ名の内部に作成されます。例えば、'''/ ssd'''を指定すると、'''/ ssd / relion_volatile'''という名前のディレクトリが作成されます。 '''/ ssd / relion_volatile'''ディレクトリがすでに存在する場合は、粒子をコピーする前に消去されます。 次に、プログラムはすべての入力粒子をこのディレクトリ内の単一の大きなスタックファイルにコピーします。 ジョブが正常に終了すると、'''/ ssd / relion_volatile'''ディレクトリが再度削除されます。 | ||
| + | |||
| + | 終了する前にjobがクラッシュした場合は、それを自分で削除したいと思うことでしょう。このとき、プログラムは、全ての人の書き込み権限付きで'''/ ssd / relion_volatile'''ディレクトリを作成します。それにより、スクラッチディレクトリとして、'''/ ssd'''を、すなわちユーザ名なしで使用することを選択することができます。そのほうが、各計算ノード上の1人のユーザーが常に1つのjobしか実行しないという条件で、ローカルディスクは、jobがクラッシュし、ユーザーがスクラッチディスクのクリーニングを忘れたときでも、ジャンクでいっぱいになる危険性はありません。(つまり、全員が同じスクラッチディレクトリ名を使えば、毎回、スタート時にクリアすることになるので、ジャンクを消し忘れるといったことがないという意味です。) | ||
| + | |||
| + | 最後に、ディスクを通して繰り返しを組み合わせるオプション【'''combine iterations through disc'''】があります。Yesに設定すると、すべてのMPIスレーブはすべての繰り返しの最後に、蓄積された結果をひとつの大きなファイルに書き出します。MPIマスターはこれらすべてのファイルを読み込み、それらをすべて結合し、結合された状態で、ひとつの新しいファイルに書き出します。すべてのMPIスレーブは、その後、その結合された結果を読み込みます。これにより、ネットワークの負荷が軽減されますが、ディスクI / Oの負荷が増加します。これは、期待値ステップ(E-step)の終わりに達する進捗バー(マウスがチーズに達する)とその後の最大化ステップ(M-Step)の開始にかかる時間に影響します。この時間は、非常に効率的なシステム設定に依存します。このオプションは当初、LMBでの古いクラスタ上でのネットワークカードのバグを回避するために実装されました。 今日では、精密化が高解像度に達すると非常に遅くなる傾向があるので、このオプションは使用しない方が望ましいです。 | ||
| + | |||
| + | === 2.3 On-the-fly processing running scheduled jobs(スケジュール化されたジョブのon-the-fly実行(撮影しながら処理を実行)) === | ||
| + | |||
| + | [[ファイル:AutoRun-RELION2.0.png|500px]] | ||
| + | |||
| + | 【'''Schedule'''】ボタンから、今後のjobの準備をできます。スケジュール化された動作により期待される出力ファイルは、既に【Browse】ボタンから他のjobへの入力として利用できるため、'''Scheduled jobs'''に複数の連続した動作の「'''パイプライン'''」を構築することが可能です。GUIの左上の【'''Autorun'''】メニューからスケジュール化されたjobを実行できます。そのプログラムは何回スケジュール化された動作を実行するか聞いてきます。1より大きい値を指定すると、すべてのスケジュールしたjobが繰り返し実行されます。この設定にした場合、スケジュール化されたjobが一周毎に、何分経過させるべきかという質問が出てきます。(もしスケジュールしたjobの実行時間がこの時間よりも短い場合、このプログラムは、次にスケジュールしてあるjobの実行を待ちます。)jobタイプが、【Import】、【Motion correction】、【CTF estimation】、【Auto-picking】、【Particle extraction】、【Movie refinment】である場合には、以前の反復で完了していない顕微鏡写真のみを処理します。他の全てのjobタイプでは、スケジュールされたjobの実行の各反復後、再度、最初から再計算を行います。 | ||
| + | |||
| + | |||
| + | この設定により、顕微鏡写真を収集する際に、高速処理を便利に行うことができます。顕微鏡写真や顕微鏡動画を顕微鏡コンピュータから処理コンピュータ(好ましいのは、高速処理のための1つまたは複数の適切なGPUを搭載しているもの)にコピーするスクリプトを設定することができます。 | ||
| + | |||
| + | ※大きなファイルをコピーするのには時間がかかり、まだコピーの終わっていない顕微鏡写真や動画をインポート【Import】するのは好ましくありません。そのため、このスクリプトはファイルを一時的なファイル名でコピーし、コピーが完全に終了したときにのみ、ファイルを適切なファイル名に変更するべきでしょう。 | ||
| + | |||
| + | 例えば、【Micrograghs/*.mrcs】ファイルの【Import】で始まる様に、スケジュールされたjobリストは、適切なテンプレートがすでに用意されている場合に葉、【Motion correction】、【CTF estimation】、【Auto-picking】へと順次実行することができるでしょう。そうでない場合には、いくつかの顕微鏡写真を取得した後で手動で粒子を選択する必要があるでしょう。その後、【Particle extraction】、【2D classification】へと順次進みます。このスケジュールしたjobリストは、それぞれの繰り返し毎に、恐らく少なくとも30分または60分で、何度も繰り返されます。インポートされた顕微鏡写真のリストはますます増えますが、スケジュールしたjob(【'''Motion correction'''】、【'''CTF estimation'''】、【'''Auto-picking'''】、【'''Particle extraction'''】)の反復では、新しい顕微鏡写真のみで実行されます。一方で、【2D classification】は、毎回の繰り返し毎に全ての粒子数を最初から計算し直します。電子顕微鏡でデータ集めている間に2次元クラス平均を調べることは、既に最初の場所で撮影された価値のあるデータが得られたかどうか、あるいは、投影方向性分布が大丈夫かどうかを決めるのに有益でしょう。後者の場合には、恐らく、より氷が厚いグリッド上で収集する必要があるでしょう。既に3Dリファレンスが良ければ、【'''3D classification'''】または【'''3D auto-refine'''】を、並列してそのまま実行できるかもしれません。また、このことは、例えば、ある補因子が目的の複合体に結合しているかどうかを確認するなどの場合に有用なことでしょう。 | ||
| + | |||
| + | 【'''subset selection'''】はスケジュールすることができますが、【'''2D classification'''】の_model.starを表示して適切なクラスを選択するなど、入力を求められることに注意してください。全てのjobの実行はユーザーがこの選択を保存するまで続きません。このステップの自動化は次のRELIONのバージョンに組み込ませるつもりです。 | ||
=== 2.4 Helical processing(ヘリカル処理) === | === 2.4 Helical processing(ヘリカル処理) === | ||
| − | Scheresグループの博士課程学生であるShaoda | + | Scheresグループの博士課程学生であるShaoda Heは、ヘリカルアセンブリ処理のためのワークフローを実装しています。これには、【Auto-picking】、【Partcle extraction】、【2Dclassification】、【3Dclassification】、【3D auto-refine】、【Particle polishing】、および【Mask create】といったjobタイプのパラメータパネルへの追加タブが含まれます。これらオプションのより詳細な説明は、ユーザーが[http://www2.mrc-lmb.cam.ac.uk/relion/index.php/Helical_processing RELION Wiki]の関連するページから参照できます。 |
=== 2.5 Other new functionalities(他の新しい機能) === | === 2.5 Other new functionalities(他の新しい機能) === | ||
| − | ==== 2.5.1 Dealing with differently scaled particles( | + | ==== 2.5.1 Dealing with differently scaled particles(異なったスケールの粒子の取扱) ==== |
| − | RELION-1. | + | RELION-1.4では、STARファイル等を修正するためのawkスクリプトを含んでいたため、粒子をどれだけダウンスケールするか(あるいはしてないか)に関する変更がいくらか可能でした。RELION-2.0では、もっと手軽に縮小ができるようになりました。【'''Particle extraction'''】 jobでは、【Manual picking】、【Auto-picking】から提供された座標の替わりに、【'''2D classification'''】、【'''3D classification'''】、【'''3D auto-refine'''】から _data.starを提供する【'''re-extract refined particle'''】オプションを備えています。このオプションは、、抽出された粒子の新しいSTARファイルに原点オフセットを書き出す際、各入力粒子のスケールの違いを正しく説明するでしょう。また入力_data.starファイルの調整された原点オフセットによって定義された中心で粒子を改めて抽出することもできます(【'''Re-center refined coordinates'''】オプションを使用します)。 |
| − | 他のRELION-1. | + | 他のRELION-1.4の不便なことは【Auto-picking】でのテンプレートが取得してきた顕微鏡写真と同じスケールでなければならないことです。RELION-2.0ではそうではなく、【Reference】タブ上で【pixel size in references】で値を与える時に(もしくは【I/O】タブ上のpixel size in micrographsオプションを利用時)、入力する顕微鏡写真のSTARファイルで定義されたピクセルサイズと違っててもよいです。 |
| − | ==== 2.5.2 Particle | + | ==== 2.5.2 Particle subtraction(粒子の減算) ==== |
| − | + | 【Particle subtraction】 jobタイプは、RELION-2.0パイプライン内部で部分的なシグナル減算の手順を正式な手順として採用しています。入力マップの投影像(入力粒子以前の3Dクラス化【3D classifiction】または自動3D精密化【3D auto-refine】からの _data.starファイルとして、提供されます。)から減算されます。マップを投影する前に、入力マスクがマップに適用されます。それゆえに、マスクは、粒子から減算したい領域では白く、それ以外では黒くする必要があります。 | |
| − | + | 【粒子から得られるシグナルをマスクに反映させ、それを一面黒のマップに適用することで、粒子のある領域では白く、それ以外では黒いマップができます】 | |
| − | + | 同じjobタイプで、(入力 _data.starファイル上のコラムを【rlnImageName】と【rlnImageOriginName】切り替えることで)減算された粒子から元の粒子へと戻すことができます。これは、たとえば、データを構造的に同種のサブセットに分類するために部分シグナル減算後に、複合体全体を精密化したい場合などに便利です。シグナル減算の手順の詳細については、eLifeの論文[[#ref1|[1]]]を参照してください。 | |
==== 2.5.3 Join STAR files ==== | ==== 2.5.3 Join STAR files ==== | ||
| − | RELION-2. | + | RELION-2.0ではパイプラインが公式に採用されたことに伴い、粒子、顕微鏡写真、または顕微鏡動画のSTARファイルへとつなげることができるようになりました。このことは、【join star file】という新しいjobタイプによって行われます。4つ以上のファイルを結合する場合は、GUIからこのユーティリティを複数回呼び出す必要があります(完全に記述されたパイプラインを維持したい場合)。または、【relion_star_combine】コマンドラインプログラムを多数のファイルで実行し、結果のSTARファイルをパイプラインに再度【Import】を用いる方法でもできます |
=== 2.6 How this tutorial works(本チュートリアルでの動作法) === | === 2.6 How this tutorial works(本チュートリアルでの動作法) === | ||
| − | RELION-1.4チュートリアルでは、チュートリアルの時間を節約するために、あらかじめ計算された結果をprojectディレクトリにコピーすることができます。RELION-2. | + | RELION-1.4チュートリアルでは、チュートリアルの時間を節約するために、あらかじめ計算された結果をprojectディレクトリにコピーすることができます。RELION-2.0では動作にパイプラインを用いたことにより、同じことができません。かわりに、PrecalculatedResultsディレクトリに完全に実行されたパイプラインを用意しています。ユーザーはbetagalディレクトリ内のすべてのステップを実行することができます(少なくとも1つの適切なGPUを使用していたら、1日以内に終わります)。またはPrecalculatedResultsディレクトリのプロセスに従います。 |
| + | |||
| + | == 3 Preprocessing(前処理) == | ||
| + | '''前書き''' | ||
| − | + | 原理上はRELIONでそれぞれのプログラムから抽出された分子を使用できますが、おすすめできません。多くのプログラムが粒子自体を例えば、段階の切り替えや帯域通過フィルタ、ウィナーフィルター、マスクなどによって変化させているためです。これらはすべてその後のRELIONの使用のために最適とはいえません。さらに、RELIONタイプのSTARファイルにフォーマットされたファイルの中に必須のメタデータを全て収集すると、エラーが発生する傾向があります。RELIONでの粒子の再抽出は単純でとても速いため、後述の手順はRELIONではとても容易な(良い)方法です。 | |
| − | + | ||
| − | + | ||
また、RELIONでいくつかのラッパーの実装が報告されています(例えば、EMAN2、SCIPION、APPION)。ラッパーが作られたとき、うまく活用しようと試みましたが、完全には制御できず、これらのラッパーが最適な方法でRELIONを使用できているかどうかわかりませんでした。そのため、ラッパーで得られた結果に疑問がある場合、後述のチュートリアルで説明している手順で行うことをお勧めします。 | また、RELIONでいくつかのラッパーの実装が報告されています(例えば、EMAN2、SCIPION、APPION)。ラッパーが作られたとき、うまく活用しようと試みましたが、完全には制御できず、これらのラッパーが最適な方法でRELIONを使用できているかどうかわかりませんでした。そのため、ラッパーで得られた結果に疑問がある場合、後述のチュートリアルで説明している手順で行うことをお勧めします。 | ||
| − | === 3.1 Getting organised ( | + | === 3.1 Getting organised (解析の準備)=== |
| − | + | プロジェクト毎、すなわち、解析したい構造ごとに一つのディレクトリを作ることをお勧めします。これをprojectディレクトリと呼ぶことにします。'''projectディレクトリからRELIONを起動することが重要です。'''projectディレクトリの中にすべての未加工の顕微鏡写真や顕微鏡動画をMRC形式で保存するための別ディレクトリを作ってください。例えば、すべての顕微鏡写真がひとつのディレクトリにある場合は、通例ではディレクトリをMicrographs/にします。また、異なるディレクトリにある場合(例えば、違う日に収集した顕微鏡写真)はMicrographs/15jan16/やMicrographs/23jan16/などにすると良いでしょう。もし何らかの理由でRELIONのprojectディレクトリの中に顕微鏡写真を入れたくない場合は、projectディレクトリの中に顕微鏡写真が保存されているディレクトリとのシンボリックリンクを作成してください。 | |
| − | + | 単一像(静止画)の顕微鏡写真には.mrc拡張子を、連続スタック像(動画)には.mrcs拡張子をつけてください。単一像顕微鏡写真の代わりに動画を集めた場合でも、それらを直接使用できます。別のプログラムで動画の平均を計算し、さらに、ポリッシングなどの過程で、顕微鏡写真と動画の平均を両方とも使用したい場合、動画のファイル名は顕微鏡写真の平均と同じルートネームにするべきです。つまり、顕微鏡写真の平均のファイルの後に、動画を表現する”_movie"と、スタックファイルを意味する".mrcs"拡張子をつけます。例えば、顕微鏡写真の平均がmic001.mrcの場合、一致する動画はルートネームであるmic001に対して、mic001_movie.mrcsとしてください(動画を表現するのは_movie)。 | |
チュートリアルのテストデータを解凍した時、projectディレクトリ(betagal/)と、記録されたすべての顕微鏡動画と一致するbetagal/Micrographsディレクトリはすでに作成されています。projectディレクトリに行き、以下の入力をすると、どのくらい顕微鏡動画があるか分かります。 | チュートリアルのテストデータを解凍した時、projectディレクトリ(betagal/)と、記録されたすべての顕微鏡動画と一致するbetagal/Micrographsディレクトリはすでに作成されています。projectディレクトリに行き、以下の入力をすると、どのくらい顕微鏡動画があるか分かります。 | ||
| 行103: | 行214: | ||
ls Micrographs/ | ls Micrographs/ | ||
| − | + | 今回のチュートリアルでは、計算した結果(calcurated_betagal)も配付しています。 | |
| + | |||
| + | cd calcurated_betagal | ||
| + | ls Micrographs/ | ||
| + | |||
| + | RELIONを起動しましょう。前述の通り、RELIONは常にprojectディレクトリからの起動が必要です。エラーを防ぐために、RELIONは新しいディレクトリで初めて起動したときに確認を要求します。そのため、新しいディレクトリでRELIONを初めて起動するときは、バックグラウンドで起動するための”&”の文字を使うべきではありません。projectディレクトリ内にいることを確認し、以下の入力でRELIONを起動します。 | ||
relion | relion | ||
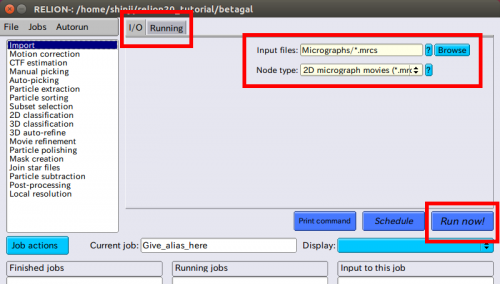
| − | + | ここで、新しいRELIONプロジェクトを作動するために”y”と答えます。すると、以下のような画面が立ち上がりますので、パイプラインの中へ録画した顕微鏡動画の取り込みを行います。そのため、jobタイプのブラウザから、赤枠で囲まれた”Import”を選択します。 | |
| + | |||
| + | [[ファイル:relion-import.png|500px]] | ||
| − | + | その後、画面が遷移し、入力ファイルを指定する画面になります。左上の赤枠で囲まれたタブが、「I/O」になっていることを確認して、右側の赤枠部のフィールドを以下のように入力します。通常は、プロジェクトディレクトリで起動すると、自動的に指定されているはずです。 | |
Input files: Micrographs/*.mrcs | Input files: Micrographs/*.mrcs | ||
Node type: 2D micrograph movies | Node type: 2D micrograph movies | ||
| − | Currrent job: | + | [[ファイル:relion-inputOutput.png|500px]] |
| + | |||
| + | Currrent job: Give_alias_hereという空欄に効果的な'''エイリアス'''を規定することができます。指定した内容が正しければ、【Run now!】のボタンをクリックし、job起動します。Import/job001/というディレクトリが作成され、一緒にImport/moviesというこのディレクトリへのシンボリックリンクが作成されます。テキストエディターでnote.txtファイルが開かれ、中には実際の実行コマンドが入っており、コメントも記入できます。必要なければ閉じてください。STARファイルとすべての動画が新しいディレクトリの中に作成されます。以下のコマンドを使って中を見ましょう。 | ||
less Import/job001/movies.star | less Import/job001/movies.star | ||
| 行120: | 行240: | ||
違うソフトウェアで粒子を抽出した場合、もしくは、RELION-1.4で抽出した粒子をRELION-2.0に移動させたい場合は、後述の事前処理を行う代わりに、STARファイルや3次元リファレンス、3次元マスクなどを取り込むために”Import”jobタイプを使用してください。 | 違うソフトウェアで粒子を抽出した場合、もしくは、RELION-1.4で抽出した粒子をRELION-2.0に移動させたい場合は、後述の事前処理を行う代わりに、STARファイルや3次元リファレンス、3次元マスクなどを取り込むために”Import”jobタイプを使用してください。 | ||
| − | === 3.2 Beam-induced motion correction( | + | === 3.2 Beam-induced motion correction(電子線により誘起される画像の動きの補正)=== |
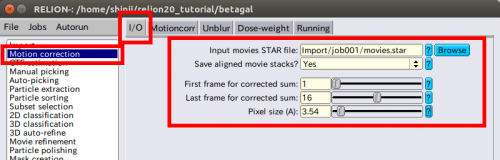
| − | Motion correction jobタイプは便利な全体フレーム動画の調節にためのUCSF MOTIONCORR [5]ラッパーを規定します。【I/ | + | Motion correction jobタイプは便利な全体フレーム動画の調節にためのUCSF MOTIONCORR [[#ref5|[5]]]ラッパーを規定します。【I/O】タブで以下の設定をします。まず、左カラムのジョブタイプを、「Motion correction」を選択し、「I/O」タブが選択されていることを確認し、それぞれのパラメータを設定します。 |
● Input movies STAR file: Import movies/movies.star | ● Input movies STAR file: Import movies/movies.star | ||
| − | ( | + | (【Browse】ボタンで参照できるファイルは、標準ではSTAR形式の動画ファイルのみです。) |
● Save aligned movie stacks? Yes | ● Save aligned movie stacks? Yes | ||
● First frame for corrected sum: 1 | ● First frame for corrected sum: 1 | ||
● Last frame for corrected sum: 16 | ● Last frame for corrected sum: 16 | ||
| − | ( | + | (ここでは16フレームの動画で、すべてのフレームを使って平均化します。) |
| + | |||
| + | [[ファイル:relion-motionCorrection-IO.png|500px]] | ||
【Motioncorr】タブは以下のように入力します。 | 【Motioncorr】タブは以下のように入力します。 | ||
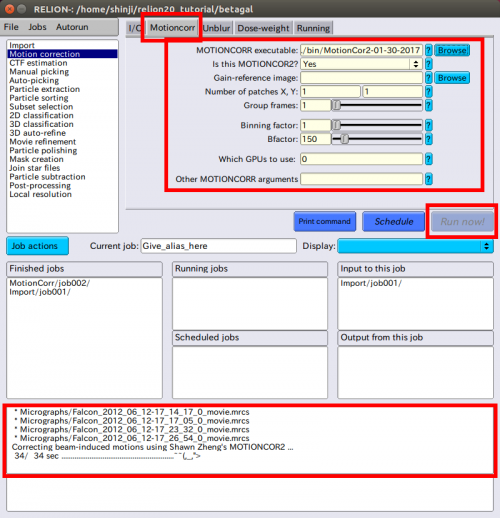
● MOTIONCORR executable: /wherever/it/is/motioncorr | ● MOTIONCORR executable: /wherever/it/is/motioncorr | ||
| − | ( | + | (環境変数$RELION_MOTIONCORR_EXECUTABLEを、設定することで、motioncorrプログラムの初期値を変更できます。) |
● Binning factor (1 or 2): 1 | ● Binning factor (1 or 2): 1 | ||
| − | ( | + | (超高解像度動画( super-resolution movies)では大抵、2を使用します。) |
● First frame for alignment: 1 | ● First frame for alignment: 1 | ||
● Last frame for alignment:16 | ● Last frame for alignment:16 | ||
| 行140: | 行262: | ||
(超高解像度動画では大きな値を使用します。) | (超高解像度動画では大きな値を使用します。) | ||
● Which GPUs to use: | ● Which GPUs to use: | ||
| − | ( | + | (MOTIONCORRはNVIDIA GPU上で動作する必要があります。GPUのデバイス番号を設定します。0と設定すると全てのプロセスが0を使います。0:1と設定するとプロセス毎に0と1を使います。) |
● Other MOTIONCORR armuments: | ● Other MOTIONCORR armuments: | ||
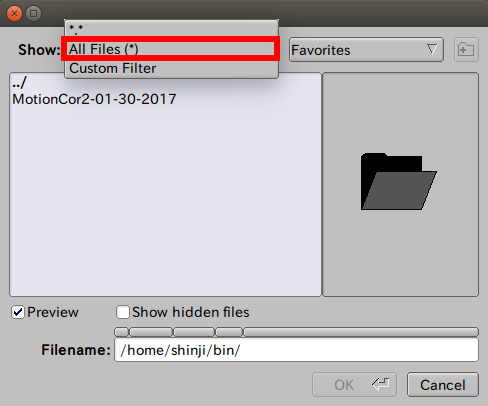
| − | このプログラムの実行にはおよそ1,2分かかります(しかし、この動作にはNVIDIA GPUが必要です)。あるいは、【Unblur】タブで利用できるNiko Grigorieff’s UNBLURプログラム [3]も使用できます。すべてのMOTIONCORR設定は無視され、初期値でUNBLURが実行できるため、高度なオプションが必要ありません。UNBLUR ラッパーはversion 1.0. | + | Motioncorrプログラムを選択する際には、 |
| + | |||
| + | [[ファイル:relion-motionCorrection-selectExe.png|500px]] | ||
| + | |||
| + | "Show:"部分を、「*.*」から「All Files (*)」へと変更しないと、表示されないかもしれません。Motioncorrタブの設定例は、次のようになります。設定完了後、赤枠の【Run now!】をクリックし、ドリフト補正を行います。 | ||
| + | |||
| + | [[ファイル:relion-motionCorrection-motioncorr.png|500px]] | ||
| + | |||
| + | 正常にプログラムが進行すると、下部赤枠のようにネズミの顔文字が、右へと進みます。今後のプロセスでも、同様にこの部分に注目することで、プログラムが正常に動作しているかがわかります。 | ||
| + | |||
| + | このプログラムの実行にはおよそ1,2分かかります(しかし、この動作にはNVIDIA GPUが必要です)。あるいは、【Unblur】タブで利用できるNiko Grigorieff’s UNBLURプログラム [[#ref3|[3]]]も使用できます。すべてのMOTIONCORR設定は無視され、初期値でUNBLURが実行できるため、高度なオプションが必要ありません。UNBLUR ラッパーはversion 1.0.2でテストされています。計算が終了した後、【Run now!】ボタンの下の【Display:】ボタンから'''out:logfile.pdf'''を選択することで推定された電子線によって誘起された変化を見ることができます。または、out:corrected_micrographs.starを選択することで、補正された(値が全て足された)顕微鏡写真を見ることができます。スクリーンサイズに依存して、見やすいように、顕微鏡写真(Scale:0.3)を縮小し、Sigma contrast:3を使い、見やすい数のコラム(例えば、Number of columns: 3(横に3つ並ぶ))にするべきです。この表示(【Display:】ボタンから表示したとき)から顕微鏡写真は選択できないことに注意してください。取り除きたい顕微鏡写真がある場合(今回はすべて良い画像なので、排除しないでしょうが)、【'''Subset selection'''】jobタイプが使用できます。 | ||
=== 3.3 CTF estimation(CTF推定)=== | === 3.3 CTF estimation(CTF推定)=== | ||
| − | 次に、補正した顕微鏡写真からCTFの値を推定します。速度のためKai | + | 次に、補正した顕微鏡写真からCTFの値を推定します。速度のためKai Zhangの GCTFのためのラッパーとして【'''CTF estimation'''】 jobタイプにします。適切なGPUがない場合、Niko Grigorieffの CTFFIND3やAlexis Rohouの CTFFIND4が使えることに留意してください(【'''CTFFIND'''】タブの値は初期値が良いでしょう)。CTFFIND4は顕微鏡動画も利用して、よりよいThon ringsを計算したり、顕微鏡動画位相板(Voltatype?)から推定した位相のずれを評価することもできます。 |
| + | |||
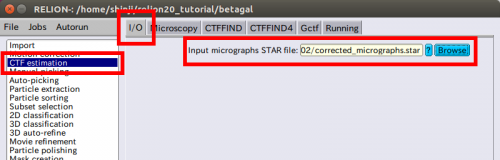
| + | まず、Jobリストより、【'''CTF estimation'''】を選択し、【I/O】タブで、【MotionCorr】 jobで作成したcorrected_micrographs.starファイルを選択するために【Browse】ボタンを使います。 | ||
| + | |||
| + | [[ファイル:relion-CTFest-IO.png|500px]] | ||
| + | |||
| + | また、他のタブは以下のように入力します。 | ||
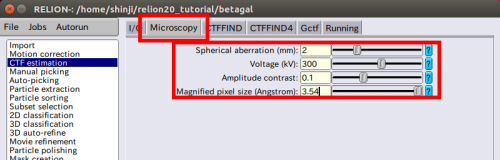
| − | + | 【Microscopy】タブでは実験条件を設定します。 | |
| − | ● | + | ● Spherical aberration(mm): 2 |
| − | ( | + | (顕微鏡メーカーがこの値を提供してくれています。) |
● Voltage(kV):300 | ● Voltage(kV):300 | ||
| + | (加速電圧を示します。) | ||
● Amplitude contrast:0.1 | ● Amplitude contrast:0.1 | ||
| − | ( | + | (強度コントラストはほとんどないことが知られていますが、10%ほどの値を与えることで、多くの構造の結果が良くなることが示されています。この方法は低周波数の弱い散乱がモデル化されていないためです。クライオ電子顕微鏡法では、7-20%(エネルギーフィルターがある場合)程度であることが知られています。) |
● Magnified pixel size(A): 3.54 | ● Magnified pixel size(A): 3.54 | ||
| − | ( | + | (今回提供したデータは2倍のビンデータです。もとのピクセルサイズは1.77Aです。) |
| − | CTF推定に使用するプログラムをNiko | + | [[ファイル:relion-CTFest-Microscopy.png|500px]] |
| + | |||
| + | CTF推定に使用するプログラムをNiko Grigorieffの CTFFIND3、Alexis Rohouの CTFFIND4、Kai Zhangの GCTFから選択することができます。例として、CTFFIND-4.0.8を使用したものを以下に示します。 | ||
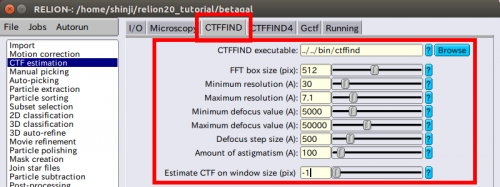
【CTFFIND】タブでの、CTFFINDの一般的な値を設定します(正確な意味についてはNikoのドキュメンテーションを参照してください)。 | 【CTFFIND】タブでの、CTFFINDの一般的な値を設定します(正確な意味についてはNikoのドキュメンテーションを参照してください)。 | ||
● CTFFIND executable: /wherever/is/your/ctffind.exe | ● CTFFIND executable: /wherever/is/your/ctffind.exe | ||
| − | 環境変数は$RELION_CTFFIND_EXECUTABLEを使うと、初期値から調節でき、同様に【Gctf】タブでGCTF実行ファイルの初期値は$RELION_GCTF_EXECUTABLEで調節できます。CTFFIND-4.0. | + | 環境変数は$RELION_CTFFIND_EXECUTABLEを使うと、初期値から調節でき、同様に【Gctf】タブでGCTF実行ファイルの初期値は$RELION_GCTF_EXECUTABLEで調節できます。CTFFIND-4.0.xを使っている場合、ダブルクォーテーションの間の実行ファイルが実行され、以下の追加の引数を規定します。 |
| − | “/path/to/ctffind.exe –omp-num-threads 1 –old-school-input” | + | |
| − | + | CTFFIND-4.1以降を使用している場合は、 | |
| − | ● FFT box size(pix): 512 | + | “/path/to/ctffind.exe –omp-num-threads 1 –old-school-input” |
| − | ● Minimun resolution(A): | + | を追加する必要ありませんが、新しいバージョンで【CTFFIND4】タブを使用する場合は表示が必要です。 |
| − | ● | + | ● FFT box size(pix): 512 |
| − | ● | + | (フーリエ変換のボックスの大きさ) |
| + | ● Minimun resolution(A): 30 | ||
| + | (利用する最低分解能(低周波側にあるノイズはCTFの推定に悪い影響を与えます)) | ||
| + | ● Maximum resolution(A): 7.1 | ||
| + | (利用する細高分解能(今回はナイキスト周波数)) | ||
| + | ● Minimum defocus cvalue(A): 5000 | ||
| + | (想定している最小デフォーカス) | ||
● Maximun defocus cvalue(A): 50000 | ● Maximun defocus cvalue(A): 50000 | ||
| + | (想定している最大デフォーカス) | ||
● Defocus step size(A): 500 | ● Defocus step size(A): 500 | ||
| + | (推定するデフォーカスの精度) | ||
● Amount of astigmatism(A): 100 | ● Amount of astigmatism(A): 100 | ||
| + | (想定している最大の非点量) | ||
● Estimate CTF on window size(pix): -1 | ● Estimate CTF on window size(pix): -1 | ||
| − | + | (正の値を設定した場合、プログラムは画像の中央を設定したサイズの正方形で切り取り、この部分の画像のみでCTFを推定します。 | |
| + | これは、余計な情報が含まれているスキャンされた画像に対して有効です。) | ||
| + | |||
| + | [[ファイル:relion-CTFest-CTFFIND.png|500px]] | ||
| + | |||
| + | CTFFIND3かCTFFIND-4.0.xを使うとき、【CTFFIND4】と【Gctf】タブは無視してください。マシンに応じて、複数のMPI処理を使用してプログラムを実行できます。5つのMPI処理を使うと、CTFFIND-4.0.8を使うと動作は5分程度かかります(CTFFIND-4.1では一つの処理に2分半かかり、GCTFを使うと数秒かかります)。一旦jobが終わると、出力の'''CtfFind/job003/Micrographs'''という出力ディレクトリの中にそれぞれの顕微鏡写真ごとに追加ファイルができます。.ctfファイルは算出されたパワースペクトルと適合したCTFモデルのMRCフォーマットの画像を含んでいます。.logファイルはCTFFINDかGCTFからの出力を含んでいます(CTFFIND3、4を使用した場合のみ、.comファイルはCTFFINDを起動するために使用したスクリプトを含んでいます)。 | ||
| + | |||
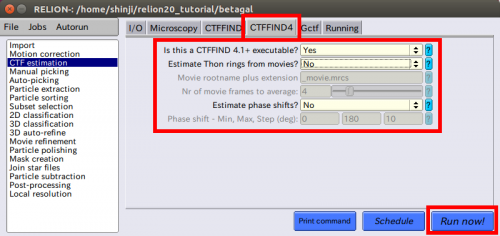
| + | CTFFIND-4.1+を使用している場合には、【CTFFIND4】タブで、次のように設定します。 | ||
| + | |||
| + | [[ファイル:relion-CTFest-CTFFIND4.png|500px]] | ||
| + | |||
| + | 本チュートリアルでは、すでにMotioncor2を使用して、モーションコレクションを行っており、またデータは位相版を使用せずに取得しているため、それぞれのパラメータは、 | ||
| + | ● Estimate Thon rings from movies?: No | ||
| + | (ドリフトがあるとフレーム間を足し合わせた画像ではThonRingが呆けてしまうため、フレーム毎にフーリエ変換したものを足し合わせます。) | ||
| + | ● Estimate phase shifts?: No | ||
| + | (位相板を用いてる場合の設定です。位相変化量の範囲を設定します。) | ||
| + | を選択します。 | ||
| + | |||
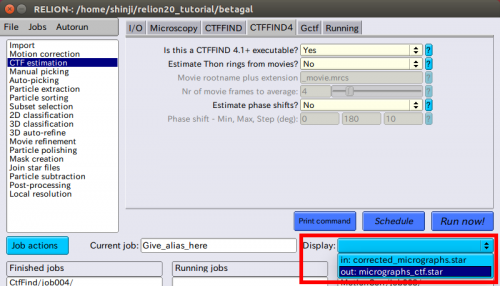
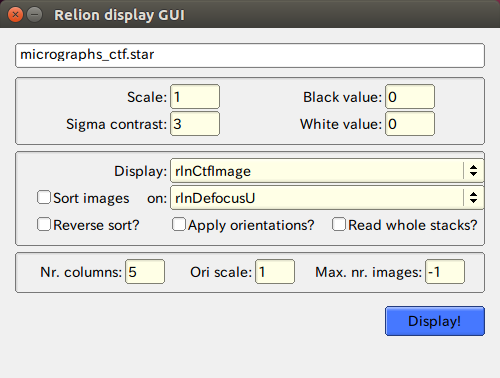
| + | それでは、計算されたThon-ring画像を見てみましょう。RELION操作画面の中央部右側の、【Display:】項目から、「out:micrographs_ctf.star」を選択します。 | ||
| + | |||
| + | [[ファイル:relion-CTFest-Display.png|500px]] | ||
| + | |||
| + | すると、次のような表示設定を行う画面が現れますので、各種パラメータを与え、【Display!】ボタンをクリックします。ここでは、コントラストや拡大・縮小を設定できます。 【Sort images】にチェックを入れて、【on:】でソート・キーを設定することで、デフォーカス、最大解像度、性能指数(FOM: Figure of Merit)などの順番の表示を並び替えることができます。 | ||
| + | |||
| + | [[ファイル:relion-CTFest-DisplaySettings.png|500px]] | ||
| + | |||
| + | 実際に、実際に表示される画像を次に示します。 | ||
| + | |||
| + | [[ファイル:relion-CTFest-Result.png|500px]] | ||
| + | |||
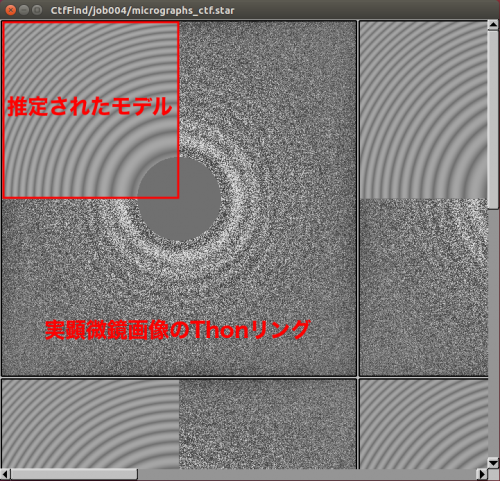
| + | 実験画像のThon-ring間のゼロ点とモデルのゼロ点は一致しているべきですが、一致していなければCTFの推定に失敗しています。もしCTFモデルが実験のThon-ringに十分に一致していない場合、それらの顕微鏡写真の.logファイルを削除して。【Finished jobs】リストからCtfFind/job003を選択し、parameter-panelの値を修正し、【Continue now】ボタンをクリックすことで、jobを再実行できます。存在しない.logファイルの顕微鏡写真のみが再処理されます。すべてのCTFモデルが十分に一致するまで実行できます。もしこれでも実行できない、または、不十分なThon-ringがあり、顕微鏡写真を削除すると決めた場合、【'''Subset selection'''】 jobタイプで破棄することができます。 | ||
| − | + | === 3.4 Manual particle picking(手動粒子抽出(座標の設定))=== | |
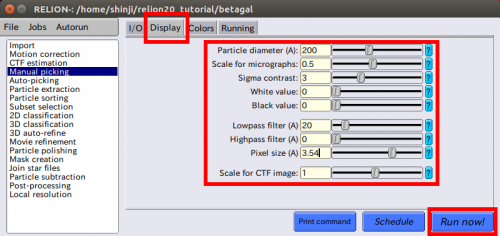
| + | 次のManual picking jobタイプでは(平均化された)顕微鏡写真の'''「粒子座標」'''を手動で選択して使います。今回のデータをよく説明できるように複数の顕微鏡写真を自分自身で選択するのがお薦めです。選択した粒子を使って、参照無2次元クラス平均を計算します。その後、これらの粒子は全データセットの粒子を自動検出するためのテンプレートとなります。 | ||
| − | + | ※ デフォーカスも異なっている複数枚の画像で、様々な投影角の粒子を選択するのがお薦めです。 | |
| − | + | 手動粒子検出は経験あるのみです!RELIONで手動で粒子の検出をするのが好みでなければ、Jude ShortのXIMDISP(任意の拡張子付きで)、XMIPP-2.4(任意の拡張子付きで)、Steven Ludtkeの E2BOXER.PY(.boxの拡張子で)の座標ファイル形式もサポートしています。もしこれらを使う場合、顕微鏡写真(動画)を取り込んだディレクトリと同じディレクトリにテキストファイルとしてまとめた座標ファイルを保存して下さい。そして、同じ顕微鏡写真のルートネームですが、異なる(suffix+)拡張子で保存されていることを確認してください。例えば、顕微鏡写真がMicrographs/006.mrcの時、Micrographs/006.boxやMicrographs/006_pick.starとなります。【Import】 jobタイプを使用し、Node type:を【2D/3D particle coordinates】に設定してください。Imput Files:の欄に整理されたファイルの拡張子に従ってlinux wildcardが含まれていることを確認してください。上記の例であれば、それぞれMicrographs/*.boxやMicrographs/*_pick.starになります。 | |
| − | + | Manual picking jobタイプの【I/O】タブではCtfFind/job003に作成されたmicrographs_ctf.starファイルを選択するために【Browse】ボタンを使用し、【Colors】タブは無視します。 | |
| − | + | ||
| − | + | [[ファイル:relion-ManualPicking-IO.png|500px]] | |
| − | + | 【Display】タブでは以下のように入力します。 | |
● Particle diameter(A): 200 | ● Particle diameter(A): 200 | ||
| − | ( | + | (これは単に顕微鏡写真に表示された円の直径を調節します。実際に切り出される大きさではありません。) |
● Scale for micrographs: 0.5 | ● Scale for micrographs: 0.5 | ||
| − | ( | + | (これは画面サイズによります。) |
● Sigma contrast: 3 | ● Sigma contrast: 3 | ||
| − | (顕微鏡写真はほとんど最適な”sigma- | + | (顕微鏡写真はほとんど最適な”sigma-contrast”で表示されています。3に設定した場合、黒が標準偏差の3倍低く、白が標準偏差の3倍高くなります。グレースケールは、黒から白への直線的に変化します。詳細は[http://www2.mrc-lmb.cam.ac.uk/relion/index.php/DisplayImages DisplayImages entry on the RELION wiki]を見てください。) |
● White value: 0 | ● White value: 0 | ||
| − | ( | + | (どの値を白にするか手動で設定してください。これが動作するには、Sigma contrastは0に設定されるべきです。) |
● Black value: 0 | ● Black value: 0 | ||
| − | ( | + | (どの値を黒にするか手動で設定してください。これが動作するには、Sigma contrastは0に設定されるべきです。) |
● Lowpass filter(A): 20 | ● Lowpass filter(A): 20 | ||
| − | ( | + | (ノイズのひどい顕微鏡写真の粒子をより見やすくする動作です。) |
● Highpass filter(A): 0 | ● Highpass filter(A): 0 | ||
| − | ( | + | (氷の厚さの違いなどに由来する顕微鏡写真全体の緩やかなグラデーションを取り除くのに役立つことがあります。) |
● Pixel size: 3.54 | ● Pixel size: 3.54 | ||
| − | ( | + | (これは粒子の直径や、low-pass filter、high-pass filterの計算に必要です。) |
● Scale for CTF image: 1 | ● Scale for CTF image: 1 | ||
| − | ( | + | (これは単に、【Run now!】後に表示されるウィンドウにおいて、各々の顕微鏡写真の【CTF】ボタンをクリックした時にThon-ring画像がどのくらい大きくなるかを調節します。) |
| − | + | [[ファイル:relion-ManualPicking-Display.png|500px]] | |
| + | |||
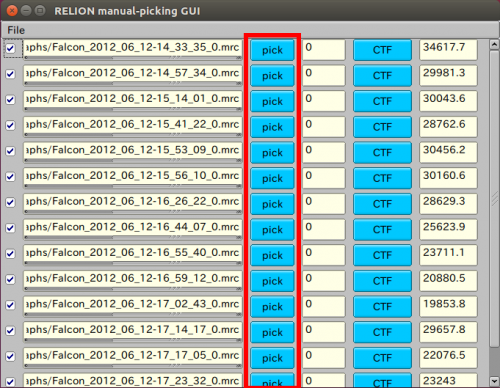
| + | 【Run now!】ボタンをクリックすることでjobが実行され、粒子ピッキングに用いる画像のリストが表示されます。 | ||
| + | |||
| + | [[ファイル:relion-ManualPicking-GUI.png|500px]] | ||
| + | |||
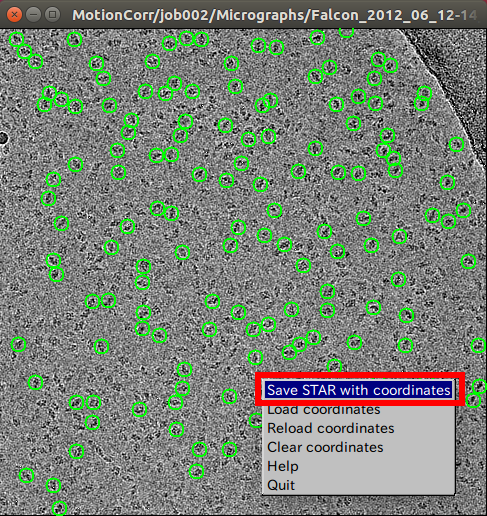
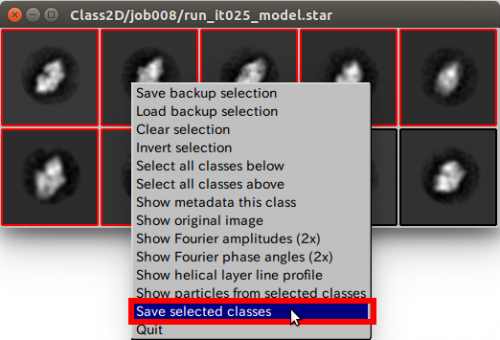
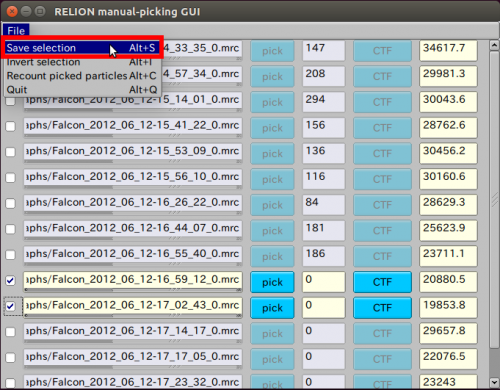
| + | 使用する、電子顕微鏡画像を【pick】ボタンを押して選択し、画像を表示した後、粒子を選択します。このマニュアルピッキング作業では、合計でおおよそ1000個の粒子を選択してください。左クリックで選択、中クリックで選択された粒子の削除、右クリックでメニュー(下図)が表示され、座標の保存ができます。【Continue now】ボタンをクリックし、Finished jobsからManualPick/job004を選択することによって、いつでも、メニューから保存した座標のSTARファイルに戻ることができます。 | ||
| + | |||
| + | [[ファイル:relion-ManualPicking-save.png|500px]] | ||
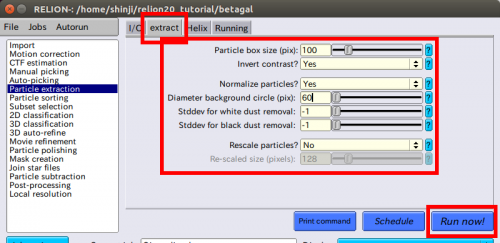
=== 3.5 Particle extraction(粒子の抽出)=== | === 3.5 Particle extraction(粒子の抽出)=== | ||
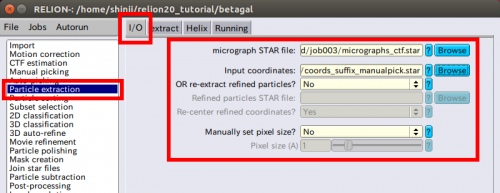
| − | + | 粒子をピックしたいすべての顕微鏡写真の座標ファイルを使って、対応する粒子を抽出し、必要なすべてのメタデータを【Particle extraction】jobタイプで取得することができます。【Particle extraction】 jobタイプの【I/O】タブで以下の設定をします。 | |
● Micrograph STAR file: CtfFind/job003/micrographs_ctf.star | ● Micrograph STAR file: CtfFind/job003/micrographs_ctf.star | ||
| − | ( | + | (ファイルの選択には【Browse】ボタンが使用できます。CTF補正の情報が入ったファイルを選択します。) |
● Coordinate-file suffix: ManulPick/job004/coords_suffix_manualpick.star | ● Coordinate-file suffix: ManulPick/job004/coords_suffix_manualpick.star | ||
| − | ( | + | (ファイルの選択には【Broswe】ボタンが使用できます。) |
● OR re-extract refined particles? No | ● OR re-extract refined particles? No | ||
| − | (これはRELION-2. | + | (これはRELION-2.0の新しいオプションです。STARファイルの粒子のみを再抽出するために、【2D classification】、【3D classification】、【3D auto-refine】 jobから_data.starファイルが使用できます。例えば、抽出時に粒子を前もって縮小していた場合、初めの分類後は元の大きさの粒子で精密化を実行したいときにも役に立ちます。RELION-1.4では面倒で、STARファイルの手動編集が必要でした。RELION-2.0ではすべてがこのオプションで自動でできます。) |
● Manually set pixel size? No | ● Manually set pixel size? No | ||
| − | ( | + | (CTF情報が含まれていない顕微鏡写真STARファイルを入力する時のみ必要です。) |
| − | + | [[ファイル:relion-ParticleExt-IO.png|500px]] | |
| + | |||
| + | 【extract】タブで、実際の粒子抽出のための値を以下のように設定します。 | ||
● Particle box size (pix): 100 | ● Particle box size (pix): 100 | ||
| − | ( | + | (これは常に偶数にすべきです!これが実際の大きさです。MRCのフォーマットはフーリエ空間が偶数にしか対応していません。) |
● Invert contrast? Yes | ● Invert contrast? Yes | ||
| − | ( | + | (これは黒を白に反転します。) |
● Normalize particles? Yes | ● Normalize particles? Yes | ||
| − | ( | + | (常に正規化します。) |
● Diameter background circle(pix): 60 | ● Diameter background circle(pix): 60 | ||
| − | ( | + | (粒子はバックグラウンド領域のすべてのピクセルでの平均値がゼロに、標準偏差が1になるように正規化されます。バックグラウンド領域とは、(縮尺変更前に)ピクセル中の指定された円の外側の部分すべてのピクセルと決められています。負の値を設定すると、粒子ボックスサイズの75%の値が使用されます。) |
● Stddev for white dust removal: -1 | ● Stddev for white dust removal: -1 | ||
● Stddev for black dust removal: -1 | ● Stddev for black dust removal: -1 | ||
| − | ( | + | (データを実際に見る場合、異常に白や黒よりの値のピクセルのみ除去されます。よく使うのは、stdevの値として5程度です。このデータセットでは異常な値のピクセルはありませんので、補正せずに、初期値の-1のままです。(すなわち、何もしません)) |
● Rescale particles? No | ● Rescale particles? No | ||
| − | ( | + | (縮小した粒子は計算速度が速くなります。そのため、処理速度を上げるために初期段階ではよく粒子を縮小します。初期の適切な粒子の分類の計算速度を上げるために使います。再構成がナイキスト周波数に近づくと、縮小せずに粒子を再抽出します。上記のように、RELION-1.4よりもRELION-2.0がとても速いです。) |
| − | + | [[ファイル:relion-ParticleExt-extract.png|500px]] | |
| − | 粒子はExtract/job005/Micrographs/ | + | 全ての自動検出された粒子を抽出するために後にも使用するものと同じjobタイプを使用するので、自動抽出と区別するためにmanualpickのようなエイリアスをjobにつけることをお勧めします。【Helix】タブは無視し、一つのMPIプロセスを使い、実行してください。 |
| + | |||
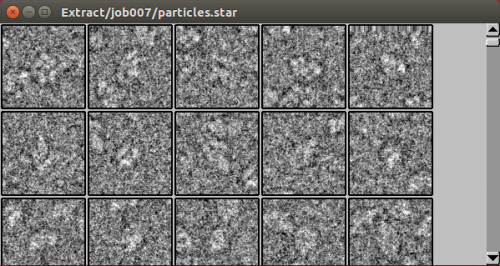
| + | 粒子はExtract/job005/Micrographs/という新しいディレクトリの中のMRCスタック(RELIONではいつも.mrcs拡張子をもつ)の中に抽出されます。【Display:】ボタンから'''out: particles.star'''に格納された抽出粒子を可視化することによって、すべてうまく進んでいるかの確認をすぐにすることはとても良い考えです。開いたウィンドウから【Disiplay!】ボタンを押して、実際に表示させると、次のようなディスプレイが現れます。 | ||
| + | |||
| + | [[ファイル:relion-ParticleExt-Display.png|500px]] | ||
| + | |||
| + | 画面上で右クリックでメニュを選択すると、すべての粒子の選択(Invert selection)や、すべての選択された粒子の(位置が合わせられてはいない、そのままの)平均の計算(Show average of selection)などができます。 | ||
=== 3.6 Making templates for auto-picking(自動検出のためのテンプレート作成)=== | === 3.6 Making templates for auto-picking(自動検出のためのテンプレート作成)=== | ||
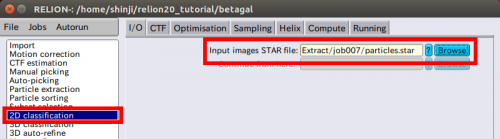
次に、全顕微鏡写真の自動検出のためのテンプレートを計算するために、jobタイプ【2D classification】を使用します。 | 次に、全顕微鏡写真の自動検出のためのテンプレートを計算するために、jobタイプ【2D classification】を使用します。 | ||
| − | 【I/ | + | 【I/O】タブ上で(【Browse】ボタンを使って)Extract / job005 / particles.starファイルを選択します。 |
| + | |||
| + | [[ファイル:relion-2Dclass-IO.png|500px]] | ||
| + | |||
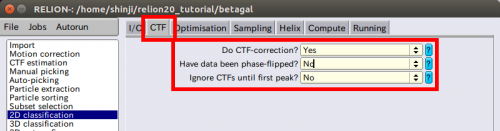
| + | 【CTF】タブ上で以下の操作を行ってください。 | ||
●Do CTF-correction? Yes | ●Do CTF-correction? Yes | ||
| − | + | (ベイズフレームワークの中で、全ての位相強度補正を行います。) | |
●Have data been phase-flipped? No | ●Have data been phase-flipped? No | ||
| − | + | (このオプションはRELION外でデータを事前処理した場合にのみ有効です。) | |
| − | + | ●Ignore CTFs until first peak? No | |
| − | + | (振幅補正によって低解像度成分が誤って強く与えられ、全ての粒子が非常に少ないぼけたクラスに分類されるような場合には、このオプションが役立ちます。) | |
| + | |||
| + | [[ファイル:relion-2Dclass-CTF.png|500px]] | ||
【Optimisation】タブの設定: | 【Optimisation】タブの設定: | ||
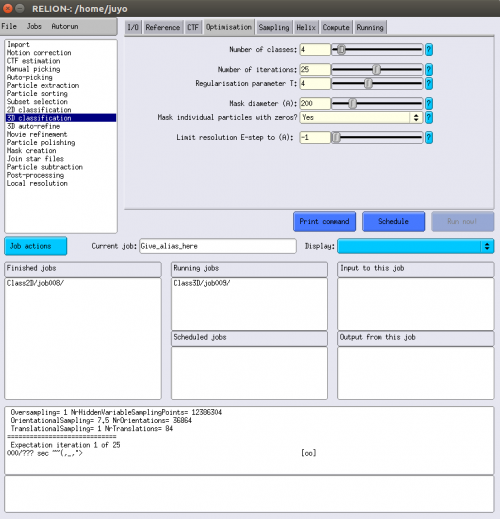
●Number of classes: 10 | ●Number of classes: 10 | ||
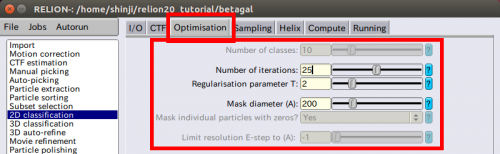
| − | クライオ電子顕微鏡(cryo-EM) | + | (クライオ電子顕微鏡(cryo-EM)では、少なくともクラス当たり約100個の粒子を使用するのが望ましいです。ネガティブ染色法の場合、1クラスで20~50程のより少ない粒子を使用します。ある程度の分解能を得るには、フーリエ空間が重なり合うことが望ましいため、多くの粒子が必要です。) |
●Number of iterations: 25 | ●Number of iterations: 25 | ||
| − | + | (これはほとんど変えた事はほとんどありません。) | |
●Regularisation parameter T: 2 | ●Regularisation parameter T: 2 | ||
| − | + | (Tの定義については[[#ref10|[10]]]を参照してください。Tが大きいほどシグナルに重きを置くことになり、結果として高分解能側が強調されます。クライオ電子顕微鏡(cryo-EM)において、2次元分類ではT=2-3が、3次元分類ではT=3-4の値が一般的に使用されます。ネガティブ染色の場合、これよりもより小さい値の方が良い場合もあります。一般的にクラス平均が非常にノイズが多く現れた場合は、一旦Tの値を減らしてください(結果として、多くの場合ローパスフィルタがかかったようになります)。もし、クラス平均の分解能が低すぎる場合はTの値を増やしてください。重要なことは、高分解能側のノイズのオーバーフィッティング(過学習)してしまっていることに気がつくことです。) | |
●Mask diameter (A): 200 | ●Mask diameter (A): 200 | ||
| − | + | (このマスクは全ての2次元クラス平均に適用されます。また、粒子画像の溶媒ノイズや隅にあるような隣接した粒子を除去するのに使われます。ノイズが大きい溶媒と隣り合った粒子が位置合わせの際に干渉してしまうかもしれないため、直径は小さく保ちたいでしょうが、他方では、クラス平均からシグナルを切り捨てたくないため、粒子の大きさよりも直径が大きいか確かめる必要があります。) | |
●Mask individual particles with zeros? Yes | ●Mask individual particles with zeros? Yes | ||
| + | (Noの場合は、乱数で埋めます。大きな粒子で高分解能での処理が可能な場合には、こちらのほうが良い結果を与える場合もあります。小さな粒子では0のほうがよいでしょう。) | ||
●Limit resolution E-step to (A): -1 | ●Limit resolution E-step to (A): -1 | ||
| − | + | (正の値を与えた場合、この値を超える周波数を位置合わせの際に利用しません。これはノイズによる過学習を防ぐのにも役立ちます。ここではそれは必要ではありませんが、10-15Aで設定されている可能性があります。難しい分類、すなわち非常にノイズの多いデータの場合は、分解能を制限することによって問題が解決することが多いです。) | |
| + | |||
| + | [[ファイル:relion-2Dclass-Optimisation.png|500px]] | ||
| + | |||
| + | 【Sampling】タブでは初期値を変更することは、ほとんどありません。 | ||
| + | ●Perform image alignment? Yes | ||
| + | (粒子毎の位置合わせを実行するかどうかを指定します。) | ||
| + | ●In-plane angular sampling 5 | ||
| + | (面内の回転角度を指定します。単位はdeg.です。粒子の大きさに依存します。) | ||
| + | ●Offset search range (pix) 5 | ||
| + | ●Offset search step (pix) 1 | ||
| + | (粒子の位置合わせの平行移動の最大範囲と刻み巾を示しています。) | ||
| + | いくつかの正20面体ウイルスでは、より細かな角度ごとでのサンプリングが必要かもしれませんが、ほとんどの場合は5°ごとの角度サンプリングで十分です。その場合、最初に5°ごとのサンプリングを25回反復実行し、2°ごとのサンプリングを、さらに5回反復して(【Continue now】ボタンを使って)同じ実行を続けます(Number of iterations: 30 【Optimisation】タブで設定)。今回のデータセットの場合、この操作は全く必要ありません。前に何らかの形で失敗したjobを、【Continue now】ボタンを使用して再開することもできます。この場合、どのパラメータも変更する必要は無いでしょう。【2D classification】、【3D classification】、または【3Dauto-refine job】を続行するには、常に【I / O】タブの【Continue from here:】に_optimiser.starファイルを指定する必要があります。 | ||
| + | |||
| − | |||
【Helix】タブは無視し、【Compute】タブで以下のように設定を行います。 | 【Helix】タブは無視し、【Compute】タブで以下のように設定を行います。 | ||
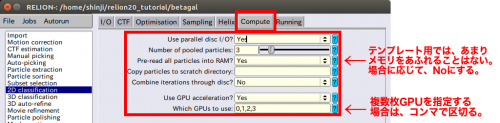
●Combine iterations through disc? No | ●Combine iterations through disc? No | ||
| − | この方法で、全てのMPIノードは、各反復の終わりにネットワークを介してデータを結合します。ネットワークがボトルネックであるか何らかの原因で問題が発生している場合は、このオプションをNoに設定できます。その場合、すべてのMPIノードはデータをディスクに書き込み / 読み出します。 | + | (この方法で、全てのMPIノードは、各反復の終わりにネットワークを介してデータを結合します。ネットワークがボトルネックであるか何らかの原因で問題が発生している場合は、このオプションをNoに設定できます。その場合、すべてのMPIノードはデータをディスクに書き込み / 読み出します。) |
| + | |||
●Use parallel disc I/O? Yes | ●Use parallel disc I/O? Yes | ||
| − | + | (この場合、すべてのMPIスレーブは自分の粒子をディスクから読み込みます。 | |
| − | + | 高速(並列)ファイルシステムを使用している場合は、このオプションを使用してください。 並列ではないファイルシステムは、複数のMPIノードからの並行したアクセスを処理できない場合があることに注意してください。そのような場合、このオプションをNoに設定するべきです。この時、マスターMPIノードだけが粒子を読み込み、ネットワーク経由でMPIスレーブにデータを送信します。) | |
| + | |||
●Number of pooled particles: 3 | ●Number of pooled particles: 3 | ||
| − | + | (粒子はMPIスレーブによって個々のバッチ処理されます。各バッチの中で、粒子画像のスタッは、ディスクアクセス時間を改善するために1回だけ開閉されます。1つのバッチのすべての粒子画像は一緒にメモリに読み込まれます。これらのバッチサイズは、少なくともスレッド毎に1つの粒子が読み込まれます。nr_pooled_particlesパラメータは、スレッドごとに読み込まれる粒子の数を制御します。3に設定され、1つが8つのスレッドを使用する場合、3x8 = 24個の粒子のバッチが一緒に読み込まれます。これにより、ディスクアクセス、特にディスクアクセスのメタデータ処理が問題となるシステムでのパフォーマンスが向上する可能性があります。わずかなRAM(ランダム・アクセス・メモリ)の使用量の増加をもたらします。) | |
●Pre-read all particles into RAM? Yes | ●Pre-read all particles into RAM? Yes | ||
| − | Yesを選択すると、すべての粒子画像がコンピュータのメモリに読み込まれ、ディスクアクセスが遅いシステムでの計算が大幅に高速化されます。ただし、使用可能なRAMの量には注意が必要です。粒子は倍精度で読み込まれるため、N個の粒子をRAMに読み込むには(N *box size*box size* | + | (Yesを選択すると、すべての粒子画像がコンピュータのメモリに読み込まれ、ディスクアクセスが遅いシステムでの計算が大幅に高速化されます。ただし、使用可能なRAMの量には注意が必要です。粒子は倍精度で読み込まれるため、N個の粒子をRAMに読み込むには(N *box size*box size* 8 /(1024 * 1024 * 1024))ギガバイトかかることになります。並列ディスクI / Oが「Yes」で選択されている場合、すべてのMPIスレーブは粒子すべて読み込みます。並列ディスクI / Oが「No」で選択されている場合、マスタのみがすべての粒子をRAMに読み込み、精密化の反復中にMPIスレーブにそれらの粒子を送信します。) |
| + | |||
●Use GPU acceleration? Yes | ●Use GPU acceleration? Yes | ||
| − | + | (これらに合ったGPUをお持ちの場合、このjobは高速化されます。) | |
| + | |||
●Which GPUs to use: 0 | ●Which GPUs to use: 0 | ||
| − | これを空のままにすると、プログラムは使用するGPUを特定しようと試みます。0または1を使ってGPU IDを入力してください。複数のMPIプロセッサを使用している場合、指定したGPUで各MPIプロセスを実行できます。異なるMPIプロセスのGPU IDは、コロンで区切られます。 0:1:0:1はGPU 0でMPIプロセス0と2を実行し、MPIプロセス1と3はGPU 1で実行されます。 | + | (これを空のままにすると、プログラムは使用するGPUを特定しようと試みます。0または1を使ってGPU IDを入力してください。複数のMPIプロセッサを使用している場合、指定したGPUで各MPIプロセスを実行できます。異なるMPIプロセスのGPU IDは、コロンで区切られます。 0:1:0:1はGPU 0でMPIプロセス0と2を実行し、MPIプロセス1と3はGPU 1で実行されます。) |
| − | + | ||
| − | + | [[ファイル:relion-2Dclass-Compute.png|500px]] | |
| + | |||
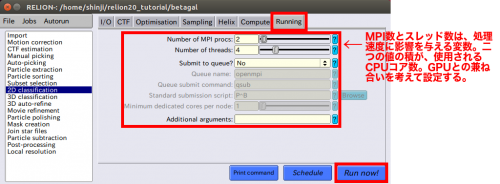
| + | 【Running】タブで、Number of MPI processors(MPIプロセスの数)とNumber of threads(スレッドの数)を指定します。要求されたCPUまたはコアの総数は、2つの値の積になります。 スレッドは、より効率的なRAM使用に関して利点がありますが、MPIの並列化はスレッドよりも優れています。多くの場合、【3D classification】と【3D auto-refine】jobでは、各(マルチコアの)計算ノードで使用可能なRAMを共有するために、多くのスレッドを使用することが必要になります。【2D claasification】(2D分類)ではメモリを消費しないので、スレッドがあまり必要ない場合があります。しかし、MPIプロセス間通信(拡張のボトルネック)が、多くのスレッドを実行する場合に比べて、速度を制限する場合もあります。さまざまなクラスタによって異なりますので、これらのパラメータを使用して、それぞれのコンピュータのセットアップによって最適なパフォーマンスを得る必要があります。 | ||
| + | |||
| + | [[ファイル:relion-2Dclass-Running.png|500px]] | ||
| + | |||
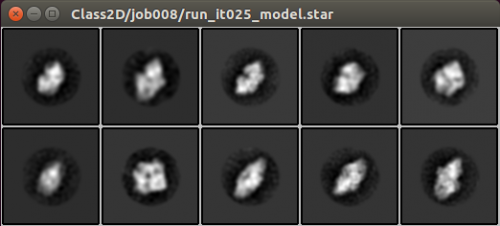
| + | 多くの【2D classification】jobを実行するので、例えばmanualpickのような意味のあるエイリアスを使用することが賢いやり方です。GPUアクセラレータまたは(合理的なサイズの)クラスタを使用している場合、このジョブは数分で終了することでしょう。そうではなくて、コア数が少ないデスクトップの場合には、15〜20分かかります。 | ||
| + | |||
| + | 計算が終了した後は、[Display:]ボタン上でrun_it025_model.starを選択し、得られたクラス平均を見ることができます。ポップアップウィンドウでは、rlnClassDistribution(クラス粒子の個数)(【reverse order】をチェックして、昇順の代わりに降順の並び替え)、またはrlnAccuracyRotationsに基づくなど特定の順序でクラス平均を調べ、選択してみることができます。 | ||
| + | |||
| + | この結果を実際に確かめたものを以下に示します。いままでの処理が正しく出来ているかを確認してください。 | ||
| + | |||
| + | [[ファイル:relion-2Dclass-Display.png|500px]] | ||
=== 3.7 Selecting templates for auto-picking (自動検出のためのテンプレート選択)=== | === 3.7 Selecting templates for auto-picking (自動検出のためのテンプレート選択)=== | ||
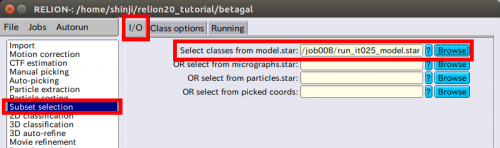
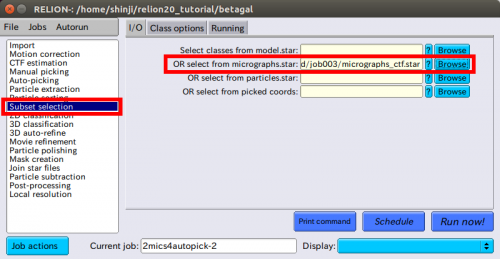
| − | RELION-1. | + | RELION-1.4と違い、RELION-2.0では【Display:】ボタンで適切なクラスを選択することができません。これは[Subset selection]jobタイプ上で行われます。Select classes from model.starと書かれた行の横にある【Browse】ボタンを利用して、【I/O】タブ上のClass2D/manualpick/run_it025_model_.starファイルを選択してください。 |
| − | + | [[ファイル:relion-SubsetSelection-IO.png|500px]] | |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | ※ これは、パイプライン処理を全てに与えるためです。将来、この選択も自動化されることが予定されています。 | |
| + | |||
| + | 【Class opitions】タブ上で下記のように入力してください。 | ||
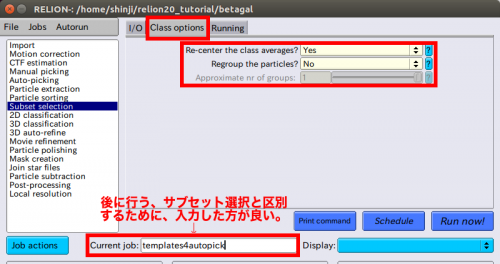
| + | ●Re-center the class averages?: Yes | ||
| + | これは、relion-2.0の新しいオプションで、2次元クラス平均の自動センタリングが可能です。画像のセンタリングはその重心を基準としており、粒子の白(黒ではない)の部分をもとに計算されます。センタリングされたテンプレートが自動検出中に必要となる一方で、2D classification実行時ではクラス平均はセンタリングされないかもしれないので、再センタリングがしばしば必要となります。 | ||
| + | ●Regroup the particles? No | ||
| + | これは、それぞれの電子顕微鏡写真にある粒子が非常に少ない場合に便利なオプションです。そのような場合、スペクトルノイズ強度とスケールファクターの推定が不安定になることがあります。デフォルトでは、スケールファクターの推定は顕微鏡写真ごとにそれぞれ計算されます。このオプションは計算実行時、複数の顕微鏡写真の粒子を一緒に計算してグループ化することが可能です。RELIONは、クラス分類や自動精密化実行の際にグループがとても小さくなっている場合に警告を出します。 | ||
| + | |||
| + | templates4autopickのようなエイリアスはとてもいいアイディアです。 | ||
| + | |||
| + | [[ファイル:relion-SubsetSelection-ClassOptions.png|500px]] | ||
| + | |||
| + | rlnClassDistributionカラムに基づいたクラス平均を再び求める可能性があります。代表となりそうな、見た目が異なる粒子のクラス平均を4〜6つ選択してください。見た目が似ているものを繰り返し選択したり、よくないクラス平均を含めたりしないでください。選択するにはクラス平均上で左クリックしてください。右クリックメニューからSave selected classes オプションを使うことで、選択したクラス平均を保存できます。 | ||
| + | |||
| + | [[ファイル:relion-SubsetSelection-Display.png|500px]] | ||
=== 3.8 Auto-picking(自動検出)=== | === 3.8 Auto-picking(自動検出)=== | ||
| − | + | 【Auto-picking】jobタイプでは、テンプレートとして選択された2次元クラス平均を使います。 | |
| − | + | しかし、全ての顕微鏡写真で自動検出を実行する前に、【'''autopicking'''】タブ上で、【'''Picking threshold'''】、【'''Minimum inter-particle distance'''】、【'''Maximum stddev noise'''】の3つの主なパラメーターを最適化する必要があります。 | |
| + | |||
| + | そのため、まずはテストとして、選択された数枚の顕微鏡写真に対して行います。そのため、Subset selection】jobタイプを利用して、Auto-Pickingを適用する数枚の顕微鏡写真を選択し、この2〜3枚の顕微鏡写真を含むSTARファイルを作成します。 | ||
| + | |||
| + | 【'''Subset selction'''】jobタイプで行うには2つの方法があります。 | ||
| + | |||
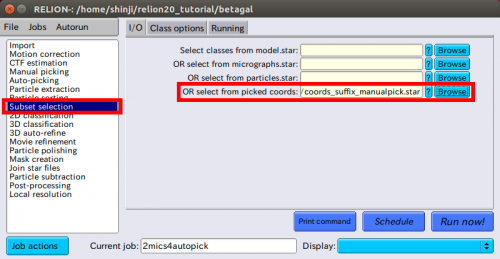
| + | (1) Auto-Pickingテスト用顕微鏡画像選択方法その1:"OR select from picked coords:"を使う | ||
| + | |||
| + | OR select from picked coords: の行からManualPick/job004/coords_suffix_manualpick.starを選択。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-SubsetSelection-IO-1.png|500px]] | ||
| + | |||
| + | パラメータを入力後、【Run now!】をクリックし、顕微鏡画像の選択ボックスを表示させます。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-SubsetSelection-Display-1.png|500px]] | ||
| + | |||
| + | この状態で、Auto-Pickingのパラメータ調整テストに用いたい画像を選びます。初期状態では、すべての顕微鏡画像にチェックが入っている状態ですので、"Invert selection"を用いると、一度にすべての画像のチェックを外せるので便利です。使用する画像を選び終わったら、"Save selection"をクリックし、STARファイルを生成します。 | ||
| + | |||
| + | (2)Auto-Pickingテスト用顕微鏡画像選択方法その2:"OR select from micrographs.star:"を使う | ||
| + | |||
| + | もう1つの方法は、[Subset selection]jobタイプの【I/O】タブからOR select from micrographs.star: の行からCtfFind/job003/micrographs/ctf.starを選択します。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-SubsetSelection-IO-2.png|500px]] | ||
| + | |||
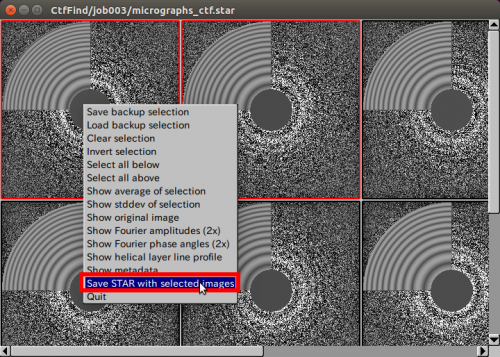
| + | インプット情報を入力後、【Run now!】をクリックすると、ディスプレイ設定がポップアップしますので、適当な値を入力し、表示させます。表示させた画像から、左クリックで顕微鏡写真を選択し、右クリックメニューからSave STAR with selected imagesを選択して下さい。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-SubsetSelection-Display-2.png|500px]] | ||
| − | + | どちらの方法を選んでも結構ですが、Job名へ"2mics4autopick"のようなエイリアスをつけたほうがよいかもしれません。これは、前項でのSubset selectionと区別をつけるためです。 | |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | 次に、上述の方法で作成した2〜3枚の電子顕微鏡画像を含む、STARファイルに対してAuto-pickingを行っていきます。 | |
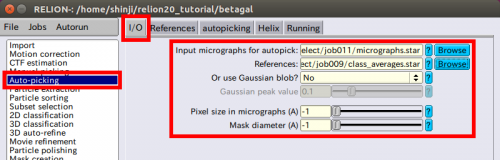
| + | 【Auto-picking】jobタイプの【I/O】タブ上で以下のように設定します。 | ||
● Input micrographs for autopick: Select/2mics4autopick/micrographs.star | ● Input micrographs for autopick: Select/2mics4autopick/micrographs.star | ||
| − | ● References: Select/templates4autopick/ | + | ● References: Select/templates4autopick/class_averages.star |
● Pixel size in microgfraphs (A): -1 | ● Pixel size in microgfraphs (A): -1 | ||
(入力STARファイルがCtfFind jobからピクセルサイズ情報を得ていますので無視されます) | (入力STARファイルがCtfFind jobからピクセルサイズ情報を得ていますので無視されます) | ||
| − | + | [[ファイル:relion-AutoPicking-pre-IO.png|500px]] | |
| + | |||
| + | 【Refernces】タブでは以下のように設定します | ||
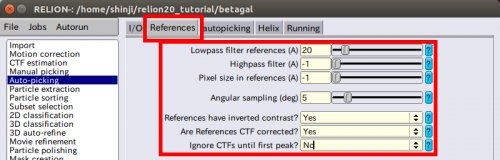
● Lowpass filter references (A): 20 | ● Lowpass filter references (A): 20 | ||
| − | ( | + | ("Einstein-from-noise"のアーティファクトを避けるために、データから得ようとしている最終的な分解能よりも'''大幅に低い'''ローパスフィルターを使用することは非常に重要です) |
● Highpass filter (A): -1 | ● Highpass filter (A): -1 | ||
| − | ( | + | (たとえば200などの正の値を渡した場合、リファレンスは顕微鏡写真はautopickingよりも高いハイパス・フィルターが適用されます。これは強いグレースケール勾配(氷の厚さなどに依存)が顕微鏡写真にかかっているときに役立ちます) |
● Pixel size in references (A): -1 | ● Pixel size in references (A): -1 | ||
| − | ( | + | (負の値を渡した場合、入力した顕微鏡写真と同じスケールであると認識します。そうでない場合、例えば粒子を抜き出す上でのリファレンスを作るために使用される粒子をリスケールする必要があるので、正しいピクセルサイズの正の値をここのリファレンスに追加します。) |
● Mask diameter (A): 200 | ● Mask diameter (A): 200 | ||
| − | ( | + | (【2D classification】 jobタイプのときに使った値と同じ値が使えます) |
● Angular sampling (deg): 5 | ● Angular sampling (deg): 5 | ||
| − | ( | + | (この値はどんな場合でもほとんど正常に動くと思われます) |
● References have inverted contrast? Yes | ● References have inverted contrast? Yes | ||
| − | ( | + | (顕微鏡写真の中にある粒子は黒い(CTF補正がかかっていないため)ので、使用するリファレンスは白を利用します) |
● Are References CTF corrected? Yes | ● Are References CTF corrected? Yes | ||
| − | ( | + | (2Dクラス平均を行った際にCTF補正をしたため) |
● Ignore CTFs until first peak: No | ● Ignore CTFs until first peak: No | ||
| − | ( | + | (【2D classification】jobでもリファレンス作製のために使用した場合のみこのオプションを使って下さい) |
| − | + | [[ファイル:relion-AutoPicking-pre-References.png|500px]] | |
| + | |||
| + | 【autopicking】タブでは以下のように設定します | ||
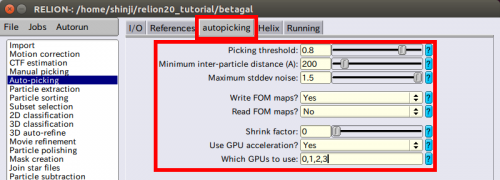
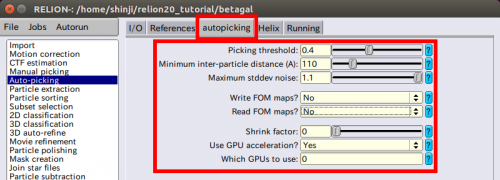
●Picking threshold: 0.8 | ●Picking threshold: 0.8 | ||
| − | ( | + | ('''主要調整パラメータ1''':これはpeak-searchアルゴリズムのためのFOMマップ中のしきい値です。この値よりも低いFOMの粒子は検出されません) |
● Minimum inter-particle distance (A): 200 | ● Minimum inter-particle distance (A): 200 | ||
| − | ( | + | ('''主張調整パラメータ2''':これは、隣接する2つの粒子間の最大許容距離です。反復的クラスタリングアルゴリズムはこの距離よりも互いに近い粒子を取り除きます。これの効果的な値は粒子直径の50%から60%の範囲であることが多いです) |
| − | + | ||
● Maximum stddev noise: 1.5 | ● Maximum stddev noise: 1.5 | ||
| − | ( | + | ('''主張調整パラメータ3''':これは炭素領域や非常に汚染されてる特徴が見て取れる領域のピッキングを防止するのに有効です。標準化された顕微鏡写真のバックグラウンド標準偏差がこの値より高い領域のピークは無視します。効果的な値は1.0から1.2の範囲であることが多いです。バックグラウンド標準偏差が高いため、ピークを除去するためにこの機能をオフにするには値を-1にして下さい。) |
● Write FOM maps? Yes | ● Write FOM maps? Yes | ||
(下の説明参照) | (下の説明参照) | ||
| 行338: | 行606: | ||
(下の説明参照) | (下の説明参照) | ||
● Shrink factor: 0 | ● Shrink factor: 0 | ||
| − | ( | + | (これを0に設定することで、自動ピッキングアルゴリズムは顕微鏡写真をリファレンスにおいてのローパスフィルターの解像度にまで縮小します。これはチュートリアルを手軽にできるように、実行をとても速く、要求メモリも軽くしてくれます。0から1の間の値は顕微鏡写真のサイズの画分の結果になります。これはshrink=1(縮小しない)にするよりもいくらか正確さの低い検出になるということに注意して下さい。これの新要素についての詳細な説明は次のサブセクションでします) |
| − | + | ||
● Use GPU acceleration? Yes | ● Use GPU acceleration? Yes | ||
| − | ( | + | (適切なGPUを備えている場合のみ使って下さい) |
● Which GPUs to use: | ● Which GPUs to use: | ||
| − | ( | + | (これを空白のままにした場合、このプログラムはどのGPUを使えばよいのか判断しようとします。しかし、もし使いたいGPUのIDが例えば0や1などと、はっきりと分かる場合は、この項目に入力して下さい。複数のMPI-processorsを使う場合(そうでない場合も)は、それぞれのMPI processを特定のGPUで動かすことができます。それぞれのMPI processesに割り振るGPU IDは0:1:0:1など、コロンで分けられています。0:1:0:1とはMPI processの0と2はGPU 0上を、MPI processの1と3はGPU 1上で動作しているという意味です。 |
| − | + | [[ファイル:relion-AutoPicking-pre-autopicking.png|500px]] | |
| − | + | ||
| − | + | ||
| − | + | 【Helix】タブは無視して下さい。 | |
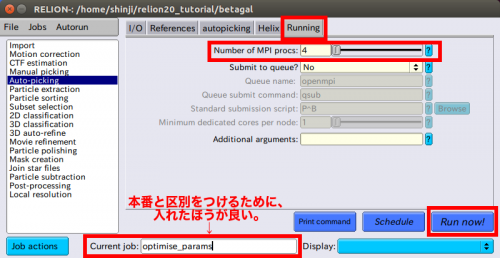
| − | + | 【Running】タブでひとつのMPI processorを使って動かして下さい。おそらくoptimise_paramsのようなエイリアスが有効かもしれません。チュートリアルでは2000×2000の顕微鏡写真を使っているので、1枚の顕微鏡写真につき、計算に5分ほどかかります。より一般的な4000×4000の顕微鏡写真を使った場合は、一般的に顕微鏡写真とリファレンスごとにだいたい5分ほどかかります。GPU-accelerationを使った場合は。10秒以内に動作完了します。そのため、FOMマップの書き込みと読み込みがあまり関係なくなります。 | |
| + | |||
| + | [[ファイル:relion-AutoPicking-pre-Running.png|500px]] | ||
| + | |||
| + | この計算の高負荷な部分は、a probability-based figure-of-merit(それぞれの回転したリファレンスと顕微鏡写真の中の全ての場所での相互相関関数に関連した値)の計算をすることです。その後、上で設定したしきい値と距離の最小値を利用したより高速なpeak-detectionアルゴリズムが実行されます。これらのパラメータは最適化する必要があるため、プログラムは【autopicking】タブで指定されているようにいわゆるFOMマップを作成します。これらはリファレンスごとに2つの大きなサイズ(顕微鏡写真のサイズ)のファイルになります。ハードディスク動作中のI/O障害を避けるために、FOMマップに書き出すときに自動検出プログラムは順次に動作するようにできます(従って上記で示した様に、一つのMPI processorで実行するべきです。) | ||
| + | |||
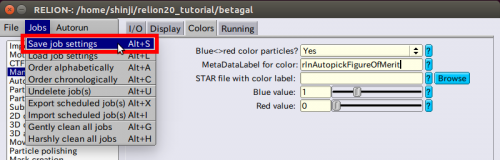
| + | FOMマップをディスクに書き出せば、より高速に検出するパラメータになるように最適化するために使用されます。最初に、動作させ終えたばかりのjobで【Display:】ボタンから coords_sufix_autopick オプションを使って、現在の設定で自動検出された粒子を調べます。ディスプレイウィンドウは、最後に実行した【Manual picking】jobのパラメーター(サイズやシグマコントラスト、カラーなど)を利用することに注意しておいて下さい。実際に【Manual picking】jobタイプでこれらのパラメーターを変更でき、左上の jobs メニューから Save job settings オプションで設定を保存できます。 | ||
| + | |||
| + | FOMの状態を表示するために、【Colors】タブで次のように設定したあとに、これを行って下さい。 | ||
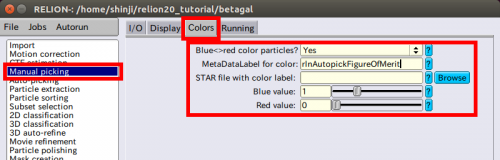
● Blue<>red color particles? Yes | ● Blue<>red color particles? Yes | ||
| − | ● MetaDataLabel for color: | + | ● MetaDataLabel for color: rlnAutopickFigureOfMerit |
| + | (選択出来ないため、書き込む必要があります。) | ||
● STAR file with color label: | ● STAR file with color label: | ||
| − | + | (この部分は空にしておいて下さい。) | |
● Blue value: 1 | ● Blue value: 1 | ||
● Red value: 0 | ● Red value: 0 | ||
| − | + | [[ファイル:relion-AutoPicking-ManualPicking-Colors.png|500px]] | |
| − | Finished | + | 条件を入力した後に、この条件設定を保存します。保存するには、左上の【Jobs】の【Save job settings】を選びます。 |
| + | |||
| + | [[ファイル:relion-AutoPicking-ManualPicking-Colors-save.png|500px]] | ||
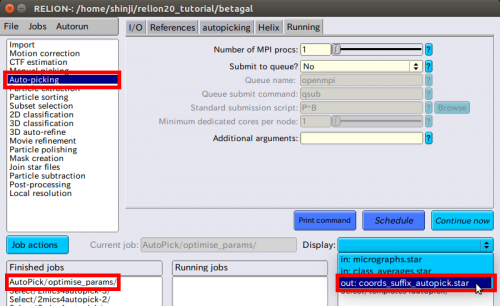
| + | |||
| + | その後、【AutoPicking】のジョブへと戻ります。今まで行っていたJobを読み出すために、【Finished jobs】の中から、【AutoPick/optimise_params】を選択します。【Auto-picking】が選ばれていることを確認した後に、【Display:】部分で、【out:coords_suffix_autopick.star】を選択し、自動ピッキング結果の電子顕微鏡画像を読みだしてみます。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-Display.png|500px]] | ||
| + | |||
| + | 顕微鏡画像を表示させると、少しわかりづらいかもしれませんが、粒子を選択している円の色が異なります。これは、FOM値に応じて色分けされており、青い円になるほどFOM値が高く、赤い円になるほどFOM値が低くなります。わかりづらいときは、上述のBlue value/Red valueを狭い範囲に設定して下さい(例えば、0.8/0.5) | ||
| + | |||
| + | この結果から、粒子をより多くか、少なく検出するか(すなわち、しきい値を小さくするか大きくするか)と、近くの粒子を同一とするか異なるものとするか(すなわち、粒子間の最小距離値の設定)を決定します。次のステップに進む間に、両方の顕微鏡写真の表示ウィンドウを開いたままにすることができます。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-pre-Display-FOM.png|500px]] | ||
| + | |||
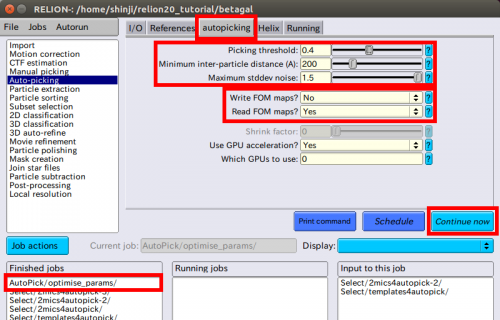
| + | [Finished jobs]から、AutoPick/optimise_params jobを選択して下さい。そして【autopicking】タブ上でパラメーターを変更して下さい。次のように変更して下さい | ||
● Write FOM maps? No | ● Write FOM maps? No | ||
● Read FOM maps? Yes | ● Read FOM maps? Yes | ||
| − | + | 【Continue now】ボタンをクリックすることで実行した時、全てのFOMの再計算の代わりに、以前書きだされたFOMマップをディスクから再読み込みします。新しい座標のその後の計算は、数秒で実行されます。 | |
| − | + | [[ファイル:relion-AutoPicking-FOM-change.png|500px]] | |
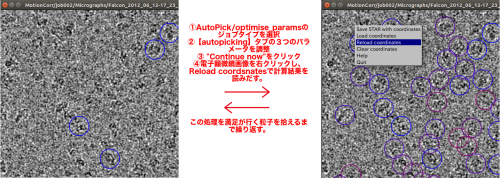
| + | |||
| + | この後に、新しい座標セットを読み込むために、顕微鏡写真の表示ウィンドウ上で右クリックメニューから Reload coordinates を選択して下さい。この動作を繰り返して、autopickingのパラメータを調節していきます。 | ||
| + | |||
| + | [[ファイル:relion-autopicking-optimiseParams.png|500px]] | ||
| + | |||
| + | 3つのパラメータをすべて使いこなして、検出結果をどのように変更するかを確認してください。 | ||
| + | |||
| + | 自動ピッキングに用いるパラメータが決まった後は、すべての電子顕微鏡画像に対してこの処理を行います。そのために、Auto-Pickingの読み込み画像を変更します。パラメータチューン用のジョブとは異なるジョブを作るために、ジョブタイプの【Auto-picking】をクリックし、あらたなジョブを作成します。その後に、【I/O】タブ上で、入力した顕微鏡写真のSTARファイルをすべての画像ファイルへと変更します。これは、【CTF estimation】jobで作られたファイル、【CtfFind/job003/micrographs_ctf.star】を指定してください。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-IO.png|500px]] | ||
| + | |||
| + | 【autopicking】タブで、先ほど求めたパラメータで設定を行います。ここでは、例として下記のようなパラメータを用います。 | ||
● Picking threshold: 0.4 | ● Picking threshold: 0.4 | ||
● Minimum inter-particle distance (A): 110 | ● Minimum inter-particle distance (A): 110 | ||
| 行373: | 行673: | ||
● Write FOM maps? No | ● Write FOM maps? No | ||
● Read FOM maps? No | ● Read FOM maps? No | ||
| − | |||
| − | |||
| − | + | [[ファイル:relion-AutoPicking-autopicking.png|500px]] | |
| + | |||
| + | ここで、jobは並列に(FOMマップが書き出せされてないのであれば)実行されるかもしれません。【Running】タブで、自動検出を実行するコアの数を指定します。MPIプロセッサの最大有効数は、入力したSTARファイルの顕微鏡写真の数となります。 | ||
| + | |||
| + | [[ファイル:relion-AutoPicking-Running.png|500px]] | ||
| + | |||
| + | FOMマップを読み込み書き出しをするかどうかによって、【Continue now】ボタンの動作の仕方に重要な違いがあることに注意して下さい。FOMマップを読み込みまたは書き出しをする場合には、【Continue now】ボタンをクリックすることで、プログラムは(一般的には少数の)全ての入力した顕微鏡写真を再度検出します。しかし、FOMマップの読み込みまたは書き込みをしない場合、すなわち、自動検出を全ての顕微鏡写真で行う2回目のjobでは、【Continue now】ボタンをクリックすることで、まだ自動検出を行っていない顕微鏡写真のみ再検出します。これはスケジュール化されたjobの反復にて有効です。たとえば、顕微鏡検査セッションの間の高速データ処理などです。また、詳細は2.3のセクションを見て下さい。もし再検出を全ての顕微鏡写真について、新しい値のセットを用いてしたい場合は(仕上がっていない顕微鏡写真だけ動作させる代わりとして)、【Run now!】ボタンの代わりに、左にあるjobタイプブラウザ上で【Auto-picking】jobタイプをクリックして下さい。そのとき、新しい出力ディレクトリが作成されます。 | ||
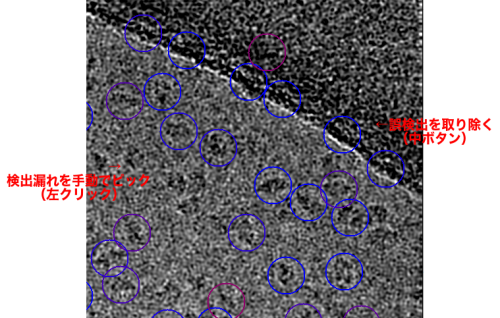
| − | + | 【Display:】ボタンからcoords_suffix_autopick オプションをクリックすることで、結果を再確認することができます。手動で全ての顕微鏡写真を調べて誤検知を取り除くのを好む方もいます。たとえば、顕微鏡写真上の炭素端や高コントラストの人工物はしばしば粒子と誤認されます。【Display:】ボタンからポップアップウィンドウを使うことで、それぞれの顕微鏡写真においてこれを実行できます。マウスの中ボタンを使って粒子を取り除いて下さい。'''中ボタン'''を押しっぱなしにすることで、広い範囲の誤認粒子を複数取り除けます。忘れずに'''右ボタン'''メニューから新しい座標を保存して下さい。 | |
| − | + | [[ファイル:relion-autopicking-manualEdit.png|500px]] | |
| − | + | ひとたび全部の結果を満たしたすと、ディスクスペースを確保するために、最初のステップで書き出したFOMマップを削除したくなるかもしれません。手軽に行うために【Job actions】ボタンから、Gentle clean オプションを使うことができます。 | |
| − | + | ひとたび座標全体セットを満たしたら、以前のデータを全て保ちつつ、【Partcle extraction】jobを再実行する必要がありますが、【Input coordinates】を自動検出で新しく生成されたものに変えて下さい。これは下記のよりよい精密化のために使われる初期単粒子データセットを作成します。おそらく、allmics_autopicked のようなエイリアスが有意義です。 | |
| − | + | ||
| − | + | ||
| − | + | ||
====3.8.1 The shrink parameter(縮小値)==== | ====3.8.1 The shrink parameter(縮小値)==== | ||
| − | + | より速い処理を可能にするために、RELIOM-2.0では、コマンドライン引数 --shrink を通して顕微鏡写真をフィルタリングするオプションを実装しています。最もシンプルな使い方は --shrink 0 と使うことです。しかし、これはもっと制御して使うことができます。その場合は、次の方法に従って動作して下さい。 | |
| − | -- | + | --shrink の初期値は1.0ですが、これは効果がなく、顕微鏡写真に全くフィルターをかけません。これは以前のバージョンのRELIONの自動検出と同一の動作をします |
| − | + | ● --shrink val = 0 では、テンプレートリファレンスのローパスと同じように、--lowpass 指定されたローパスフィルタをかけた顕微鏡写真となります。これは単粒子解析(らせん状検出のためではなく)のために使うことをお勧めします。 | |
| − | + | ||
| − | + | ● --shrink 0 < val ≤ 1 では、画像サイズが、size= val ·micrograph size にした結果になります。すなわち、valとは顕微鏡写真のオリジナルサイズに適用させたスケールファクターです。 | |
| − | + | ||
| − | + | ● --shrink val > 1 では、size = val − (val mod 2)、すなわち、valより小さい最小の偶数の整数値になります。これは顕微鏡写真に特定の(偶数の)整数サイズ与える方法です。 | |
| − | + | ||
| − | + | 新しいサイズが--lowpassよりも小さい場合、これは要求された(あるいは初期設定の)分解能を超えて、分解能を制限することになるので、致命的ではありませんが警告をならす結果となります。RELIONの自動検出は多くのFFTを実行するため、顕微鏡写真のサイズもFFT計算の大きな落とし穴を避けるために自動的に調整されるようになりました。これらに関する注釈とどのようにパラメータが上書きされるかは、ユーザーが実行する各自動検出の最初の出力に表示されます。 | |
=== 3.9 Particle sorting(粒子ソート) === | === 3.9 Particle sorting(粒子ソート) === | ||
| + | RELIONには、各粒子とそれらの調節されたCTF補正リファレンスとの間の差異に基づいて粒子を素早く並べ替える機能もあります。ソートプログラムは【Particle sorting】jobタイプからアクセスすることができ、それぞれの粒子でリファレンスが利用できるときであれば、いつでも動かせます。たとえば、【Auto-picking】、【2D classification】、【3D classificationo】、【3D auto-refaine】の後などです。その使用方法を示すために、自動検出の後に実行します。 | ||
| − | + | 【I/O】タブでは以下のように設定してください。 | |
| − | + | ||
| − | + | ||
● Input particles to be sorted: Extract/allmics autopicked/particles.star | ● Input particles to be sorted: Extract/allmics autopicked/particles.star | ||
| − | ( | + | (このファイルは自動検出後の抽出プログラムによって生成されます。【2D classification】、【3D classification】または、【3D auto-refine】jobの場合は、最後の反復での_data.starを入力するべきです。) |
● Are these from an Extract job? Yes | ● Are these from an Extract job? Yes | ||
| − | (入力粒子が | + | (入力粒子が 【2D classification】、【3D classification】、【3D auto-refine】job由来の場合、これをNoにセットしてください。) |
● Autopicking references: Select/templates4autopick/class averages.star | ● Autopicking references: Select/templates4autopick/class averages.star | ||
| − | ( | + | (抽出された粒子のための座標を与える自動検出jobに使われたリファレンスを入力してください。) |
| − | + | 【CTF】タブ上では以下のように設定してください。 | |
● Are References CTF corrected? Yes | ● Are References CTF corrected? Yes | ||
● Ignore CTFs until first peak? No | ● Ignore CTFs until first peak? No | ||
| − | + | このプログラムはとても速く(分単位で)動作します。複数のMPI processorsを動かす必要がないほどです。おそらくエイリアスとしてafter_sortingを使えると思います。このjobは出力ディレクトリに particles_sort.star と呼ばれる入力ファイルと同じファイルを生成しますが、rlnPartileSelectZScore と呼ばれるカラムの追加があります。【Subset selection】jobタイプの 【OR select from particles.star】 とある行で、このファイルを選択することが出来ます。ポップアップ表示ウィンドウ内にて、その新しいカラムに基づいて粒子を可視化できます。ディスプレイの上に良い粒子、下に良くない粒子をもつ強い傾向が見えてきます。下から上にスクロールして、下のすべての粒子を破棄する必要があるポイントを決定でき、右マウスメニューを使用して Select all above および、または1つずつ粒子を選択することができます。 | |
| − | + | ||
| + | 右クリックメニューを使って、出力STARファイルに忘れずに保存しましょう | ||
== 4. Reference-free 2D class averaging (参照無2次元クラス平均) == | == 4. Reference-free 2D class averaging (参照無2次元クラス平均) == | ||
| − | + | '''前書き''' | |
| + | |||
良くない粒子を取り除くために、大抵は参照なし2Dクラス平均を使います。 | 良くない粒子を取り除くために、大抵は参照なし2Dクラス平均を使います。 | ||
| 行437: | 行736: | ||
=== 4.1 Running the jobs (動作の実行)=== | === 4.1 Running the jobs (動作の実行)=== | ||
| − | + | このチュートリアルでは動作タイプを【2D classificiation(2次元分類)】にセットし、その中の【I/O】タブは次の通りにセットします。その他のオプションは、前述の自動検出のテンプレートを作成した時と同じです。 | |
Input images STAR file: Select/after_sorting/particle.star | Input images STAR file: Select/after_sorting/particle.star | ||
現在約10,000個の粒子が存在するので、もっと多くのクラスを利用することができます。従って、【Optimisation(最適化)】タブを次の通りにセットします。 | 現在約10,000個の粒子が存在するので、もっと多くのクラスを利用することができます。従って、【Optimisation(最適化)】タブを次の通りにセットします。 | ||
Number of class: 100 | Number of class: 100 | ||
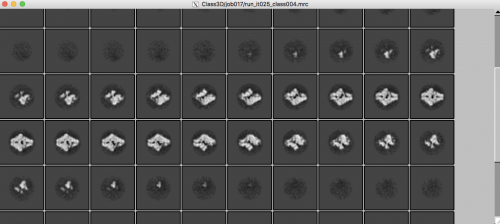
| − | + | この動作は以前に述べた動作のものよりはるかに負担がかかる(アルゴリズムは粒子の数とクラスの数に比例してスケーリングされます)ので、この動作を並列処理することをお勧めします。1つもしくは複数のGPUがある場合、この動作を1時間半分程度で実行できます。aftersortのようなエイリアスを使用できます。 | |
| − | + | B動作が終了したら、_model.staファイルを選択して【Subset select】を起動することができます。class2d_aftersortのようなエイリアスは意味があります。ここで、すべての見栄えの良いクラスをクリックして選択します。(もしくは右クリックメニューオプション【Select all classes above】を使用する。)この時、自動検出において低いしきい値を使用して、高解像度のノイズが蓄積している場合、低解像度のゴーストのように見える【Einstein-from-noise】クラスに気を付けてください。こうしたクラスの選択は避けて下さい。より良いクラスがすべて選択されたら、'''マウス右クリック'''メニューオプションから選択したものを保存します。 | |
| − | この手順は、複数回繰り返すことがあるので、覚えておきましょう。また、2D | + | この手順は、複数回繰り返すことがあるので、覚えておきましょう。また、2D classficationのあとのデータに残っている外れ画像を識別するために、ソート(並べ替え)アルゴリズムを再実行できることに注意してください。 |
| + | |||
| + | ※ 【2D classification】、【3D classifiction】は共に、最初にランダムにクラス分けをするため、外れ画像が多く混じっているとうまく分類できない可能性があります。そのため、粒子を選び直して、改めて分類するとよりよい分類が可能になります。 | ||
| + | |||
| + | |||
| + | 【Number of class: 100】とした場合の結果 | ||
| + | [[ファイル:RELION-2DRefine.png|500px]] | ||
=== 4.2 Analysing the results in more detail(より詳細な結果の分析) === | === 4.2 Analysing the results in more detail(より詳細な結果の分析) === | ||
| 行457: | 行762: | ||
● Class2D/allparts_run1/run_it025_classes.mrcs | ● Class2D/allparts_run1/run_it025_classes.mrcs | ||
| − | + | これは、クラス平均結果をMRCスタックとして保存しています。これらはメインGUIのDisplay: ボタンから _model.star を選択したときに表示される画像です。RELION は GUIから選択した場合、完全なCTF補正がかかることに注意して下さい。そのためクラス平均はおそらく黒い背景に対して白いものになります。もしデータが良ければ、その画像はローパスフィルターをかけた原子モデルにとてもそっくりな投影になります。 | |
| − | 2D | + | 2D クラス平均の質は3Dマップがうまくいくかどうかの指標になります。我々はタンパク質ドメインの投影を用いた内部構造と、理想的には平らになっているであろう粒子周りの溶媒領域を期待したいところです。 |
| − | + | ||
| + | 溶媒領域の放射状に伸びた筋は典型的な過剰適合の兆候です。もしこれがおきたときは、2D classification アルゴリズムのE-step内で分解能に制限をかけてみて下さい。 | ||
●Class2D/allparts_run1/run_it025_model.star | ●Class2D/allparts_run1/run_it025_model.star | ||
| − | + | これは実際のクラス平均に沿って精密されたモデルパラメーター(クラス間の画像の分布、再構成された構造中のシグナル対ノイズ比の球面平均、全てのグループのノイズスペクトラム、など)を含んでいます。 | |
| − | + | less コマンドを使って、このファイルの中身を見てください。特に、'''data_model_classes'''テーブルのそれぞれのクラスの粒子分布を確認して下さい。これとクラス平均を比較すると、低分解能のクラスにある粒子はほとんどない一方で、高分解能のクラスに存在する粒子は多いと分かる。 | |
| − | + | 平均的に少ない粒子を通しての平均化は自然と低いシグナル対ノイズ比になりますので、このときにベイズ推論的な考えを持つことが重要になります。それぞれのクラスにおいて概算されたスペクトラル、シグナル対ノイズ比は'''data_model_class_N''' テーブルに保存されます。同様に、それぞれのグループにおいて概算されたノイズスペクトラムは'''data_model_group_N'''テーブルに保存されます。'''data_model_groups'''テーブルはそれぞれのグループにて精密されたscale-factor強度を保存します。(平均よりも高い値をもつグループはより強力なシグナルを持っている、平均よりも低い値をもつグループは相対的に低いシグナルである) | |
| − | + | これらの値はデフォーカスと相互関係があることが多いだけでなく、汚れのの蓄積や氷の厚さに影響される。 | |
●Class2D/allparts_run1/run_it025_data.star | ●Class2D/allparts_run1/run_it025_data.star | ||
| − | + | これはそれぞれの粒子に関わる全てのメタデータを含んでいます。そのメタデータとは、particles.star fileに入力した情報の他に、最適な方向、最適なクラス同定、対数尤度への寄与、などに関する追加情報です。 | |
このファイルは再度新しく洗練されたデータをstarファイル同様の形式のでの入力に使用される。 | このファイルは再度新しく洗練されたデータをstarファイル同様の形式のでの入力に使用される。 | ||
| 行480: | 行786: | ||
●Class2D/allparts_run1/run_it025_optimiser.star | ●Class2D/allparts_run1/run_it025_optimiser.star | ||
| − | + | これは終了しなかった場合の再起動に必要な精密化過程についての全体的な情報が含まれています。たとえば、もし25反復後でもまだ収束しなかった過程を考える場合(評価のために反復24と25から出したクラス平均を比較できる)、Finished jobs パネルの中で以下の機能を選択できる。I/O タブ上で、'''Continue from here'''から上記のファイルを選択することが出来る。そのときに、Optimisationタブ上で | |
| + | Number of iterations:40 | ||
| + | と設定して下さい。また、Sampling タブで緻密な角度や並列処理をするサンプリング比率を選ぶかもしれません。optimiser.starファイルのもう1つの有用な特徴として、1行目に、これを動かすために与えられた正確なコマンドライン引数がコメントとして含まれています。 | ||
●Class2D/allparts_run1/run_it025_sampling.star | ●Class2D/allparts_run1/run_it025_sampling.star | ||
| − | + | これはサンプリング間隔の採用についての情報が含まれています。このファイルもまた再起動に必要なファイルです。 | |
=== 4.3 Making groups === | === 4.3 Making groups === | ||
| 行491: | 行799: | ||
''お急ぎの方はこのセクションをスキップしても構いません。'' | ''お急ぎの方はこのセクションをスキップしても構いません。'' | ||
| − | + | RELIONは、粒子をグループ化する際に、以下のふたつのことを実行します。ひとつは、平均のノイズのスペクトラム強度(σij^2)の推定と単一数としての強度スケールファクター(τi^2)の推定です。後者は、そのデータの異なる部分の間での、全領域シグナル対ノイズ比(SNRi)の違いを表現しており、例えば、氷の厚さ、デフォーカスまたは汚染等によるものです。 | |
| − | + | 通常、顕微鏡写真上の全ての粒子は別々のグループとして扱っています。この動作は顕微鏡写真ごとに多くの粒子が存在する場合には良いです。しかし、高倍率で扱っている場合、サンプルは非常に希釈されるか、最終的に選択されたものは顕微鏡写真ごとには少数しか粒子が含まれておらず、強度スケールファクター(またノイズスペクトラ)強度の推定は不安定なものとなります。一般的に各グループに少なくとも10〜20の粒子がある方が良いとしていますが、2D,3Dクラス化後、各グループの粒子の初期数はより少なくなるだろうということに注意しておいて下さい。 | |
| − | + | 顕微鏡写真ごとに粒子が殆ど無い場合、複数の顕微鏡写真からの粒子をグループ化することをお薦めします。このことから、RELION 2.0での新しいGUIとして【'''Subset selection'''】の中に便利な機能が追加されています。それは【I/O】タブ上で _model.starファイルを選択した時、【Regroup particle?】と表示された時【Yes】と入力し、【'''Approximate nr of groups : 5'''】と【Class option】タブで入力することで、すべての粒子を5つのグループに分類できます。(実際の数は入力値によって幾分変化するため、入力上での近似値となる)この方法は以前の最終バージョンのRELIONでは避けられた方法でした。このチュートリアルの顕微鏡写真には十分な粒子が含まれているので、ここではこの手順を使用しません。 | |
| − | + | RELIONは、他のプログラムで時々使用されるデフォーカスグループとは非常に異なることに注意しておいて下さい。RELIONは常に各粒子に使用されたグループとは無関係に(異方性の)CTF補正がされます。 | |
== 5. Unsupervised 3D classification (教師無3次元クラス分類) == | == 5. Unsupervised 3D classification (教師無3次元クラス分類) == | ||
| − | + | '''前書き''' | |
| − | + | ||
| − | + | 全てのデータセットは異成分からなっています。問題は どのくらい寛容でいられるかです。RELIONの3Dマルチリファレンス精密化手順は、強力な教師無し3次元分類へのアプローチを提供しています。しかし、まずは低解像度の単一の合意が得られるモデルが必要です。初期の結晶構造(PDB-ID 3I3E)から生成された低分解能のベータガラクトシダーゼモデルを使用します。これはすでに projectディレクトリの中に 3i3e_lp50A.mrcという名前で入っています。典型的なプロジェクトでは、良い初期モデルがないかもしれません。RELIONは、まだ初期モデル生成を行わないので、別のプログラムで生成する必要があります。 | |
| − | + | ||
| + | ※例えば、chimeraで、PDBを読みだ後、コマンドラインを立ち上げて、50Aのマップをつくるなら | ||
| + | molmap #0 50 | ||
| + | |||
| + | ※例えば、Eosでは、[[pdb2mrc]]を利用して作成する事ができます。 | ||
| + | $ pdb2mrc -i pdb -o mrc .... | ||
| + | |||
| + | すでにリファレンスモデルを正しいピクセルとボックスサイズで作っています。もしこの事例に当てはまらないモデルを使う場合は、コマンドラインからrelion_image_handlerというプログラムを使って、ピクセルサイズ(--anpixelと--rescale_angpixというオプションを使用)とボックスサイズ(--new_box)を変更してもかまいません。 | ||
| + | この場合、まず、【Import】jobタイプを使用し、直接プロジェクトに初期モデルをインポート出来ます。ファイルを入力したら、3i3a_lp50A.mrc を選択して、the Node typeで3D referenceを選択して下さい。同じ 【Import】jobを区別するために、initial-3drefのようなエイリアスを使います。 | ||
| − | + | === 5.1 Running the job (ジョブを開始) === | |
| − | === 5.1 Running the job=== | + | |
| − | + | 教師無し3次元分類は、【3D classification】jobタイプから実行可能です。 | |
| − | 【I / O】タブの設定: | + | 【I/O】タブの設定: |
| − | + | ● Input images STAR file: Select/class2d_aftersort/particles.star | |
| + | (選択された良い粒子が記述されたstarファイルを設定します。) | ||
| − | + | ● Reference map: Import/initial_3dref/3i3e_lp50A.mrc | |
| + | (比較するための参照構造を設定します) | ||
| − | + | ● Reference mask (optional): | |
| − | + | (これは空白のままにしておきます。これは、例えば、注目しているリボソーム精密化のために大/小サブユニットマスクを提供するところです。この項目を空のままにしておくと、[Optimisation]タブで指定された粒径の球形マスクが使用されます。 これは分類へのバイアスを最小限にする働きがあります。) | |
| − | + | ||
【Reference】タブの設定: | 【Reference】タブの設定: | ||
| − | + | ● Ref. map is on absolute greyscale: No | |
| − | + | (RELION内の同じデータから再構築されていないマップは、おそらく、正しいグレースケール上にないと考慮しておきましょう。) | |
| − | + | ● Initial low-pass filter (A): 50 | |
| − | + | (精密化プロセスにバイアスが導入する可能性があるため、高解像度の初期参照モデルは使用してはいけません。[[#ref9|[9]]]で説明したように、初期マップをできる限りフィルタリングする必要があります。リボソームでは大抵、70Åを使用し、小さい粒子では通常40〜60Åの値を使用します。) | |
| − | + | ● Symmetry: C1 | |
| − | + | (このサンプルはD2の対称性があることはわかっていますが、対称性を持たない初期分類を実行することをお勧めします。良くない粒子は適切なものから分離することができ、対称性は再構成されたマップで確認することができます。) | |
【CTF】タブの設定: | 【CTF】タブの設定: | ||
| − | + | ● Do CTF corection? Yes | |
| − | + | ● Has reference been CTF-correced? Yes | |
| + | (このモデルはPDBファイルで作成されているため、振幅はCTFの影響を受けていません。これは"CTF修正済み"とみなすことができます。ほとんどの他のEMパッケージから作られたモデルは、適切な低周波振幅補正を実行するパッケージが多くないため、ほとんどの場合CTF補正されていないことが多いです。(いくつかは補正されているかもしれません。その場合、モデルを通してスライスをチェックすることができます。もし黒の背景に対して白く見えるならば、モデルはおそらく適切にCTF修正されたものでしょう。一方で、黒いハローを伴って灰色に見えるのであればそのモデルはおそらく正しくCTF補正されていないでしょう。)これまでRELIONでCTF補正を行ったモデルは、適切に補正されています。) | ||
| − | + | ● Have data been phase flipped? No | |
| − | + | (位相補正だけを実施しているかどうかです。) | |
| − | + | ||
| − | + | ||
| + | ● Ignore CTFs untill first peak? No | ||
| + | (リファレンスを作成するために使用した[2D classification]jobでもこのオプションを使用した場合にのみ、このオプションを使用します。) | ||
【Optimisation】タブの設定: | 【Optimisation】タブの設定: | ||
| − | + | ● Number of class: 4 | |
| − | + | (より多くのクラスを使用すると、多様な情報が含まれているデータセットがより多くのサブセットに分割されます。計算コストは、CPU時間と必要なコンピュータメモリの両方の観点から、クラス数に比例して変化します。) | |
| − | + | ● Number of iterations: 25 | |
| − | + | (これは一般的には変更する必要はありません。) | |
| − | + | ● Regularisation parameter T: 4 | |
| − | Tの定義については[10] | + | (Tの定義については[[#ref10|[10]]]を参照してください。クライオEMの、2次元分類では、通常、Tは1~2を設定し、3次元分類では2~4を設定します。ネガティブ染色の場合、少し低い値が効果的な時があります。クラス平均にノイズが見える場合はTの値を低くし、逆にクラス平均の分解能が低すぎる場合はTの値を高くします。主に気をつけなければならない点は、高解像度のノイズの過学習です。あらかじめ計算された結果に2という値を使用しました。おそらく4を設定しても同じようにうまくいくでしょう。) |
| − | + | ※ Tの値は、tiと呼ばれるシグナルが大きいと値が大きくなる値を定数倍して使うことになります。つまり、SNRを過大評価するようになり、SRN低い領域まで利用する様になります。実際には、フーリエ空間のボクセル毎に決まりますが、大抵の場合、高分解能の情報まで用いる事を示しています。) | |
| − | + | ||
| − | + | ● Mask diameter (A): 200 | |
| + | (前回の[2D classification]のjobタイプと同じ値を使用してください。) | ||
| − | + | ● Mask individual particles with zeros? Yes | |
| − | + | (Yes: 0 / No: 乱数) | |
| − | + | ● Limit resolution E-step to (A): -1 | |
| + | (正の値を指定すると、この値を超える周波数はアラインメントに含まれません。これは過学習を防ぐのにも役立ちます。実際に必要にではありませんが、10~15Aに設定されている可能性があります。) | ||
| − | + | 【Sampling】タブでは、通常何かを変更する必要はありません(正20面体ウイルスのような大型で高い対称性をもつ粒子の場合のみ、典型的には、この時点で3.7度の角度サンプリングを使用します)。 | |
| − | + | 【Helix】タブは無視して下さい。 | |
| − | + | 以前の[2D-classification]の場合と同様に【Compute】タブに記入してください。 | |
| − | === 5.2 Analysing the results in more detail === | + | ここでも、【Running】タブで、使用するNumber of MPI processors(MPIプロセッサの数)とthreads(スレッド数)を指定できます。[2D classification]のjobタイプで説明したように、3次元分類は2次元分類よりも多くのメモリを必要とするため、より多くのスレッドが使用されることがあります。しかし、この場合、画像はかなり小さく、RAM不足はそれほど大きな問題ではないかもしれません。おそらく、最初の3次元分類を表示するためにfirst_exhaustiveのようなエイリアスを使用でき、徹底的な角度検索を使用しているのではないでしょうか。 |
| + | |||
| + | 精密化された再構成を分析する際には、UCSFキメラなどのしきい値マップだけでなく、スライスでそれらを見ることも非常に便利です。スライスビューでは、未解決の異質性のより良い印象を受けるでしょう。これは、スライスに不鮮明な領域や縞状の領域として表示されます。スライスはまた、溶媒領域の平坦性の良好な印象を確認できます。【Display:】ボタンを使用して、最後の反復からの再構成のいずれかを選択して、RELIONのスライスビューを開きます。 | ||
| + | |||
| + | UCSFキメラなどを使用し、表示されたマップを3Dで見ると、マップが精密化の間にわずかに回転する可能性があるので、それらをすべて最良のものに合わせることはしばしば良い考えです。キメラでは、【Tools】 - > 【Volume Data】 - > 【Fit in Map】ツールを使用します。お互いに並んでいる複数のマップを見るには、【Tools】 - > 【Structure Comparison】 - >【Title Structures】ツールと、【Viewing】ウィンドウの【independent】中心回転方法を組み合わせたものが好ましいです。 | ||
| + | |||
| + | 2次元分類の場合と同様に、【Subset selection】を使用して、1つ以上のクラスに割り当てられた粒子のサブセットを選択することができます。【I/O】タブで、'''_model.star'''ファイルのサブセットを最後の反復から選択します。表示ウィンドウには、4つの精密化されたモデルの中央スライスが表示されます。最適なクラスを選択し、右クリックメニューを使用して対応する粒子を保存します。class3d_first_exhaustiveのようなエイリアスを使用してください。 | ||
| + | |||
| + | [[ファイル:RELION-3D-FInest.png|500px]] | ||
| + | |||
| + | 4つのクラスの内の1つ | ||
| + | |||
| + | === 5.2 Analysing the results in more detail (詳細な結果の解析) === | ||
''ここではRELIONへの興味が深い人向けの、より詳細な情報を含んでいます。'' | ''ここではRELIONへの興味が深い人向けの、より詳細な情報を含んでいます。'' | ||
''お急ぎの方はこのセクションをスキップしても構いません。'' | ''お急ぎの方はこのセクションをスキップしても構いません。'' | ||
| − | + | 出力ファイルは基本的に2次元分類の実行時と同じです。(実際に2次元,3次元精密時に同じ記法を利用しています)唯一の違いは、以前に出力された2次元クラス平均に関するシングルMRCスタックとは対照的に、各クラスのマップが別々のMRCマップとして保存されることです。例えば、'''run_it025_class00?.mrc'''というMRCマップです。 | |
| − | + | 今まで通り、小さいクラスは大きなクラスより精密にローパスフィルターにかけられ、スペクトルの'''シグナル対ノイズ比(SNR)'''が_model.starファイルのdata_model_class_Nテーブル(N = 1,,,,K)に保存されます。おそらく今がSTARファイルから任意のタイプのデータを抽出するのに便利な2つの手軽なスクリプトを導入するいい機会です。以下の通りに入力して下さい。 | |
relion_star_printtable Class3D/first_exhaustive/run_it025_model.star data_model_class_1 rlnResolution rlnSsnrMap | relion_star_printtable Class3D/first_exhaustive/run_it025_model.star data_model_class_1 rlnResolution rlnSsnrMap | ||
| − | + | 上記はdata_model_class_1テーブルから解像度に関する(rlnResolution)コラムとスペクトルのシグナル対ノイズ比に関する(rlnSsnrMap)コラムを画面に出力します。ここでそのファイルを参照して、お好みの言語でプロットできます。もし、使用者のコンピューターにgnplotがインストールされているのであれば、以下の通りに入力して下さい。 | |
relion_star_plottable Class3D/first_exhaustive/run_it025_model.star data_model_class_1 rlnResolution rlnSsnrMap | relion_star_plottable Class3D/first_exhaustive/run_it025_model.star data_model_class_1 rlnResolution rlnSsnrMap | ||
| 行591: | 行921: | ||
grep _rlnChangesOptimalClasses Class3D/first_exhaustive/run_it???_optimiser.star | grep _rlnChangesOptimalClasses Class3D/first_exhaustive/run_it???_optimiser.star | ||
| − | + | これまででお分かりのように、STARファイルはさまざまなタイプの入出力データを扱うのに非常に便利なツールです。得られた結果の分析するための柔軟で強力なツールとなるrelion_star_printtableのようなスクリプトは、だいたいgrepやawkのようなlinuxのシェルコマンドと併用して実行できます。 | |
| − | == 6 High-resolution | + | == 6 High-resolution 3D refinement(三次元精密化) == |
| − | + | '''前書き''' | |
| − | + | ||
| + | 十分に均質なサブセットは一旦選択されると、このサブセットを自動的に高い解像度に精密化するために[3D auto-refine]を使用することができます。この手順は分解能を推定するために、gold-standard Fourier Shell Correlation(FSC)(二つの群に分け、互いに独立して再構成した場合に、どの分解能まで相関があるかにより判断)と言われる判断基準を用います。これにより、過学習を避けることができます[[#ref13|[13]]]。角度の割当の正確さを推定する新しい手順([[#ref11|[11]]])と組み合わせ、精密化が収束するときに自動的に決定されます。そのため、この手順は使用者の入力をほとんど必要としません。すなわち、客観性をのこしたまま、多くのデータセットのための優れたマップが生成がされていると述べられています。もう一つの長所は、一般的にこの手順は必要な実行回数が一回のみであり、最適化のための引数はほとんどありません。 | ||
=== 6.1 Running the job (動作の実行)=== | === 6.1 Running the job (動作の実行)=== | ||
| − | + | RELIONで、【3D auto-refine】jobタイプを選択してください。RELIONでの入力引数の大部分は【3D classification】jobタイプと同様のものが残っていますが、一部は利用できません。【Sampling】タブでの配向性のサンプリング間隔は反復の最初の数回のみ使用されており、ここでのアルゴリズムは収束するまで、角度サンプリング間隔が自動的に細かくなっていきます。したがって、8面体または正20面体対称よりも小さい角度で、全ての精密化仮定でで、一般的に大域探索では、初期値の角度である7.5度のサンプルを使用し、局所探索では1.8度のサンプルから行います。より正確な対称の精密化を行う場合のみ、3.7度でのサンプリングを行い、局所探索では0.9度ごとにサンプリングを行います。 | |
| − | RELION-2. | + | RELION-2.0は、【Optimisation】タブでUse solvent-flattend FSCsと呼ばれる新しいオプションがあります。このオプションの設定はRELION-1.4のようにNoで実行します。もしYesで設定した場合、自動精密化は、[Post-processing]jobタイプで使用するものと似ている代表的なFSC曲線で溶媒平滑化補正が使われます。その結果、この動作では【I/O】タブでマスクの提供も必要です。タンパク質が粒子ボックスの中に比較的小さい体積を占有している時、このオプションは非常に役に立ちます。他にも、伸長された分子や、マスクを使用して小さい部分の精密化に注目した時などに役立ちます。このような場合、マスクが使用されていない代表的なFSCは、精密化の分解能が低く見積もられます。今回の精密化では、このオプションはNoのままにしてください。(マスクを提供していないからです) |
| − | 入力STARファイルとして、Select/class3d_first_exhaustive/particles. | + | 入力STARファイルとして、Select/class3d_first_exhaustive/particles.starを選択し、【3D classification】実行(例えば、Class3D/first_exhaustive/run_it025_class004.mrc)から初期モデルとして、精密化されたマップのひとつを使用します。あるいは、以前に使用したものと同じ、3i3e_lp50A.mrcモデルを使用します。どちらでもおそらく違いはほとんどありません。さらに、マップ内の高周波成分へ偏るのを防ぐためと、初期マップよりも高い解像度で完全に独立した精密な”gold-standard”を保持するために、初期マップにローパスフィルターを'''強くかける'''ことが最も良いです。再び50Åを使いましょう。分子が対称に見えるときは、精密化したものに2D symmentryをかけましょう。 |
| − | + | MPIのノードは1つのマスター(他のスレーブを管理するだけのもの)と2つのスレーブ(2つの半分のセットに作用するもの)のセットで分かれているので、奇数のMPI処理装置を使用するのが最も効率が良いです。ナイキスト周波数までのすべての周波数が考慮されているため、必要メモリは最終反復でかなり増加します。従って、使っているクラスターノードのコアと同じ数のスレッドで動かしたいデータセットのボックスより大きいサイズで作られたボックスが必要です。もしかするとafter_first_class3dのようなエイリアスが良いかもしれません。 | |
=== 6.2 Analysing the results (結果の分析)=== | === 6.2 Analysing the results (結果の分析)=== | ||
| − | + | 出力ファイルは3D classificationを実行したときのものと大部分が同じです。しかし、全てにおけるプログラムの反復では、2つのrun_it0??_half?_model.starと2つのrun_it0??_half?_class001.mrcファイルを書き出します。それぞれデータの半分が精密化されます。収束した場合にのみ、ひとつのrun_model.starとrun_class001.mrcファイルを書き出します(名前に_it0??がありません)。最終反復では、2つの独立した半分の復元データが一緒に結合されているので、一般的に最終反復で解像度がかなり改良されます。プログラムはすべてのデータでナイキスト周波数を使用しているので、この反復では更に多くのメモリとCPUが必要です。 | |
| − | + | 角度サンプリングでの自動増加が自動精密化手順の様子が重要な側面であることに注意してください。[[#ref11|[11]]]で説明されているシグナル対ノイズの考察が基礎となっており、角度の精度と併進運動の割り当てを評価します。プログラムは必要だと判断したものよりも細かい角度や並進運動サンプリング比は使用しません(結果がよくならないためです)。評価された精度と採用したサンプリング比は現在の解像度評価と一緒にすべて_optimiser.starと_model.starファイルに保存されますが、stdoutファイルからも抽出されるかもしれません。 | |
| − | + | 例えば、以下のように試してみてください。 | |
| + | |||
| + | grep Auto Refine3D/after_first_class3d/run.out | ||
== 7 Mask creation & Postprocessing(マスク作成&後処理) == | == 7 Mask creation & Postprocessing(マスク作成&後処理) == | ||
| − | + | '''前書き''' | |
| − | + | ||
| + | 3D自動精密化の実行後、マップを先鋭化する必要があります。その場合も、自動精密化の内部で生み出される'''gold standard FSC'''カーブはマスク化されていないマップのみ使います(Use solvent-flattened FSCsオプションを使っていない場合に限ります)。これは実際の精密化中には、実際の分解能は下回っている(under-estimated)ということを意味しています。なぜなら、溶媒領域のノイズがFSCカーブを低くするからです。RELIONにおけるBファクターでの先鋭化と、マスク化したFSCカーブ[[#ref2|[2]]]を計算するための手順は、"post-processing(後処理)"と呼ばれています。しかし最初には、タンパク質の末端と溶媒領域の開始点を精密化するためのマスクを作る必要があります。これは【Mask Creation】ジョブタイプを使って行います。 | ||
| − | |||
=== 7.1 Making a mask (マスク作成)=== | === 7.1 Making a mask (マスク作成)=== | ||
| − | + | 【I/O】タブ上で3D自動精密化が終わった出力マップ、'''Refine3D/after_first_class3d/run_class001.mrc'''を選択してください。【Mask】タブ上で以下のように設定してください。 | |
● Lowpass filter map (A): 15 | ● Lowpass filter map (A): 15 | ||
| − | ( | + | (15Åのローパスフィルターが滑らかな溶媒マスクのために良い選択と思われます) |
● Pixel size (A): 3.54 | ● Pixel size (A): 3.54 | ||
| − | ( | + | (この値はローパスフィルターの計算にのみ使われます) |
● Initial binarisation threshold: 0.02 | ● Initial binarisation threshold: 0.02 | ||
| − | ( | + | (これは例としてタンパク質領域の外側にノイズスポットが完全になく表示するキメラのように、ローパスフィルターのかかったマップの描写でのしきい値となります。適切なスポットを見つけるために、しきい値を上げたり下げたりしてみて下さい。入力マップのローパスフィルターがかけられたバージョンを得るために --lowpass 15 --angpix 3.54オプションを付与させてrelion_image_handlerと呼ばれるコマンドラインプログラムを使用できることを覚えておいてください。初期しきい値としてよい値はだいたい、0.01から0.04のあたりです) |
● Extend binary map this many pixels: 2 | ● Extend binary map this many pixels: 2 | ||
| − | ( | + | (上記のしきい値は白黒画像マスクを生成するのに使われます。このマップ中のホワイト値は全方向の多くのピクセルで増加します。これを使用して、最初のバイナリマスクのサイズを小さくします。) |
● Add a soft-edge of this many pixels: 3 | ● Add a soft-edge of this many pixels: 3 | ||
| − | ( | + | (これはマスクに余弦形状のソフトエッジを配置します。これは、とても鋭いマスクにかなり繊細である可能性があるFSCカーブに、マスクが及ぼす影響を計測する補正手順として重要です。マスクの生成は相対的に速いので、しばしば分解能の最も適した概算を得るためのマスクパラメーターと共に使います。) |
| − | + | 【Helix】タブを無視して下さい。 | |
| − | + | 例えば、first3dref_th002_ext2_edg3のようなエイリアスを使って下さい。【'''Display:'''】ボタンを使うことで、マスクの結果を通した切片を見たり、UCSFキメラにマスクを読み込ませることができます。後者は、マスクが構造全体をカプセル化するが、マスク内に多くの溶媒を残さないことを確認するために、自動精密手順のマップと一緒に良いアイデアになるかもしれません。 | |
| − | == | + | === 7.2 Postprocessing (後処理)=== |
| − | + | [Post-processing]jobタイプを選択して、【I/O】タブで以下のように設定して下さい。 | |
| + | ● One of the 2 unfiltered half-maps: Refine3D/after first class3d/run half1 class001 unfil.mrc | ||
| + | ● Solvent mask: MaskCreate/first3dref th002 ext2 edg3/mask.mrc | ||
| + | ● Calibrated pixel size (A): 3.54 | ||
| − | + | (ときどき、正確なピクセルサイズと思って作り始めたモデルが、実は数%異なっていたということがわかることがあります。RELION内では、この時までになしたもの全ては、まだ一貫しています。そのためマップの再精密化や、データのクラスの再分類をする必要はありません。正確なマップと最終解像度の推定のために必要なことが、ここに正しいピクセルサイズを提供することです。) | |
| − | + | ||
| − | + | ||
| − | + | 【Sharpen】タブで以下のように設定してください。 | |
| − | + | ● MTF of the detector (STAR file): mtf falcon2 300kV.star | |
| + | このメーカーは、自身のディテクターのために、プロジェクトディレクトリの中に保存されている mtf_falcon_300kV_down4.starファイルと同じ形式で保存することになる適切なMTFカーブを提供します。RELION wikiに記録した解析結果から、2,3個の一般的なカーブをダウンロードすることもできます。 | ||
| + | ● Estimate B-factor automatically: Yes | ||
| + | この手順は本誌[[#ref8|[8]]]に基づき、最終解像度が確実に10Åを超えるように伸ばす必要があります。もしマップの解像度が10Åに満たない場合、独自の “ad-hoc” Bファクターを代わりに使う必要があるかもしれません。 | ||
| + | ● Lowest resolution for auto-B fit (A): 10 | ||
| + | これは通常変える必要はありません。 | ||
| + | ● Use your own B-factor? No | ||
| − | + | 【Filter】タブでは以下のように設定してください。 | |
| + | ● Skip FSC-weighting? No | ||
| + | このオプションはときに、全体のマップ分解能を超えるような解像度へと拡張するマップ範囲を解析するのに便利です。今回は当てはまらないので、Noにします。 | ||
| − | + | jobを実行(クラスターは必要ないほど、このjobはすぐ終わります)し、first3dref のようなエイリアスを使用して下さい。【Display】ボタンを使って、後処理されるマップと、この構造のFSCカーブとGuinierプロットを含んだPDFを通して、スライスを表示します。ChimeraでPostProcess/first3dref/postprocess.mrcマップを開くこともできます。そこでは、3D自動精密化の手順となる収束したマップでの全てのα-へリックスの場所を簡単に見ることが出来ます。分解能の推定は、以前に発表された[[#ref2|[2]]]のフェイズランダムの手順に基づいてます。フェイズランダムマップのFSC(赤いカーブ)が、多かれ少なかれ、推定していた後処理マップの解像度のゼロ点であることを確認してください。 | |
| − | + | そうでない場合は、マスクが鮮明すぎるか、細部の描写が多すぎます。この場合は。強いローパスフィルターや、以前の[Mask creation]ステップでの、より広く、より薄いマスクを使用し、後処理を繰り返して下さい。実際に少し薄いマスクを作りたくなるかもしれないので、その場合はエッジがだいたい5ピクセルになるように試してみてください。このために【Continue now】ボタンを使った場合、以前の結果が上書きされます。左側にあるjobタイプブラウザをクリックし、【Run now!】ボタンで新しい出力ディレクトリが出来ます。 | |
| − | + | ※マスクをきつく掛けるとマスクの影響による分解能を見ることになります。それを避けるためにマスク内が乱数であった場合(つまり、構造が無い場合)のFSCカーブと比較します。 | |
| − | + | == 8 Local-resolution estimate(ローカル解像度推定) == | |
| + | 後処理プログラム【Post-processing】からの推定される分解能は広域的な推定となります。しかしながら、たった一つの数では、高分子複合体の再構築でしばしば観測される分解能が場所毎で異なることを描写できません。Alp Kucukelbir and Hemant Tagareはマップ全体の解像度の変化を推定する効果的なプログラムを記述しています[[#ref4|[4]]]。RELION は、【Local resolution】 jobタイプを通してプログラムのラッパーを実装しています。【I/O】タブ上で以下のように設定してください。 | ||
| − | + | ● One of the two unfiltered half-maps: Refine3D/after first class3d/run half1 class001 unfil.mrc | |
| + | ● User-provided solvent mask: MaskCreate/first3dref th002 ext2 edg3/mask.mrc | ||
| + | ● Calibrated pixel size: 3.54 | ||
| − | + | (正しいピクセルサイズであると考えたモデルを構築し始めたときに、実際には数パーセントずれていたことがわかることがあります。RELION内では、この時点までの全てが一貫しています。そのため、再度、マップの精密化とデータの分類をする必要はありません。正確なマップと最終的な解像度推定のために正しいピクセルサイズをここで指定するだけで良いです。) | |
| + | 【ResMap】タブ上で以下のように設定してください。 | ||
| + | |||
| + | ● ResMap executable: /where/ever/it/is/resmap.exe | ||
| + | |||
| + | (RESMAP実行可能ファイルへのパスを指定します。シェル上で環境変数$RELION_RESMAP_EXECUTABLEを定義することで、どのディレクトリ上でもコマンドを打てることに注意して下さい。) | ||
| + | |||
| + | ● P-value: 0.05 | ||
| + | (これを変更することはほとんどありません) | ||
| + | |||
| + | ● Highest resolution (A): 7 | ||
| + | (ナイキスト周波数は7Åです) | ||
| + | |||
| + | ● Lowest resolution (A): 10 | ||
(計算上での最低の解像度を指定します) | (計算上での最低の解像度を指定します) | ||
| − | Resolution step size (A): 0.5 | + | ● Resolution step size (A): 0.5 |
| + | (ステップ数を増やせば、より正確なローカル解像度マップの結果が導き出せますが、更に実行時間は長くなります。) | ||
| − | + | first3refのようなエイリアスを使用して下さい。RASMAPプログラムからポップアップ画面上に表示されたら、緑色の線が幾分水平であるかどうかを確認してください。それを変更するためには、RESMAPのGUIからフィルターボタンを利用して下さい。しかしながら、RELION上で生成されたマップに変更を加える必要がないことにしばしば気がつくでしょう。RESMAPプログラムが完了するまでに時間がかかることがあります。 | |
| − | + | Resmap/first3dref/half1_resmap.mrcの結果は、UCSF chimera上でTools -> Volume data -> Surface colorと選択した後、volume data valueで選択したローカル解像度によってPostprocess/first3dref/postprocess.mrcのマップに色をつけ、rasmapファイルをブラウズ(閲覧)するために使われます。特別な場合では、データがダウンサンプリングされ、ベータガラクトシダーゼはあまり柔軟な粒子ではないために、局所分解能の程度は見るためにあまり興味深いものではありません。しかし、とにかくそのことを知るのはいいことです。 | |
== 9 Moving-processing & particle polishing == | == 9 Moving-processing & particle polishing == | ||
=== 9.1 Getting organized(準備) === | === 9.1 Getting organized(準備) === | ||
| − | 3. | + | [[#3.2_Beam-induced_motion_correction.28.E9.9B.BB.E5.AD.90.E7.B7.9A.E3.81.AB.E3.82.88.E3.82.8A.E8.AA.98.E8.B5.B7.E3.81.95.E3.82.8C.E3.82.8B.E7.94.BB.E5.83.8F.E3.81.AE.E5.8B.95.E3.81.8D.E3.81.AE.E8.A3.9C.E6.AD.A3.29|3.2]]で説明したように、RELIONパイプラインを通して、電子線に誘導されたモーションの補正を実行しているなら、全ての設定が既に終わっている状態です。RELONでの作業以外で、モーション補正を行った場合は、最初にインポートした顕微鏡写真と同じディレクトリ内(このチュートリアルではMicrographs)に全ての(補正に利用した)動画を置く必要があります。このディレクトリ内で、以前粒子を選択して平均化した顕微鏡写真と同じrootnameで動画が呼び出されているかを確認してください。例えば今回はrootnameが"movie"なので、mic001.mrcのムービーはmic001_movie.mrcsとして呼び出されます。[Import]jobを使って動画を取り込んでください。また、2D micrograph moviesオプションを選択してください。 |
| + | |||
| + | また後ほど、動画-粒子の標準化は動画となった元の顕微鏡写真ごとの平均だと仮定されています。これがあなた自身のデータではそうではない場合、全体の画像処理を開始する前に、relion_image_handlerプログラムを使用して、任意の数の動画フレームから顕微鏡写真の平均を計算することができます。 | ||
=== 9.2 Movie refinement(動画修正)=== | === 9.2 Movie refinement(動画修正)=== | ||
| − | + | 【Movie refinement】jobは、動画フレームから粒子を抽出し、それらを3次元モデルに対応させる、この二つの処理を組み合わせて行います。 | |
【I/O】タブの設定: | 【I/O】タブの設定: | ||
| − | • Input movies STAR file: | + | • Input movies STAR file: MotionCorr/job002/corrected_micrograph movies.star |
| − | MotionCorr/job002/ | + | |
• Rootname of movies files: movie | • Rootname of movies files: movie | ||
| − | + | (これらの動画のファイル名は_movie.mrcsで終わるためです。) | |
| + | |||
| + | • Continue 3D auto-refine from: Refine3D/after_first_class3d/run it020 optimiser.star | ||
| + | ([3D auto-refine]を実行後、含まれていた粒子のみ動画から抽出されます。これは、relion-1.4でのmovie-particle抽出とは大きく異なっています。 | ||
| − | |||
| − | |||
| − | |||
• Process micrographs in batches of: -1 | • Process micrographs in batches of: -1 | ||
| − | relion-2.0には、大きな動画データセットを処理する際に計算が不安定になることがあることが分かっているため、顕微鏡動画をより小さなバッチで処理するオプションがあります。このテストデータセットには15個のムービーのみが含まれており、バッチ処理は不要であるため、-1の値が使用されます。大きなデータセットの場合、50- | + | (relion-2.0には、大きな動画データセットを処理する際に計算が不安定になることがあることが分かっているため、顕微鏡動画をより小さなバッチで処理するオプションがあります。このテストデータセットには15個のムービーのみが含まれており、バッチ処理は不要であるため、-1の値が使用されます。大きなデータセットの場合、50-100のバッチで顕微鏡写真を処理するのが通例です。) |
| + | |||
(データセット:コンピューターで処理されるデータのまとまり。一般的に属性によって分類されたり、ある目的で集められたりしたデータが記録されたファイル群) | (データセット:コンピューターで処理されるデータのまとまり。一般的に属性によって分類されたり、ある目的で集められたりしたデータが記録されたファイル群) | ||
| + | |||
【extract】タブの設定: | 【extract】タブの設定: | ||
| 行696: | 行1,058: | ||
• Last movie frame to extract: 16 | • Last movie frame to extract: 16 | ||
• Average every so many frames: 1 | • Average every so many frames: 1 | ||
| − | + | (少なくとも平均化したフレームで、約1 electron/Ų線量を持っているためこの値を調節します。各ムービーフレームで0.5-1 electron/Ų線量未満であれば1よりも高い値を使用します。) | |
• Maximum number of MPI nodes: 8 | • Maximum number of MPI nodes: 8 | ||
| − | + | (このjobの第一ステップでは、動画粒子の抽出はI/Oディスク上で非常に集中します。 | |
| − | したがって、動作のこの部分で多数のMPI | + | したがって、動作のこの部分で多数のMPI processes使用するのは効率的ではない可能性があります。【Running】タブ上で、与えられたMPI processes数にかかわらず、ここでは与えられた数より多くのProcessesを使うことはないでしょう。我々のクラスタでは、8がいいでしょう。) |
| + | |||
• Particle box suze (pix): 100 | • Particle box suze (pix): 100 | ||
| − | + | (【Particle extraction】jobで以前使用したサイズと同じか確認してください。) | |
• Invert contrast? Yes | • Invert contrast? Yes | ||
| − | + | (【Particle extraction】jobの時と同じものを使用してください) | |
• Rescale particles? No | • Rescale particles? No | ||
| − | + | (【Particle extraction】jobの時と同じものを使用してください) | |
【normalise】タブの設定: | 【normalise】タブの設定: | ||
• Normalize movie-particles? Yes | • Normalize movie-particles? Yes | ||
• Diameter background circle (pix): 60 | • Diameter background circle (pix): 60 | ||
| − | + | (【Particle extraction】jobの時と同じものを使用してください) | |
• Stddev for white dust removal: -1 | • Stddev for white dust removal: -1 | ||
| 行720: | 行1,083: | ||
【refine】タブの設定: | 【refine】タブの設定: | ||
• Running average window: 7 | • Running average window: 7 | ||
| − | + | (最適値は、粒子のサイズや線量率などによって決まります。基本的に、この粒子をどの程度正確に累積線量に合わせることができるかということが主な問題となるでしょう。リボソームのような大きな分子では、実行するためには3-5 electron/Ų線量程あれば十分でしょう。それよりも小さい粒子、例えば、β-ガラクシダーゼなどでは、これを7-9 electrons/Ųまで増やしてもよい) | |
• Stddev on the translations (pix): 1 | • Stddev on the translations (pix): 1 | ||
| − | これにより、動画フレームが平均位置からどれだけ遠くに移動できるかが制限されます。 | + | (これにより、動画フレームが平均位置からどれだけ遠くに移動できるかが制限されます。) |
• Also include rotational searches? No | • Also include rotational searches? No | ||
| − | + | (回転を含めると計算上、コストがかかってしまいます。そして、粒子の研磨手順はビーム誘導の回転を無視してしまいます。ビーム誘起回転を含む(かつ、研磨段階をスキップできる)非常に大きなオブジェクトにのみ有用です。それでも、リボソームでさえ、以下に概説する研磨手順が好ましいです。) | |
| − | + | ||
| − | + | ||
| − | + | ||
| − | + | 上で述べたように、この動作では最初に動画粒子を抽出し、3次元レファレンスとの配置合わせを行います。最初の段階では、制限されたMPI processesで実行されますが、2番目のパートは更に多くのMPI processesで実行する必要があります。そしてこれを【Runnning】タブ上で選択しないといけません。必要なメモリは、3次元自動レファレンスのときとほぼ同じです。したがって、スレッド数に応じて調整してください。おそらく、first3drefのようなエイリアスを再度使うことができるのではないでしょうか? | |
| − | + | ||
| − | == 10 Wrapping up == | + | このjobから出力された唯一のファイルは、全ての動画粒子を精密化した動画フレーム粒子のSTARファイルとなります。これはMovieRefine / first3dref / run_avg7_off1_data.starという名前で呼び出されます。実行中の平均幅と変換の標準偏差の値が出力名に反映されるのは、これらのパラメータの値が異なる場合が多く、各設定に対してすべての動画粒子を再抽出しないようにするためです。 |
| − | + | 毎回、同じ動画抽出ステップを繰り返さないためにするためには、【Continue now】ボタンを使用して、同じ出力ディレクトリに対応するファイル名を持つ別のファイルを選択してください。 | |
| − | 以上です!このチュートリアルを楽しんで、それが役に立つことを願います。もし他にRELIONについて質問があるならば、最初にRELION | + | |
| − | + | === 9.3 Particle polishing (粒子の洗練(研磨)) === | |
| + | 粒子のpolishingアルゴリズムは、MovieRefine/first3dref/run_avg7_off1_data.star から(おそらく非常にノイズの多い)移動軌跡(movement tracks)を取得し、それを利用して直線になるように適合させます。これらのトラックのノイズを更に減らすために、顕微鏡写真上の隣接する粒子がしばしば同様の方向に移動するという観測も利用します。これは、複数の隣接する粒子のトラックを一緒に評価することによって行われる。ここで、各粒子のその隣接トラックへの寄与は、粒子間距離のガウス関数によって決定される。このガウス分布の(ピクセルの)標準偏差は、ユーザによって決まります。第2のステップでは、polishingアルゴリズムは、Bファクターおよび線形強度ファクタを推定して、個々のムービーフレームにおけるシグナルの解像度依存パワーを記述する。このように、分解能に依存する電子線ダメージは、各粒子のすべての位置合わせしたムービーフレームの加重平均を行うことによりモデル化することができます。得られる粒子は、シグナル対ノイズ比(SNR)が増加するので、"polished"または"shiny"だと言われます。 | ||
| + | |||
| + | [Particle polishing]jobタイプの【I/O】タブにて以下のようにして下さい | ||
| + | ●Input STAR file with aligned movies: MovieRefine/first3dref/run avg7 off1 data.star | ||
| + | |||
| + | ●Mask for the reconstruction: MaskCreate/first3dref th002 ext2 edg3/mask.mrc | ||
| + | (後処理で良い結果を出したものと同じものを使用してください。) | ||
| + | |||
| + | 【Movement】タブの設定: | ||
| + | ●Linear fit particle movements? Yes | ||
| + | |||
| + | ●Stddev on particle distance (pix): 300 | ||
| + | (粒子がより大きければ、ムービーの精密化からの移動軌跡の騒音は少なくなります。したがって、ここでは小さな値を使用できます。 リボソームについては、100ピクセルくらいをよく使用します。 値が小さいほど、複雑な動きのパターンが記述できます。) | ||
| + | |||
| + | 【Damage】タブの設定: | ||
| + | ●Perform B-factor weighting? Yes | ||
| + | |||
| + | ●Highres-limit per-frame maps (A): 6 | ||
| + | (このオプションは、単にコンピューターリソースを節約するためのものです。 個々のフレームの再構成は、ナイキストにまで拡張されないため、この値をここに指定して、シングルフレームの再構成を実行する際に考慮する周波数を制限することができます。) | ||
| + | |||
| + | ●Lowres-limit B-factor estimation (A): 20 | ||
| + | (このアルゴリズムは、個々のフレーム再構成と、一緒に平均化されたすべてのムービーフレームからの再構成との間の相対的なギニアプロットを推定します。 これらのプロットは直線にフィットするために使用されることがあり、[[#ref8|[8]]]で提案された元のギニアプロットよりも頻繁に低い周波数がこれらのプロットに含まれることがあります。) | ||
| + | |||
| + | ●Average frames B-factor estimation: 1 | ||
| + | (常に最初に1の値を使用するようにしてください。 Bファクターの形状を信頼して推定するために適切な高解像度を得られない再構成があれば、このオプションを使用して3、5ムービーフレーム7つを平均することができます。 奇数の値のみが許可されます。) | ||
| + | |||
| + | ●• Symmetry: D2 | ||
| + | |||
| + | 【normalise】タブの設定: | ||
| + | ●Diameter background circle (pix): 60 | ||
| + | |||
| + | (前と同じように、[Particle extraction]jobタイプで使用します。) | ||
| + | ●Stddev for white dust removal: -1 | ||
| + | |||
| + | ●Stddev for black dust removal: -1 | ||
| + | |||
| + | 【Helix】タブを無視し、first3drefのようなエイリアスを使用して、jobを実行します。 粒子の研磨はGPU加速されていないので、おそらくこれをクラスタ上で実行したいと考えるでしょう。このjobの最大使用MPIプロセッサ数は、データのムービーフレーム数の二倍です。粒子ボックスが比較的大きい場合、メモリの要求は重要ですが、通常は3D自動精密化最後の反復実行時よりは大きくありません。 | ||
| + | |||
| + | プログラムは、モーショントラックの線形フィッティング、電子線ダメージの計量のBファクタおよびスケールファクタを推定するための単一フレーム再構成を実行し、新しい光沢のある粒子を作成します。推定されたBファクタとスケールファクタは、これらのファクタを計算するために使用されたGuinierプロット(および直線とのフィット)と共に、そして動作軌跡と線形フィットはすべて、【Display】ボタンから開けるPolish/first3dref/logfile.pdf という大きなPDFファイルにあります。 | ||
| + | |||
| + | 私たちはサンプルが大きく動く最初の1-2フレームではしばしば比較的大きな負のBファクタを観測し、その後に電子線ダメージが入る後のフレームでも観測します。このチュートリアルのムービーはムービーフレームあたり1電子/Å2で記録されているため、16電子/Å2の合計線量はそれほど高くなく、電子線損傷のみが重要な役割を果たす訳ではありません。多くの場合、最初の数フレームは比較的悪い可能性があるが、GuinierプロットのY軸(すなわち、線形強度スケールファクタ)を備えたフィッティングされたラインの切片は線量とともに減少する。 | ||
| + | |||
| + | 何らかの形で推定されたBファクタまたはスケールファクタが奇妙に見える場合、Guinierプロットを通じて線形フィットを調べることができます。時には、Guinierプロットは、【Damage】タブに入力された全周波数領域にわたって直線でよくフィットすることができません。その後、異なるパラメータでjobを再実行する価値があります。また、シングルフレームの再構成が上記の20Åの周波数を超えて拡張されない場合、ラインにフィットせず、(推定されたB因子についてNaNを与える)プログラムは動作しません。そのような場合、Bファクタの計量をスキップするか、または同じタブの Average frames B-factor estimation: 1 を使用して複数のフレームを平均することを選択します。【Display】ボタンから出力された研磨粒子(shiny.star)を使ってSTARファイルを視覚化して、すべてが正しく行われていることを確認することは、おそらく悪い考えではありません。 | ||
| + | |||
| + | 何らかの形で研磨結果に満足していない場合は、【Continue run】ボタンを使用して計算の一部を繰り返すことができます。 それをクリックすると、プログラムは次のファイルを探し、そこにあればその計算ステップをスキップして次のステップに移ります: | ||
| + | |||
| + | ●frame0??_half?_class001_unfil.mrc (これらの地図のいずれかが欠落している場合は、再計算されます。それらが既に存在する場合、それらの再構成計算はスキップされる。Average frames B-factor estimation オプションを変更した場合は、すべての再構成を再計算すべきです。その場合は、古いものを続けるのではなく、新しいjobを開始する方が良いでしょう。あるいは、既存のフレームの再構築をすべて取り除く必要があります。) | ||
| + | |||
| + | ●bfactors.star (このファイルが見つからない場合、プログラムは既存のすべてのフレーム再構成を読み込み、BファクタとスケールファクタをGuinierプロットを通して線形フィットするかどうか決定します。 したがって、たとえば【Damage】タブでフィットした解像度範囲を変更したが、Average frames B-factor estimation を変更しなかった場合、既存のbfactors.starファイルを削除すると非常に便利なので、新しいBファクタとスケールファクタが計算されますが、すべてのマップをもう再構築する必要はありません。 これはとても高速に進むでしょう。) | ||
| + | |||
| + | ●shiny.star (このファイルがない場合、既存の bfactors.starファイルが読み込まれ、磨き上げられた粒子(polished particles)はこれらのパラメータに従って再計量されます。 たとえば、以前に決定されたBファクタとスケールファクタのプロットに滑らかな曲線をフィットさせることを決定し、既存の bfactors.starファイルを新しくフィットしたものに置き換えた場合など、これは非常に便利です。 この場合、プログラムは磨き上げられた粒子(polished particles)のみを再作成します。) | ||
| + | |||
| + | === 9.4 Re-refine the polished particles(磨き上げられた(洗練された)粒子の再精密化) === | ||
| + | shiny.starの磨かれた粒子は、元のものと比べてシグナル対ノイズ比が向上しています。 これは、さらに分類分けするときに役立ちます。 この時点での異なる分類分けの戦略の詳細な説明については、Methods in Enzymology [[#ref12|[12]]]の最新の章を参照してください。 この章はまだオンラインでは入手できないため、以下コマンドで初期の資料をダウンロードできます。 | ||
| + | |||
| + | wget ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relionreview2016.pdf | ||
| + | |||
| + | 私達はいつでもpolished particlesを使って最終的な再構成【3D auto-refine】を実行します。より高い解像度マップをもたらすように、「確率加重 angular assignment(角度の割り当て) 」を実行する替わりに、最も可能性の高い方向として各粒子の角度を決定します。したがって、shiny.star を入力として3D auto-refineを繰り返してください。 この精密化の後、後処理と局所分解の推定のやり直しをすることを忘れないでください。 | ||
| + | |||
| + | このテストケースでは、【movie-refinement】 と 【particle polishing】 の前に、再構成は、既に(ダウンサンプリングされた)データのナイキスト周波数に達しました。 したがって、後者は三次元画像にほとんど影響しませんでした。 このデモンストレーションは、動画処理の仕方を教える上でまだ役立つことを願っています。 | ||
| + | |||
| + | == 10 Wrapping up(まとめ) == | ||
| + | どうやって最終的な再構築をするのか気になるでしょう。[Finished jobs]リストから実行する最後の動作を選択し、【Job actions】ボタンからMake flowchartオプションを試して下さい。これを実行するには、【LATEX】や【TikZ】をコンピューターにインストールする必要があります。最初のフローチャートはどうやってここまで終わらせるかについて示します。10ステップ以上からなるフローチャートは複数の塊に分けられることに注意してください。ワークフローにブランチ(分岐)があるかもしれません。そのため、最後のjobのフローチャートに従った後にも、またそれぞれのブランチでフローチャートがあるでしょう。ブルーボックスをクリックすることで、PDBファイル内の位置に相当するデータを得られます。最後のページには、正確なjob名のないフローチャートが表示されます。これは、出版目的に役立ちます。 | ||
| + | |||
| + | 以上です!このチュートリアルを楽しんで、それが役に立つことを願います。もし他にRELIONについて質問があるならば、最初にRELION WikiのFAQとCCPEMメーリングリストを確認してみてください。もし望んだ情報が得られなければ、CCPEMリストを利用して質問してみてください。決してSjorsに直接メールをしないで下さい。質問すべてに返答できるわけではありませんのでご了承下さい。 | ||
| + | |||
| + | もしRELIONが利用者の研究に有益であると判断できましたら、私たちの[http://www2.mrc-lmb.cam.ac.uk/groups/scheres/publications.html 論文]を引用したり、周りに伝えてみてください。 | ||
== References == | == References == | ||
| − | [1] Xiao-chen Bai, Eeson Rajendra, Guanghui Yang, Yigong Shi, and Sjors HW Scheres. Sampling the conformational space of the catalytic subunit of human -secretase. eLife, 4, December 2015. | + | <span id="ref1">[1]</span> Xiao-chen Bai, Eeson Rajendra, Guanghui Yang, Yigong Shi, and Sjors HW Scheres. Sampling the conformational space of the catalytic subunit of human -secretase. eLife, 4, December 2015. |
| + | |||
| + | <span id="ref2">[2]</span> Shaoxia Chen, Greg McMullan, Abdul R. Faruqi, Garib N. Murshudov, Judith M. Short, Sjors H W Scheres, and Richard Henderson. High-resolution noise substitution to measure overfitting and validate resolution in 3d structure determination by single particle electron cryomicroscopy. Ultramicroscopy, 135:24–35, December 2013. | ||
| + | |||
| + | <span id="ref3">[3]</span> Timothy Grant and Nikolaus Grigorieff. Measuring the optimal exposure for single particle cryo-EM using a 2.6 reconstruction of rotavirus VP6. eLife, 4:e06980, 2015. | ||
| + | |||
| + | <span id="ref4">[4]</span> Alp Kucukelbir, Fred J Sigworth, and Hemant D Tagare. Quantifying the local resolution of cryo-EM density maps. Nature methods, 11(1):63–65, January 2014. | ||
| + | |||
| + | <span id="ref5">[5]</span> Xueming Li, Paul Mooney, Shawn Zheng, Christopher R Booth, Michael B Braunfeld, Sander Gubbens, David A Agard, and Yifan Cheng. Electron counting and beam-induced motion correction enable near-atomicresolution single-particle cryo-EM. Nature methods, 10(6):584–590, June 2013. | ||
| + | |||
| + | <span id="ref6">[6]</span> Joseph A. Mindell and Nikolaus Grigorieff. Accurate determination of local defocus and specimen tilt in electron microscopy. Journal of Structural Biology, 142(3):334–347, June 2003. | ||
| + | |||
| + | <span id="ref7">[7]</span> Alexis Rohou and Nikolaus Grigorieff. CTFFIND4: Fast and accurate defocus estimation from electron micrographs. Journal of Structural Biology, 192(2):216–221, November 2015. | ||
| + | |||
| + | <span id="ref8">[8]</span> Peter B Rosenthal and Richard Henderson. Optimal determination of particle orientation, absolute hand, and contrast loss in single-particle electron cryomicroscopy. Journal of Molecular Biology, 333(4):721–745, October 2003. | ||
| + | |||
| + | <span id="ref9">[9]</span> Sjors H W Scheres. Classification of Structural Heterogeneity by MaximumLikelihood Methods. In Cryo-EM, Part B: 3-D Reconstruction, volume 482 of Methods in Enzymology, pages 295–320. Academic Press, 2010. | ||
| + | |||
| + | <span id="ref10">[10]</span> Sjors H W Scheres. A Bayesian view on cryo-EM structure determination. Journal of Molecular Biology, 415(2):406–418, January 2012. | ||
| + | |||
| + | <span id="ref11">[11]</span> Sjors H W Scheres. RELION: Implementation of a Bayesian approach to cryo-EM structure determination. Journal of Structural Biology, 180(3):519–530, December 2012. | ||
| + | |||
| + | <span id="ref12">[12]</span> Sjors H W Scheres. Processing of structurally heterogeneous cryo-em data in relion. Methods in Enzymology, in press, 2016. | ||
| + | |||
| + | <span id="ref13">[13]</span> Sjors H W Scheres and Shaoxia Chen. Prevention of overfitting in cryo-EM structure determination. Nature methods, 9(9):853–854, September 2012. | ||
| + | |||
| + | <span id="ref14">[14]</span> Sjors H W Scheres, R. Nunez-Ramirez, C. O. S Sorzano, J. M Carazo, and R. Marabini. Image processing for electron microscopy single-particle analysis using XMIPP. Nature Protocols, 3(6):977–90, 2008. | ||
| + | |||
| + | <span id="ref15">[15]</span> J M Smith. Ximdisp–A visualization tool to aid structure determination from electron microscope images. Journal of structural biology, 125(2-3):223–228, May 1999. | ||
| + | |||
| + | <span id="ref16">[16]</span> Guang Tang, Liwei Peng, Philip R. Baldwin, Deepinder S. Mann, Wen Jiang, Ian Rees, and Steven J. Ludtke. EMAN2: an extensible image processing suite for electron microscopy. Journal of Structural Biology, 157(1):38–46, January 2007. | ||
| + | |||
| + | <span id="ref17">[17]</span> Kutti R. Vinothkumar, Greg McMullan, and Richard Henderson. Molecular Mechanism of Antibody-Mediated Activation of -galactosidase. Structure, 22(4):621–627, April 2014. | ||
| − | [ | + | <span id="ref18">[18]</span> Kai Zhang. Gctf: Real-time CTF determination and correction. Journal of Structural Biology, 193(1):1–12, January 2016. |
| − | + | == FAQ == | |
| + | Q1. ルートネームとはなんですか。 | ||
| + | A1. Relionでは、ルートネーム+画像の意味を表す拡張+フォーマットを表現する拡張子といった形で、それぞれの画像を表現しています。同じ画像に由来するものはルートネームを共通にもつ必要があります。 | ||
| − | |||
| − | + | == 脚注 == | |
| + | {{Reflist}} | ||
2017年6月1日 (木) 19:38時点における最新版
前書き
ここに示したものは、Relion2のTutorialをDr. Scheresに許可を得て、日本語訳を行ったものを元にし、それに注釈等を加えたものです。誤訳等がある場合には、ご連絡頂けますと幸いです。少しずつ、良いものにしたいと思います。
@http://www.yasunaga-lab.bio.kyutech.ac.jp/EosJ/index.php?title=RELIONチュートリアル
目次
- 1 1 Getting prepared(準備)
- 1.1 1.1 Recommended reading(参考文献)
- 1.2 1.2 Install MPI(MPIのインストール)
- 1.3 1.3 Install CUDA(CUDAのインストール)
- 1.4 1.4 Install RELION(RELIONのインストール)
- 1.5 1.5 Install motion-correction software(モーション補正ソフトウェアのインストール)
- 1.6 1.6 Install CTF-estimation software(CTF推定ソフトウェアのインストール)
- 1.7 1.7 Install RESMAP(RESMAPのインストール)
- 1.8 1.8 Download the test data(テストデータのダウンロード)
- 1.9 1.9 キューイングシステムの利用
- 2 2 What's new in RELION-2.0?
- 2.1 2.1 The GUI(GUIについて)
- 2.1.1 2.1.1 A pipeline approach(パイプラインアプローチ)
- 2.1.2 2.1.2 The upper half: jobtype-browser and parameter-panel(上半分:jobタイプブラウザとパラメータパネル)
- 2.1.3 2.1.3 The lower half: job-lists and stdout/stderr windows(下半分:jobリストとstdout/stderrウィンドウ)
- 2.1.4 2.1.4 The Display button(ディスプレイボタン)
- 2.1.5 2.1.5 The Job action button(jobアクションボタン)
- 2.1.6 2.1.6 Clean-up to save disk space(ディスク領域を節約するためのクリーンアップ)
- 2.2 2.2 Optimise computations for your setup(それぞれの環境における計算を最適化)
- 2.3 2.3 On-the-fly processing running scheduled jobs(スケジュール化されたジョブのon-the-fly実行(撮影しながら処理を実行))
- 2.4 2.4 Helical processing(ヘリカル処理)
- 2.5 2.5 Other new functionalities(他の新しい機能)
- 2.6 2.6 How this tutorial works(本チュートリアルでの動作法)
- 2.1 2.1 The GUI(GUIについて)
- 3 3 Preprocessing(前処理)
- 3.1 3.1 Getting organised (解析の準備)
- 3.2 3.2 Beam-induced motion correction(電子線により誘起される画像の動きの補正)
- 3.3 3.3 CTF estimation(CTF推定)
- 3.4 3.4 Manual particle picking(手動粒子抽出(座標の設定))
- 3.5 3.5 Particle extraction(粒子の抽出)
- 3.6 3.6 Making templates for auto-picking(自動検出のためのテンプレート作成)
- 3.7 3.7 Selecting templates for auto-picking (自動検出のためのテンプレート選択)
- 3.8 3.8 Auto-picking(自動検出)
- 3.9 3.9 Particle sorting(粒子ソート)
- 4 4. Reference-free 2D class averaging (参照無2次元クラス平均)
- 5 5. Unsupervised 3D classification (教師無3次元クラス分類)
- 6 6 High-resolution 3D refinement(三次元精密化)
- 7 7 Mask creation & Postprocessing(マスク作成&後処理)
- 8 8 Local-resolution estimate(ローカル解像度推定)
- 9 9 Moving-processing & particle polishing
- 10 10 Wrapping up(まとめ)
- 11 References
- 12 FAQ
- 13 脚注
1 Getting prepared(準備)
RELIONのインストールに関わる他の情報は下記を参考にして下さい。
[RELION]
1.1 Recommended reading(参考文献)
RELIONの精密化処理に関する理論的詳細は、以下の論文で述べられています。
●S.H.W. Scheres (2012) "RELION: Implementation of a Bayesian approach to cryo-EM structure determination" J.Struc. Biol., 180, 519-530.
●S.H.W. Scheres (2012) "A Bayesian view on cryo-EM structure determination" J.Mol. Biol. 415, 406-418.
RELIONの使い方に関する、包括的な概要は以下の論文が参考になります。
●S.H.W. Scheres (2016) "Processing of structurally heterogeneous cryo-EM data in RELION" Meth. Enzym., in press.
1.2 Install MPI(MPIのインストール)
計算量が多いため、並列処理による高速化のためにMPI(massage passing interface)を利用しています。MPIがインストールされた計算機クラスタ、もしくは、NVIDIA GPU搭載のマルチコアデスクトップマシンが必要になることに注意してください。RELIONをコンパイルするには、mpi-develパッケージが必要です。まだmpi-develがインストールされていない場合は、openMPI([http://www.open-mpi.org])をインストールすることをお勧めしますが、恐らく、その種類(openMPI、MPICH、LAM-MPIなど)またはバージョンはそれほど重要ではありません。
※ MPIは、マルチプロセスの並列処理により高速化を行うための共通プロトコルの一つです。
1.3 Install CUDA(CUDAのインストール)
NVIDIAの比較的新しいGPU(compute capability 3.5+以上)をお持ちの場合、オートピッキング、分類、精密化作業を高速化することができます。GPUアクセラレータをサポートするRELIONをコンパイルするには、CUDAをインストールする必要があります。このチュートリアルの準備の際には、CUDA-7.5を使用しました。 NVIDIAのWebサイトからダウンロードしてください。
※ CUDAは、NVIDIA社が開発した、GPU(グラフィカル・プロセッシング・ユニット)を用いて、マルチスレッドの並列計算を行うためのライブラリです。
1.4 Install RELION(RELIONのインストール)
RELIONはオープンソースのソフトウェアです。the RELION wikiから無料でダウンロードして、手順に従ってインストールしてください。
もしジョブ投函システム(Sun Grid EngineやPBS/TORQUEなど、ジョブの順次処理(ジョブキューイングシステム)に詳しくない場合は、インストール手順の説明にあるように、qsub.cshスクリプトの設定に関して、システム管理者に助けを求めて下さい。
皆さんは、分散メモリ並列化のためのMPIと、共用メモリの並列化のためのpthreadsの両方を使用する、いわゆるハイブリッド並列計算を実行したいと考えるでしょう。その場合、ジョブ投函キューシステムは、これを可能にするためにいくつかの調整が必要な場合があります。その場合は再度、システム管理者に問い合わせてください。
※ ジョブキューイングシステム(job submission queueing system) キュー(待ち行列)とは、実行したいと考えるジョブを投函した後、投函されたものから順番に実行するためのシステムです。一台のホストを大人数で使用する場合などには、一度に利用するとホストのメモリやCPU が足りなくなり、利用できなくなったり、異常に遅くなったりします。そのため、計算機リソースを順番に使うために利用されています。一台を1人で使う場合などには余り考える必要はありません。
※プロセスとスレッド プロセスとスレッドはいずれも並列に動作させる単位を指していますが、その実行方法が異なります。プロセスは仮想計算機と呼ばれ、メモリ空間・CPUの利用等が独立して動く事になります。そのために、複数のプ ロセスを並列して動かすためには、プロセス間で情報の受け渡し(message passing)が必要になります。MPIはその情報の受け渡しのための一つのプロトコル(規格)です。 それに対してスレッドは
1.5 Install motion-correction software(モーション補正ソフトウェアのインストール)
RELION-2.0は、全フレーム顕微鏡写真ムービーアライメント[5]に使用される、UCSFプログラムMOTIONCORRへのラッパーを提供します。Yigan Chengのページからプログラムをダウンロードし、インストール手順に従ってください。また、Niko GrigorieffのグループからUNBLUR [3]を使用することもできます。 Niko's UNBLUR Webサイトからダウンロードしてください。
※ 全フレーム顕微鏡写真ムービーアライメント(Motion Correction(モーション補正))とは、CMOSカメラ等で撮影された連続写真を使って、グリッドのドリフト(移動)などの補正を行う為の操作です。
※ ここで述べるラッパーとは、他のプログラム等と結合するためのプログラムのことを示しています。
1.6 Install CTF-estimation software(CTF推定ソフトウェアのインストール)
CTF推定は、RELIONには含まれていません。代わりに、RELIONは、Niko GrigorieffのCTFFIND3 [6]、またはAlexis Rohou、CTFFIND4 [7]によって書かれたこのプログラムの次世代へのラッパーを提供します。Niko's CTFFINDのWebサイトからダウンロードし、インストール手順に従ってください。また、あなたのマシンにNVIDIAグラフィックスカード(GPU)を持っている場合は、Kai ZhangのGCTF [18]を使用することもできます。これはKai's website at LMBからダウンロードできます。
#CTF (Contrast transfer function)とは、電子顕微鏡写真における撮影上の変調を表現した関数(フーリエ空間で定義)です。
1.7 Install RESMAP(RESMAPのインストール)
局所分解能推定もRELIONの一部ではありません。 代わりに、RELIONはAlp KucukelbirのRESMAP [4]にラッパーを提供します。 Alp'sRESMAP websiteからダウンロードし、インストール手順に従ってください。
# RESMAPは、再構成されたプログラムを利用して、場所毎の分解能を計算するためのプログラムです。
1.8 Download the test data(テストデータのダウンロード)
このチュートリアルでは、さまざまなベータガラクトシダーゼの再構成を計算するために使用される顕微鏡写真のサブセットであるテストデータセットを使用しています[13,2,17]。計算負荷を軽減するために、これらのデータを2倍ダウンサンプリングしたものを利用しています。データは、直接ブラウザからftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion20_tutorial.tar.gzダウンロードするか、次のコマンドを使用してダウンロードして解凍することができます。
wget ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion20_tutorial.tar.gz gunzip relion20_tutorial.tar.gz tar -xf relion20_tutorial.tar
より便利な使い方
また、日本国内からアクセスしている場合以下のURLの方が高速かもしれません。
http://www.yasunaga-lab.bio.kyutech.ac.jp/pub/relion20_tutorial.tar.gz Checksum MD5: c62ff74922283389c5318f3e852365ce
ダウンロードしたファイルを解凍する際には、「pigz」プログラムを使うと更に高速です。「pigz」プログラムは、aptまたはyumで入手できます。それぞれのOSでのインストール方法は、
Ubuntu: apt install pigz CentOS: yum install pigz
です。インストールには管理者権限が必要です。また、このファイルの解凍方法は、
tar xvf relion20_tutorial.tar.gz --use-compress-program=pigz
です。おおよそ、場合によりますがおおよそ1.5-2倍程度高速化されます。
1.9 キューイングシステムの利用
このチュートリアルに従って演習をする際に、独自のコンピュータシステムを使用している場合には注意が必要です。特に、計算機クラスタで実行するには、それぞれ特定のキューイングシステム用に構成されたqsub.cshスクリプトが必要となるでしょう。 正しいスクリプトを持っているかどうかわからない場合は、システム管理者に相談してください。
2 What's new in RELION-2.0?
2.1 The GUI(GUIについて)
2.1.1 A pipeline approach(パイプラインアプローチ)
RELION-1.4のGUIは、便利なコマンドラインプログラムを自動生成するだけにすぎませんでしたが、RELION-2.0のGUIは、新しいパイプラインのアプローチにおいて、より中心的な役割を果たすようになりました。
以前のバージョンでは、ユーザーがそれぞれの異なるjobから出力ファイルを紐づけする必要がありました。また、以前のプログラムはプロジェクトの履歴を認識できませんでした。
RELION-2.0では、GUIがすべてのjobの動作経歴と、それぞれのjobがどのような出力をし、それを他のjobに入力したのかを保存します。そのため、パイプラインを形成できます。その結果、ユーザは、出力ファイルがどこに格納されるかを、ユーザ自身が管理しておく必要は無くなりました。各jobタイプはそれぞれ独自の出力ディレクトリがあり、これらのjobタイプのディレクトリ内(例えばClass2D)では、新しいjobは連続する番号(Class2D/job010)が与えられます。これら個々のjobディレクトリ内では、Class2D/job010/runのように出力名が固定されています。各jobに意味のある名前を付ける仕組みを提供するために、ファイルシステム上の個々のjobディレクトリへのシンボリックリンクとして実装されたジョブ "エイリアス"のシステムが使用されています。
パイプラインに関するすべての情報は、default_pipeline.starというファイルに保存されますが、通常の状況ではユーザーはこのファイルを調べる必要はありません。
※ ここで述べるパイプラインとは、一つのプログラムの出力を次のプログラムの入力として順次繋いでいくような情報の受け渡し方法のことを示しています。
2.1.2 The upper half: jobtype-browser and parameter-panel(上半分:jobタイプブラウザとパラメータパネル)
GUIは上半分と下半分に分かれています。上半分はRELION-1.4に似ています。左側にjobタイプ(【2D classification】のようなjobタイプを選ぶ縦の)ブラウザがあります。このリストはRELION-2.0で拡張されました。右側には複数のタブがあるパネルがあり、それぞれのjobタイプへのパラメータを入力できます。GUIの左上には3つの異なるメニューがあり、さまざまな機能が提供されています。RELION1.4の【Run!】ボタンが【Schedule】と【Run now!】の2つの新しいボタンに置き換えられました。前者のものは新しく、今後の実行のためにjobをスケジュールするために使用できます。これは、データが収集される際にリアルタイムで反復的に実行できる完全に自動化された「パイプライン」の準備に特に役立ちます。詳細は以下を参照してください。GUIの左側のjobtype-browserをクリックすると、新しい動作(【Run now!】ボタン付き)が右側のパラメータパネルにロードされます。
2.1.3 The lower half: job-lists and stdout/stderr windows(下半分:jobリストとstdout/stderrウィンドウ)
GUIの下半分には、まだ実行中のジョブ(【Running jobs】)、すでに完了しているジョブ(【Finished jobs】)、または後で実行するようにスケジュールされたジョブ(【Scheduled jobs】)のリストが含まれています。これらのリスト内の動作をクリックすると、その動作のパラメータがパラメータパネルにロードされ、【Run now!】ボタンの色が変わり、【continue now!】に変わります。それをクリックすると、新しい出力jobディレクトリは作成されませんが、パラメータパネルで指定されたパラメータに従ってjobが続行されます。【2D classification】、【3D classification】、【3D auto-refine】jobでは、_optimiser.starファイルが必要になり、ファイル名には、run_ct23のような継続された繰り返しのファイル名が付きます。他のjobタイプでは、それらが以前に実行されるまで、その時点から継続することができます。【モーション補正】、【CTF推定】、【自動ピッキング】、【粒子抽出】は、これまでに行われていなかった顕微鏡写真でのみ実行されます。【Input to this job】(このjobへの入力)と【Output from this job】(このjobからの出力)は、リンクjobをまとめてリストし、プロジェクト履歴内を前後にブラウズするために使用できます。
GUIの下半分の下部には、選択された(実行済みまたは実行中の)jobの標準出力(stdout)と標準エラー出力(stderr)がそれぞれ黒と赤のテキストで表示されます。は理想的には空白でなければなりません。これらのテキスト表示は、jobリスト内のjobをクリックするたびに更新されます。stdoutまたはstderrのディスプレイをダブルクリックすると、より便利なスクロールのためにテキスト全体を含むポップアップウィンドウが開きます。
2.1.4 The Display button(ディスプレイボタン)
RELION-1.4には、GUI上に汎用の【Display!】ボタンがあり、粒子、顕微鏡写真、マップ、マスクなどの表示に使用できました。一般的なファイルブラウザが開いて、これらのファイルを選択することができました。このボタンは、RELION-2.0のGUIからは消えましたが、GUIの左上にある【File】メニューの【Display】オプションを使用してアクセスすることができるように変わっています。
【便利になった点】RELION-2.0では、汎用の【Display】ボタンの代わりに、【Run】ボタンと【Schedule】ボタンの下に、もっと特別な【Display:】ボタンができました。GUIの下半分にあるjobリストのうちのひとつのjobを選択した後にこのボタン【Display:】をクリックすると、選択されたjobの入力と出力がすべて(例えば、パーティクル、顕微鏡写真、座標、 PDFファイルなど)がポップアップメニューとして表示できます。 これにより、新しい【Display】ボタンはファイルブラウザを起動する必要がなくなりました。
もう1つの違いは、RELION-1.4での【Display】ボタンは、(_model.starを表示することによって)クラスファイルを選択するために使用されていましたが、RELION-2.0では、【Display:】ボタンでこれを行うことができなくなりました。その代わりに、【Subset selection】の新しいjobタイプができました。このjobでは、クラスや顕微鏡写真を選択することができます。このjobタイプには、顕微鏡写真を再グループ化し、質量中心に応じて自動的に2Dクラス平均をセンタリングできる(中心を移動させる)タブもあります。後者は、これらのクラス平均を自動ピッキングのテンプレートとして使用する場合に便利です。
2.1.5 The Job action button(jobアクションボタン)
【Job action】ボタンは、選択した(実行中、完了後、またはスケジュールされた)ジョブの設定を含む小さなメニューを開きます。ここで、note.txtというファイルにアクセスすることができます。このファイルは個々のジョブディレクトリに保存されており、ユーザーのコメントを格納したり、jobのエイリアスを変更したり、jobを終えたものをマークすることができたり、job履歴のフローチャートを作ったり(LATEXとTikZパッケージがシステムにインストールされている場合は、第10章を参照してください)、ディスク容量を節約するためにjobを削除またはクリーンアップ(下記参照)したりすることができます。
2.1.6 Clean-up to save disk space(ディスク領域を節約するためのクリーンアップ)
jobを削除すると、jobディレクトリ全体がProjectディレクトリからTrash/というディレクトリに移動します。RELIONの左上にある【File】メニューの【Trash】フォルダを空にすると、完全に削除され空き領域が増えます。これを行うまでは、左上の[job]メニューから対応する設定を使用して、jobを復元することができます。
ディスク容量を節約するため、jobを「clean」にすると、refine jobのすべての中間的な繰り返しに対して書き出されたファイルのような中間ファイルをごみ箱フォルダに移動します。2つのクリーニングオプションがあり、片方は【gentle clean】穏やかなクリーニング方法です。この方法では、他のジョブへの入力として使用できるすべてのファイルをそのまま残します。もう一方は【harsh clean】完全なクリーニング方法です。この方法では全てのファイルを消しますので、より多くの容量を確保でき、【Motion correction】、【Particle extraction】、【Movie refinement】、【Particle polishing】のjobタイプから選択できる粒子スタックまたは顕微鏡写真を含むディレクトリに関しては特に、大きく容量を確保できます。
また、RELIONの左上にある【job】メニューの対応するオプション(【Gently clean all jobs】あるいは、【Harshly clean all jobs】)を使用して、プロジェクト内のすべてのディレクトリをワンクリックで消去することもできます。 その際、特定のディレクトリを削除対象から除外したい場合には、NO_HARSH_CLEANというファイルをその中に置いてください。
例えば、保護したい粒子データがjob098にある場合、次のコマンドを実行してください。
$ touch Polish/job098/NO_HARSH_CLEAN ※ touchコマンドは、指定したファイルが存在しない場合には空のファイルを作成するプログラムです。存在している場合には、ファイルの修正時刻を変更します。
2.2 Optimise computations for your setup(それぞれの環境における計算を最適化)
2.2.1 GPU-acceleration(GPUによる高速化)
Erik Lindahl(ストックホルム)のグループのDari KimaniusとBjoern Forsbergは、RELIONの計算コストの高い部分を移植して、GPUを使うようにしました。彼らはNVIDIAのcuda-librariesを使ってこの作業を行ったため、RELION内のGPUアクセラレータはnvidiaカードでしか動作しません。これらは、NVIDIAのGPUが、compute campacity 3.5以上である必要があります。単精度と倍精度の両方のカードが動作するため、高価な倍精度カードに限らず安価なゲーミングカードを使用することもできます。
2つの異なるRELIONプログラムがGPUにより高速化されています。それは、relion_autopick(自動ピッキング)とrelion_refine(2次元分類、3次元分類、3次元自動精密化job)です。これらのプログラムのシーケンシャル版とMPI版の両方が加速されました。
2.2.2 Disk access(ディスクへのアクセス)
GPUアクセラレータによって画像処理の速度がはるかに向上したために、ハードディスクへのアクセスがますますネックになります。RELION-2.0のGUIには、データセットとコンピュータセットアップのディスクアクセスを最適化するためのいくつかのオプションが用意されています。2次元分類、3次元分類、3次元自動精密化のために、パラレルディスクI/Oの使用【use parallel disc I/O】を選択できます。Yesに設定すると、すべてのMPIプロセスがハードディスクから同時に粒子を読み込みます。それ以外の場合は、マスターだけがイメージを読み取り、ネットワークを介してスレーブに送信します。fhgfsのglusterのような並列ファイルシステムは、並列ディスクI/Oで優れています。NFSは、多くのスレーブが並行して読み込みを行うと中断することがあります。
メモリ中にプールされた粒子の数【number of pooled particles】を設定することもできます。粒子はMPIスレーブによって個々のバッチで処理されます。各バッチ中に、粒子画像のスタックは、一度だけ開閉され、ディスクアクセス時間を改善します。1つのバッチのすべての粒子画像は、一度にメモリに読み込まれます。これらのバッチのサイズは、使用されるスレッド当たり少なくとも粒子1つ分です。このパラメータは、スレッドごとにバッチ内で一緒に読み込まれる粒子の数を制御します。例えば、3に設定し、1つにつき8つのスレッドを使用する場合、3x8 = 24個の粒子のバッチが一緒に読み込まれます。これにより、ディスクアクセス、特にディスクアクセスのメタデータ処理が問題となるシステムでのパフォーマンスが向上する可能性があります。それは、控えめなRAM使用を与えます。
比較的小さなデータセット(および/または大量のRAMを持つコンピュータ)を使用する場合は、計算の開始時にすべての粒子をRAMに事前に読み込むことができます【pre-reqd all particles into RAM】。これにより、ディスクアクセスが比較的遅いシステムでの計算が大幅に高速化されます。ただし、使用可能なRAMの量には注意が必要です。粒子はフロート精度で読み込まれるため、N個の粒子をRAMに読み込むには(N×boxsize×boxsize×4)/(1024×1024×1024)GBが必要です。200ピクセルのボックスサイズを持つ100,000個の粒子は15Gbになり、400ピクセルのボックスサイズでは同じ数の粒子に対して60Gbになります。
データセットが大きすぎてRAMに先読みできないが、各計算ノードに同じ名前でマウントされたローカルの高速ディスク(ソリッドステートドライブなど)(スクラッチディスクと呼ぶ)がある場合、各MPIスレーブはすべての粒子を 計算を開始する前にローカルディスクを使用することができます。これは、粒子をスクラッチディレクトリにコピーすることによって行われます【Copy particles to scratch directory.】。複数のスレーブが同じノード上で実行される場合、最初のスレーブだけが粒子をコピーします。ローカルディスクが小さすぎてデータセット全体を保持できない場合、もはやスクラッチディスクに置くことができない粒子は元の場所から読み込まれます。relion_volatileというサブディレクトリが、指定されたディレクトリ名の内部に作成されます。例えば、/ ssdを指定すると、/ ssd / relion_volatileという名前のディレクトリが作成されます。 / ssd / relion_volatileディレクトリがすでに存在する場合は、粒子をコピーする前に消去されます。 次に、プログラムはすべての入力粒子をこのディレクトリ内の単一の大きなスタックファイルにコピーします。 ジョブが正常に終了すると、/ ssd / relion_volatileディレクトリが再度削除されます。
終了する前にjobがクラッシュした場合は、それを自分で削除したいと思うことでしょう。このとき、プログラムは、全ての人の書き込み権限付きで/ ssd / relion_volatileディレクトリを作成します。それにより、スクラッチディレクトリとして、/ ssdを、すなわちユーザ名なしで使用することを選択することができます。そのほうが、各計算ノード上の1人のユーザーが常に1つのjobしか実行しないという条件で、ローカルディスクは、jobがクラッシュし、ユーザーがスクラッチディスクのクリーニングを忘れたときでも、ジャンクでいっぱいになる危険性はありません。(つまり、全員が同じスクラッチディレクトリ名を使えば、毎回、スタート時にクリアすることになるので、ジャンクを消し忘れるといったことがないという意味です。)
最後に、ディスクを通して繰り返しを組み合わせるオプション【combine iterations through disc】があります。Yesに設定すると、すべてのMPIスレーブはすべての繰り返しの最後に、蓄積された結果をひとつの大きなファイルに書き出します。MPIマスターはこれらすべてのファイルを読み込み、それらをすべて結合し、結合された状態で、ひとつの新しいファイルに書き出します。すべてのMPIスレーブは、その後、その結合された結果を読み込みます。これにより、ネットワークの負荷が軽減されますが、ディスクI / Oの負荷が増加します。これは、期待値ステップ(E-step)の終わりに達する進捗バー(マウスがチーズに達する)とその後の最大化ステップ(M-Step)の開始にかかる時間に影響します。この時間は、非常に効率的なシステム設定に依存します。このオプションは当初、LMBでの古いクラスタ上でのネットワークカードのバグを回避するために実装されました。 今日では、精密化が高解像度に達すると非常に遅くなる傾向があるので、このオプションは使用しない方が望ましいです。
2.3 On-the-fly processing running scheduled jobs(スケジュール化されたジョブのon-the-fly実行(撮影しながら処理を実行))
【Schedule】ボタンから、今後のjobの準備をできます。スケジュール化された動作により期待される出力ファイルは、既に【Browse】ボタンから他のjobへの入力として利用できるため、Scheduled jobsに複数の連続した動作の「パイプライン」を構築することが可能です。GUIの左上の【Autorun】メニューからスケジュール化されたjobを実行できます。そのプログラムは何回スケジュール化された動作を実行するか聞いてきます。1より大きい値を指定すると、すべてのスケジュールしたjobが繰り返し実行されます。この設定にした場合、スケジュール化されたjobが一周毎に、何分経過させるべきかという質問が出てきます。(もしスケジュールしたjobの実行時間がこの時間よりも短い場合、このプログラムは、次にスケジュールしてあるjobの実行を待ちます。)jobタイプが、【Import】、【Motion correction】、【CTF estimation】、【Auto-picking】、【Particle extraction】、【Movie refinment】である場合には、以前の反復で完了していない顕微鏡写真のみを処理します。他の全てのjobタイプでは、スケジュールされたjobの実行の各反復後、再度、最初から再計算を行います。
この設定により、顕微鏡写真を収集する際に、高速処理を便利に行うことができます。顕微鏡写真や顕微鏡動画を顕微鏡コンピュータから処理コンピュータ(好ましいのは、高速処理のための1つまたは複数の適切なGPUを搭載しているもの)にコピーするスクリプトを設定することができます。
※大きなファイルをコピーするのには時間がかかり、まだコピーの終わっていない顕微鏡写真や動画をインポート【Import】するのは好ましくありません。そのため、このスクリプトはファイルを一時的なファイル名でコピーし、コピーが完全に終了したときにのみ、ファイルを適切なファイル名に変更するべきでしょう。
例えば、【Micrograghs/*.mrcs】ファイルの【Import】で始まる様に、スケジュールされたjobリストは、適切なテンプレートがすでに用意されている場合に葉、【Motion correction】、【CTF estimation】、【Auto-picking】へと順次実行することができるでしょう。そうでない場合には、いくつかの顕微鏡写真を取得した後で手動で粒子を選択する必要があるでしょう。その後、【Particle extraction】、【2D classification】へと順次進みます。このスケジュールしたjobリストは、それぞれの繰り返し毎に、恐らく少なくとも30分または60分で、何度も繰り返されます。インポートされた顕微鏡写真のリストはますます増えますが、スケジュールしたjob(【Motion correction】、【CTF estimation】、【Auto-picking】、【Particle extraction】)の反復では、新しい顕微鏡写真のみで実行されます。一方で、【2D classification】は、毎回の繰り返し毎に全ての粒子数を最初から計算し直します。電子顕微鏡でデータ集めている間に2次元クラス平均を調べることは、既に最初の場所で撮影された価値のあるデータが得られたかどうか、あるいは、投影方向性分布が大丈夫かどうかを決めるのに有益でしょう。後者の場合には、恐らく、より氷が厚いグリッド上で収集する必要があるでしょう。既に3Dリファレンスが良ければ、【3D classification】または【3D auto-refine】を、並列してそのまま実行できるかもしれません。また、このことは、例えば、ある補因子が目的の複合体に結合しているかどうかを確認するなどの場合に有用なことでしょう。
【subset selection】はスケジュールすることができますが、【2D classification】の_model.starを表示して適切なクラスを選択するなど、入力を求められることに注意してください。全てのjobの実行はユーザーがこの選択を保存するまで続きません。このステップの自動化は次のRELIONのバージョンに組み込ませるつもりです。
2.4 Helical processing(ヘリカル処理)
Scheresグループの博士課程学生であるShaoda Heは、ヘリカルアセンブリ処理のためのワークフローを実装しています。これには、【Auto-picking】、【Partcle extraction】、【2Dclassification】、【3Dclassification】、【3D auto-refine】、【Particle polishing】、および【Mask create】といったjobタイプのパラメータパネルへの追加タブが含まれます。これらオプションのより詳細な説明は、ユーザーがRELION Wikiの関連するページから参照できます。
2.5 Other new functionalities(他の新しい機能)
2.5.1 Dealing with differently scaled particles(異なったスケールの粒子の取扱)
RELION-1.4では、STARファイル等を修正するためのawkスクリプトを含んでいたため、粒子をどれだけダウンスケールするか(あるいはしてないか)に関する変更がいくらか可能でした。RELION-2.0では、もっと手軽に縮小ができるようになりました。【Particle extraction】 jobでは、【Manual picking】、【Auto-picking】から提供された座標の替わりに、【2D classification】、【3D classification】、【3D auto-refine】から _data.starを提供する【re-extract refined particle】オプションを備えています。このオプションは、、抽出された粒子の新しいSTARファイルに原点オフセットを書き出す際、各入力粒子のスケールの違いを正しく説明するでしょう。また入力_data.starファイルの調整された原点オフセットによって定義された中心で粒子を改めて抽出することもできます(【Re-center refined coordinates】オプションを使用します)。
他のRELION-1.4の不便なことは【Auto-picking】でのテンプレートが取得してきた顕微鏡写真と同じスケールでなければならないことです。RELION-2.0ではそうではなく、【Reference】タブ上で【pixel size in references】で値を与える時に(もしくは【I/O】タブ上のpixel size in micrographsオプションを利用時)、入力する顕微鏡写真のSTARファイルで定義されたピクセルサイズと違っててもよいです。
2.5.2 Particle subtraction(粒子の減算)
【Particle subtraction】 jobタイプは、RELION-2.0パイプライン内部で部分的なシグナル減算の手順を正式な手順として採用しています。入力マップの投影像(入力粒子以前の3Dクラス化【3D classifiction】または自動3D精密化【3D auto-refine】からの _data.starファイルとして、提供されます。)から減算されます。マップを投影する前に、入力マスクがマップに適用されます。それゆえに、マスクは、粒子から減算したい領域では白く、それ以外では黒くする必要があります。
【粒子から得られるシグナルをマスクに反映させ、それを一面黒のマップに適用することで、粒子のある領域では白く、それ以外では黒いマップができます】
同じjobタイプで、(入力 _data.starファイル上のコラムを【rlnImageName】と【rlnImageOriginName】切り替えることで)減算された粒子から元の粒子へと戻すことができます。これは、たとえば、データを構造的に同種のサブセットに分類するために部分シグナル減算後に、複合体全体を精密化したい場合などに便利です。シグナル減算の手順の詳細については、eLifeの論文[1]を参照してください。
2.5.3 Join STAR files
RELION-2.0ではパイプラインが公式に採用されたことに伴い、粒子、顕微鏡写真、または顕微鏡動画のSTARファイルへとつなげることができるようになりました。このことは、【join star file】という新しいjobタイプによって行われます。4つ以上のファイルを結合する場合は、GUIからこのユーティリティを複数回呼び出す必要があります(完全に記述されたパイプラインを維持したい場合)。または、【relion_star_combine】コマンドラインプログラムを多数のファイルで実行し、結果のSTARファイルをパイプラインに再度【Import】を用いる方法でもできます
2.6 How this tutorial works(本チュートリアルでの動作法)
RELION-1.4チュートリアルでは、チュートリアルの時間を節約するために、あらかじめ計算された結果をprojectディレクトリにコピーすることができます。RELION-2.0では動作にパイプラインを用いたことにより、同じことができません。かわりに、PrecalculatedResultsディレクトリに完全に実行されたパイプラインを用意しています。ユーザーはbetagalディレクトリ内のすべてのステップを実行することができます(少なくとも1つの適切なGPUを使用していたら、1日以内に終わります)。またはPrecalculatedResultsディレクトリのプロセスに従います。
3 Preprocessing(前処理)
前書き
原理上はRELIONでそれぞれのプログラムから抽出された分子を使用できますが、おすすめできません。多くのプログラムが粒子自体を例えば、段階の切り替えや帯域通過フィルタ、ウィナーフィルター、マスクなどによって変化させているためです。これらはすべてその後のRELIONの使用のために最適とはいえません。さらに、RELIONタイプのSTARファイルにフォーマットされたファイルの中に必須のメタデータを全て収集すると、エラーが発生する傾向があります。RELIONでの粒子の再抽出は単純でとても速いため、後述の手順はRELIONではとても容易な(良い)方法です。
また、RELIONでいくつかのラッパーの実装が報告されています(例えば、EMAN2、SCIPION、APPION)。ラッパーが作られたとき、うまく活用しようと試みましたが、完全には制御できず、これらのラッパーが最適な方法でRELIONを使用できているかどうかわかりませんでした。そのため、ラッパーで得られた結果に疑問がある場合、後述のチュートリアルで説明している手順で行うことをお勧めします。
3.1 Getting organised (解析の準備)
プロジェクト毎、すなわち、解析したい構造ごとに一つのディレクトリを作ることをお勧めします。これをprojectディレクトリと呼ぶことにします。projectディレクトリからRELIONを起動することが重要です。projectディレクトリの中にすべての未加工の顕微鏡写真や顕微鏡動画をMRC形式で保存するための別ディレクトリを作ってください。例えば、すべての顕微鏡写真がひとつのディレクトリにある場合は、通例ではディレクトリをMicrographs/にします。また、異なるディレクトリにある場合(例えば、違う日に収集した顕微鏡写真)はMicrographs/15jan16/やMicrographs/23jan16/などにすると良いでしょう。もし何らかの理由でRELIONのprojectディレクトリの中に顕微鏡写真を入れたくない場合は、projectディレクトリの中に顕微鏡写真が保存されているディレクトリとのシンボリックリンクを作成してください。
単一像(静止画)の顕微鏡写真には.mrc拡張子を、連続スタック像(動画)には.mrcs拡張子をつけてください。単一像顕微鏡写真の代わりに動画を集めた場合でも、それらを直接使用できます。別のプログラムで動画の平均を計算し、さらに、ポリッシングなどの過程で、顕微鏡写真と動画の平均を両方とも使用したい場合、動画のファイル名は顕微鏡写真の平均と同じルートネームにするべきです。つまり、顕微鏡写真の平均のファイルの後に、動画を表現する”_movie"と、スタックファイルを意味する".mrcs"拡張子をつけます。例えば、顕微鏡写真の平均がmic001.mrcの場合、一致する動画はルートネームであるmic001に対して、mic001_movie.mrcsとしてください(動画を表現するのは_movie)。
チュートリアルのテストデータを解凍した時、projectディレクトリ(betagal/)と、記録されたすべての顕微鏡動画と一致するbetagal/Micrographsディレクトリはすでに作成されています。projectディレクトリに行き、以下の入力をすると、どのくらい顕微鏡動画があるか分かります。
cd relion20_tutorial/betagal ls Micrographs/
今回のチュートリアルでは、計算した結果(calcurated_betagal)も配付しています。
cd calcurated_betagal ls Micrographs/
RELIONを起動しましょう。前述の通り、RELIONは常にprojectディレクトリからの起動が必要です。エラーを防ぐために、RELIONは新しいディレクトリで初めて起動したときに確認を要求します。そのため、新しいディレクトリでRELIONを初めて起動するときは、バックグラウンドで起動するための”&”の文字を使うべきではありません。projectディレクトリ内にいることを確認し、以下の入力でRELIONを起動します。
relion
ここで、新しいRELIONプロジェクトを作動するために”y”と答えます。すると、以下のような画面が立ち上がりますので、パイプラインの中へ録画した顕微鏡動画の取り込みを行います。そのため、jobタイプのブラウザから、赤枠で囲まれた”Import”を選択します。
その後、画面が遷移し、入力ファイルを指定する画面になります。左上の赤枠で囲まれたタブが、「I/O」になっていることを確認して、右側の赤枠部のフィールドを以下のように入力します。通常は、プロジェクトディレクトリで起動すると、自動的に指定されているはずです。
Input files: Micrographs/*.mrcs Node type: 2D micrograph movies
Currrent job: Give_alias_hereという空欄に効果的なエイリアスを規定することができます。指定した内容が正しければ、【Run now!】のボタンをクリックし、job起動します。Import/job001/というディレクトリが作成され、一緒にImport/moviesというこのディレクトリへのシンボリックリンクが作成されます。テキストエディターでnote.txtファイルが開かれ、中には実際の実行コマンドが入っており、コメントも記入できます。必要なければ閉じてください。STARファイルとすべての動画が新しいディレクトリの中に作成されます。以下のコマンドを使って中を見ましょう。
less Import/job001/movies.star
違うソフトウェアで粒子を抽出した場合、もしくは、RELION-1.4で抽出した粒子をRELION-2.0に移動させたい場合は、後述の事前処理を行う代わりに、STARファイルや3次元リファレンス、3次元マスクなどを取り込むために”Import”jobタイプを使用してください。
3.2 Beam-induced motion correction(電子線により誘起される画像の動きの補正)
Motion correction jobタイプは便利な全体フレーム動画の調節にためのUCSF MOTIONCORR [5]ラッパーを規定します。【I/O】タブで以下の設定をします。まず、左カラムのジョブタイプを、「Motion correction」を選択し、「I/O」タブが選択されていることを確認し、それぞれのパラメータを設定します。
● Input movies STAR file: Import movies/movies.star
(【Browse】ボタンで参照できるファイルは、標準ではSTAR形式の動画ファイルのみです。)
● Save aligned movie stacks? Yes ● First frame for corrected sum: 1 ● Last frame for corrected sum: 16
(ここでは16フレームの動画で、すべてのフレームを使って平均化します。)
【Motioncorr】タブは以下のように入力します。
● MOTIONCORR executable: /wherever/it/is/motioncorr
(環境変数$RELION_MOTIONCORR_EXECUTABLEを、設定することで、motioncorrプログラムの初期値を変更できます。)
● Binning factor (1 or 2): 1
(超高解像度動画( super-resolution movies)では大抵、2を使用します。)
● First frame for alignment: 1 ● Last frame for alignment:16 ● Bfactor: 150
(超高解像度動画では大きな値を使用します。)
● Which GPUs to use:
(MOTIONCORRはNVIDIA GPU上で動作する必要があります。GPUのデバイス番号を設定します。0と設定すると全てのプロセスが0を使います。0:1と設定するとプロセス毎に0と1を使います。)
● Other MOTIONCORR armuments:
Motioncorrプログラムを選択する際には、
"Show:"部分を、「*.*」から「All Files (*)」へと変更しないと、表示されないかもしれません。Motioncorrタブの設定例は、次のようになります。設定完了後、赤枠の【Run now!】をクリックし、ドリフト補正を行います。
正常にプログラムが進行すると、下部赤枠のようにネズミの顔文字が、右へと進みます。今後のプロセスでも、同様にこの部分に注目することで、プログラムが正常に動作しているかがわかります。
このプログラムの実行にはおよそ1,2分かかります(しかし、この動作にはNVIDIA GPUが必要です)。あるいは、【Unblur】タブで利用できるNiko Grigorieff’s UNBLURプログラム [3]も使用できます。すべてのMOTIONCORR設定は無視され、初期値でUNBLURが実行できるため、高度なオプションが必要ありません。UNBLUR ラッパーはversion 1.0.2でテストされています。計算が終了した後、【Run now!】ボタンの下の【Display:】ボタンからout:logfile.pdfを選択することで推定された電子線によって誘起された変化を見ることができます。または、out:corrected_micrographs.starを選択することで、補正された(値が全て足された)顕微鏡写真を見ることができます。スクリーンサイズに依存して、見やすいように、顕微鏡写真(Scale:0.3)を縮小し、Sigma contrast:3を使い、見やすい数のコラム(例えば、Number of columns: 3(横に3つ並ぶ))にするべきです。この表示(【Display:】ボタンから表示したとき)から顕微鏡写真は選択できないことに注意してください。取り除きたい顕微鏡写真がある場合(今回はすべて良い画像なので、排除しないでしょうが)、【Subset selection】jobタイプが使用できます。
3.3 CTF estimation(CTF推定)
次に、補正した顕微鏡写真からCTFの値を推定します。速度のためKai Zhangの GCTFのためのラッパーとして【CTF estimation】 jobタイプにします。適切なGPUがない場合、Niko Grigorieffの CTFFIND3やAlexis Rohouの CTFFIND4が使えることに留意してください(【CTFFIND】タブの値は初期値が良いでしょう)。CTFFIND4は顕微鏡動画も利用して、よりよいThon ringsを計算したり、顕微鏡動画位相板(Voltatype?)から推定した位相のずれを評価することもできます。
まず、Jobリストより、【CTF estimation】を選択し、【I/O】タブで、【MotionCorr】 jobで作成したcorrected_micrographs.starファイルを選択するために【Browse】ボタンを使います。
また、他のタブは以下のように入力します。
【Microscopy】タブでは実験条件を設定します。
● Spherical aberration(mm): 2
(顕微鏡メーカーがこの値を提供してくれています。)
● Voltage(kV):300
(加速電圧を示します。)
● Amplitude contrast:0.1
(強度コントラストはほとんどないことが知られていますが、10%ほどの値を与えることで、多くの構造の結果が良くなることが示されています。この方法は低周波数の弱い散乱がモデル化されていないためです。クライオ電子顕微鏡法では、7-20%(エネルギーフィルターがある場合)程度であることが知られています。)
● Magnified pixel size(A): 3.54
(今回提供したデータは2倍のビンデータです。もとのピクセルサイズは1.77Aです。)
CTF推定に使用するプログラムをNiko Grigorieffの CTFFIND3、Alexis Rohouの CTFFIND4、Kai Zhangの GCTFから選択することができます。例として、CTFFIND-4.0.8を使用したものを以下に示します。
【CTFFIND】タブでの、CTFFINDの一般的な値を設定します(正確な意味についてはNikoのドキュメンテーションを参照してください)。
● CTFFIND executable: /wherever/is/your/ctffind.exe
環境変数は$RELION_CTFFIND_EXECUTABLEを使うと、初期値から調節でき、同様に【Gctf】タブでGCTF実行ファイルの初期値は$RELION_GCTF_EXECUTABLEで調節できます。CTFFIND-4.0.xを使っている場合、ダブルクォーテーションの間の実行ファイルが実行され、以下の追加の引数を規定します。
CTFFIND-4.1以降を使用している場合は、
“/path/to/ctffind.exe –omp-num-threads 1 –old-school-input”
を追加する必要ありませんが、新しいバージョンで【CTFFIND4】タブを使用する場合は表示が必要です。
● FFT box size(pix): 512
(フーリエ変換のボックスの大きさ)
● Minimun resolution(A): 30
(利用する最低分解能(低周波側にあるノイズはCTFの推定に悪い影響を与えます))
● Maximum resolution(A): 7.1
(利用する細高分解能(今回はナイキスト周波数))
● Minimum defocus cvalue(A): 5000
(想定している最小デフォーカス)
● Maximun defocus cvalue(A): 50000
(想定している最大デフォーカス)
● Defocus step size(A): 500
(推定するデフォーカスの精度)
● Amount of astigmatism(A): 100
(想定している最大の非点量)
● Estimate CTF on window size(pix): -1 (正の値を設定した場合、プログラムは画像の中央を設定したサイズの正方形で切り取り、この部分の画像のみでCTFを推定します。 これは、余計な情報が含まれているスキャンされた画像に対して有効です。)
CTFFIND3かCTFFIND-4.0.xを使うとき、【CTFFIND4】と【Gctf】タブは無視してください。マシンに応じて、複数のMPI処理を使用してプログラムを実行できます。5つのMPI処理を使うと、CTFFIND-4.0.8を使うと動作は5分程度かかります(CTFFIND-4.1では一つの処理に2分半かかり、GCTFを使うと数秒かかります)。一旦jobが終わると、出力のCtfFind/job003/Micrographsという出力ディレクトリの中にそれぞれの顕微鏡写真ごとに追加ファイルができます。.ctfファイルは算出されたパワースペクトルと適合したCTFモデルのMRCフォーマットの画像を含んでいます。.logファイルはCTFFINDかGCTFからの出力を含んでいます(CTFFIND3、4を使用した場合のみ、.comファイルはCTFFINDを起動するために使用したスクリプトを含んでいます)。
CTFFIND-4.1+を使用している場合には、【CTFFIND4】タブで、次のように設定します。
本チュートリアルでは、すでにMotioncor2を使用して、モーションコレクションを行っており、またデータは位相版を使用せずに取得しているため、それぞれのパラメータは、
● Estimate Thon rings from movies?: No
(ドリフトがあるとフレーム間を足し合わせた画像ではThonRingが呆けてしまうため、フレーム毎にフーリエ変換したものを足し合わせます。)
● Estimate phase shifts?: No
(位相板を用いてる場合の設定です。位相変化量の範囲を設定します。) を選択します。
それでは、計算されたThon-ring画像を見てみましょう。RELION操作画面の中央部右側の、【Display:】項目から、「out:micrographs_ctf.star」を選択します。
すると、次のような表示設定を行う画面が現れますので、各種パラメータを与え、【Display!】ボタンをクリックします。ここでは、コントラストや拡大・縮小を設定できます。 【Sort images】にチェックを入れて、【on:】でソート・キーを設定することで、デフォーカス、最大解像度、性能指数(FOM: Figure of Merit)などの順番の表示を並び替えることができます。
実際に、実際に表示される画像を次に示します。
実験画像のThon-ring間のゼロ点とモデルのゼロ点は一致しているべきですが、一致していなければCTFの推定に失敗しています。もしCTFモデルが実験のThon-ringに十分に一致していない場合、それらの顕微鏡写真の.logファイルを削除して。【Finished jobs】リストからCtfFind/job003を選択し、parameter-panelの値を修正し、【Continue now】ボタンをクリックすことで、jobを再実行できます。存在しない.logファイルの顕微鏡写真のみが再処理されます。すべてのCTFモデルが十分に一致するまで実行できます。もしこれでも実行できない、または、不十分なThon-ringがあり、顕微鏡写真を削除すると決めた場合、【Subset selection】 jobタイプで破棄することができます。
3.4 Manual particle picking(手動粒子抽出(座標の設定))
次のManual picking jobタイプでは(平均化された)顕微鏡写真の「粒子座標」を手動で選択して使います。今回のデータをよく説明できるように複数の顕微鏡写真を自分自身で選択するのがお薦めです。選択した粒子を使って、参照無2次元クラス平均を計算します。その後、これらの粒子は全データセットの粒子を自動検出するためのテンプレートとなります。
※ デフォーカスも異なっている複数枚の画像で、様々な投影角の粒子を選択するのがお薦めです。
手動粒子検出は経験あるのみです!RELIONで手動で粒子の検出をするのが好みでなければ、Jude ShortのXIMDISP(任意の拡張子付きで)、XMIPP-2.4(任意の拡張子付きで)、Steven Ludtkeの E2BOXER.PY(.boxの拡張子で)の座標ファイル形式もサポートしています。もしこれらを使う場合、顕微鏡写真(動画)を取り込んだディレクトリと同じディレクトリにテキストファイルとしてまとめた座標ファイルを保存して下さい。そして、同じ顕微鏡写真のルートネームですが、異なる(suffix+)拡張子で保存されていることを確認してください。例えば、顕微鏡写真がMicrographs/006.mrcの時、Micrographs/006.boxやMicrographs/006_pick.starとなります。【Import】 jobタイプを使用し、Node type:を【2D/3D particle coordinates】に設定してください。Imput Files:の欄に整理されたファイルの拡張子に従ってlinux wildcardが含まれていることを確認してください。上記の例であれば、それぞれMicrographs/*.boxやMicrographs/*_pick.starになります。
Manual picking jobタイプの【I/O】タブではCtfFind/job003に作成されたmicrographs_ctf.starファイルを選択するために【Browse】ボタンを使用し、【Colors】タブは無視します。
【Display】タブでは以下のように入力します。
● Particle diameter(A): 200
(これは単に顕微鏡写真に表示された円の直径を調節します。実際に切り出される大きさではありません。)
● Scale for micrographs: 0.5
(これは画面サイズによります。)
● Sigma contrast: 3
(顕微鏡写真はほとんど最適な”sigma-contrast”で表示されています。3に設定した場合、黒が標準偏差の3倍低く、白が標準偏差の3倍高くなります。グレースケールは、黒から白への直線的に変化します。詳細はDisplayImages entry on the RELION wikiを見てください。)
● White value: 0
(どの値を白にするか手動で設定してください。これが動作するには、Sigma contrastは0に設定されるべきです。)
● Black value: 0
(どの値を黒にするか手動で設定してください。これが動作するには、Sigma contrastは0に設定されるべきです。)
● Lowpass filter(A): 20
(ノイズのひどい顕微鏡写真の粒子をより見やすくする動作です。)
● Highpass filter(A): 0
(氷の厚さの違いなどに由来する顕微鏡写真全体の緩やかなグラデーションを取り除くのに役立つことがあります。)
● Pixel size: 3.54
(これは粒子の直径や、low-pass filter、high-pass filterの計算に必要です。)
● Scale for CTF image: 1
(これは単に、【Run now!】後に表示されるウィンドウにおいて、各々の顕微鏡写真の【CTF】ボタンをクリックした時にThon-ring画像がどのくらい大きくなるかを調節します。)
【Run now!】ボタンをクリックすることでjobが実行され、粒子ピッキングに用いる画像のリストが表示されます。
使用する、電子顕微鏡画像を【pick】ボタンを押して選択し、画像を表示した後、粒子を選択します。このマニュアルピッキング作業では、合計でおおよそ1000個の粒子を選択してください。左クリックで選択、中クリックで選択された粒子の削除、右クリックでメニュー(下図)が表示され、座標の保存ができます。【Continue now】ボタンをクリックし、Finished jobsからManualPick/job004を選択することによって、いつでも、メニューから保存した座標のSTARファイルに戻ることができます。
3.5 Particle extraction(粒子の抽出)
粒子をピックしたいすべての顕微鏡写真の座標ファイルを使って、対応する粒子を抽出し、必要なすべてのメタデータを【Particle extraction】jobタイプで取得することができます。【Particle extraction】 jobタイプの【I/O】タブで以下の設定をします。
● Micrograph STAR file: CtfFind/job003/micrographs_ctf.star
(ファイルの選択には【Browse】ボタンが使用できます。CTF補正の情報が入ったファイルを選択します。)
● Coordinate-file suffix: ManulPick/job004/coords_suffix_manualpick.star
(ファイルの選択には【Broswe】ボタンが使用できます。)
● OR re-extract refined particles? No
(これはRELION-2.0の新しいオプションです。STARファイルの粒子のみを再抽出するために、【2D classification】、【3D classification】、【3D auto-refine】 jobから_data.starファイルが使用できます。例えば、抽出時に粒子を前もって縮小していた場合、初めの分類後は元の大きさの粒子で精密化を実行したいときにも役に立ちます。RELION-1.4では面倒で、STARファイルの手動編集が必要でした。RELION-2.0ではすべてがこのオプションで自動でできます。)
● Manually set pixel size? No
(CTF情報が含まれていない顕微鏡写真STARファイルを入力する時のみ必要です。)
【extract】タブで、実際の粒子抽出のための値を以下のように設定します。
● Particle box size (pix): 100
(これは常に偶数にすべきです!これが実際の大きさです。MRCのフォーマットはフーリエ空間が偶数にしか対応していません。)
● Invert contrast? Yes
(これは黒を白に反転します。)
● Normalize particles? Yes
(常に正規化します。)
● Diameter background circle(pix): 60
(粒子はバックグラウンド領域のすべてのピクセルでの平均値がゼロに、標準偏差が1になるように正規化されます。バックグラウンド領域とは、(縮尺変更前に)ピクセル中の指定された円の外側の部分すべてのピクセルと決められています。負の値を設定すると、粒子ボックスサイズの75%の値が使用されます。)
● Stddev for white dust removal: -1 ● Stddev for black dust removal: -1
(データを実際に見る場合、異常に白や黒よりの値のピクセルのみ除去されます。よく使うのは、stdevの値として5程度です。このデータセットでは異常な値のピクセルはありませんので、補正せずに、初期値の-1のままです。(すなわち、何もしません))
● Rescale particles? No
(縮小した粒子は計算速度が速くなります。そのため、処理速度を上げるために初期段階ではよく粒子を縮小します。初期の適切な粒子の分類の計算速度を上げるために使います。再構成がナイキスト周波数に近づくと、縮小せずに粒子を再抽出します。上記のように、RELION-1.4よりもRELION-2.0がとても速いです。)
全ての自動検出された粒子を抽出するために後にも使用するものと同じjobタイプを使用するので、自動抽出と区別するためにmanualpickのようなエイリアスをjobにつけることをお勧めします。【Helix】タブは無視し、一つのMPIプロセスを使い、実行してください。
粒子はExtract/job005/Micrographs/という新しいディレクトリの中のMRCスタック(RELIONではいつも.mrcs拡張子をもつ)の中に抽出されます。【Display:】ボタンからout: particles.starに格納された抽出粒子を可視化することによって、すべてうまく進んでいるかの確認をすぐにすることはとても良い考えです。開いたウィンドウから【Disiplay!】ボタンを押して、実際に表示させると、次のようなディスプレイが現れます。
画面上で右クリックでメニュを選択すると、すべての粒子の選択(Invert selection)や、すべての選択された粒子の(位置が合わせられてはいない、そのままの)平均の計算(Show average of selection)などができます。
3.6 Making templates for auto-picking(自動検出のためのテンプレート作成)
次に、全顕微鏡写真の自動検出のためのテンプレートを計算するために、jobタイプ【2D classification】を使用します。 【I/O】タブ上で(【Browse】ボタンを使って)Extract / job005 / particles.starファイルを選択します。
【CTF】タブ上で以下の操作を行ってください。
●Do CTF-correction? Yes
(ベイズフレームワークの中で、全ての位相強度補正を行います。)
●Have data been phase-flipped? No
(このオプションはRELION外でデータを事前処理した場合にのみ有効です。)
●Ignore CTFs until first peak? No
(振幅補正によって低解像度成分が誤って強く与えられ、全ての粒子が非常に少ないぼけたクラスに分類されるような場合には、このオプションが役立ちます。)
【Optimisation】タブの設定:
●Number of classes: 10
(クライオ電子顕微鏡(cryo-EM)では、少なくともクラス当たり約100個の粒子を使用するのが望ましいです。ネガティブ染色法の場合、1クラスで20~50程のより少ない粒子を使用します。ある程度の分解能を得るには、フーリエ空間が重なり合うことが望ましいため、多くの粒子が必要です。)
●Number of iterations: 25
(これはほとんど変えた事はほとんどありません。)
●Regularisation parameter T: 2
(Tの定義については[10]を参照してください。Tが大きいほどシグナルに重きを置くことになり、結果として高分解能側が強調されます。クライオ電子顕微鏡(cryo-EM)において、2次元分類ではT=2-3が、3次元分類ではT=3-4の値が一般的に使用されます。ネガティブ染色の場合、これよりもより小さい値の方が良い場合もあります。一般的にクラス平均が非常にノイズが多く現れた場合は、一旦Tの値を減らしてください(結果として、多くの場合ローパスフィルタがかかったようになります)。もし、クラス平均の分解能が低すぎる場合はTの値を増やしてください。重要なことは、高分解能側のノイズのオーバーフィッティング(過学習)してしまっていることに気がつくことです。)
●Mask diameter (A): 200
(このマスクは全ての2次元クラス平均に適用されます。また、粒子画像の溶媒ノイズや隅にあるような隣接した粒子を除去するのに使われます。ノイズが大きい溶媒と隣り合った粒子が位置合わせの際に干渉してしまうかもしれないため、直径は小さく保ちたいでしょうが、他方では、クラス平均からシグナルを切り捨てたくないため、粒子の大きさよりも直径が大きいか確かめる必要があります。)
●Mask individual particles with zeros? Yes
(Noの場合は、乱数で埋めます。大きな粒子で高分解能での処理が可能な場合には、こちらのほうが良い結果を与える場合もあります。小さな粒子では0のほうがよいでしょう。)
●Limit resolution E-step to (A): -1
(正の値を与えた場合、この値を超える周波数を位置合わせの際に利用しません。これはノイズによる過学習を防ぐのにも役立ちます。ここではそれは必要ではありませんが、10-15Aで設定されている可能性があります。難しい分類、すなわち非常にノイズの多いデータの場合は、分解能を制限することによって問題が解決することが多いです。)
【Sampling】タブでは初期値を変更することは、ほとんどありません。
●Perform image alignment? Yes
(粒子毎の位置合わせを実行するかどうかを指定します。)
●In-plane angular sampling 5
(面内の回転角度を指定します。単位はdeg.です。粒子の大きさに依存します。)
●Offset search range (pix) 5 ●Offset search step (pix) 1
(粒子の位置合わせの平行移動の最大範囲と刻み巾を示しています。) いくつかの正20面体ウイルスでは、より細かな角度ごとでのサンプリングが必要かもしれませんが、ほとんどの場合は5°ごとの角度サンプリングで十分です。その場合、最初に5°ごとのサンプリングを25回反復実行し、2°ごとのサンプリングを、さらに5回反復して(【Continue now】ボタンを使って)同じ実行を続けます(Number of iterations: 30 【Optimisation】タブで設定)。今回のデータセットの場合、この操作は全く必要ありません。前に何らかの形で失敗したjobを、【Continue now】ボタンを使用して再開することもできます。この場合、どのパラメータも変更する必要は無いでしょう。【2D classification】、【3D classification】、または【3Dauto-refine job】を続行するには、常に【I / O】タブの【Continue from here:】に_optimiser.starファイルを指定する必要があります。
【Helix】タブは無視し、【Compute】タブで以下のように設定を行います。
●Combine iterations through disc? No
(この方法で、全てのMPIノードは、各反復の終わりにネットワークを介してデータを結合します。ネットワークがボトルネックであるか何らかの原因で問題が発生している場合は、このオプションをNoに設定できます。その場合、すべてのMPIノードはデータをディスクに書き込み / 読み出します。)
●Use parallel disc I/O? Yes
(この場合、すべてのMPIスレーブは自分の粒子をディスクから読み込みます。 高速(並列)ファイルシステムを使用している場合は、このオプションを使用してください。 並列ではないファイルシステムは、複数のMPIノードからの並行したアクセスを処理できない場合があることに注意してください。そのような場合、このオプションをNoに設定するべきです。この時、マスターMPIノードだけが粒子を読み込み、ネットワーク経由でMPIスレーブにデータを送信します。)
●Number of pooled particles: 3
(粒子はMPIスレーブによって個々のバッチ処理されます。各バッチの中で、粒子画像のスタッは、ディスクアクセス時間を改善するために1回だけ開閉されます。1つのバッチのすべての粒子画像は一緒にメモリに読み込まれます。これらのバッチサイズは、少なくともスレッド毎に1つの粒子が読み込まれます。nr_pooled_particlesパラメータは、スレッドごとに読み込まれる粒子の数を制御します。3に設定され、1つが8つのスレッドを使用する場合、3x8 = 24個の粒子のバッチが一緒に読み込まれます。これにより、ディスクアクセス、特にディスクアクセスのメタデータ処理が問題となるシステムでのパフォーマンスが向上する可能性があります。わずかなRAM(ランダム・アクセス・メモリ)の使用量の増加をもたらします。)
●Pre-read all particles into RAM? Yes
(Yesを選択すると、すべての粒子画像がコンピュータのメモリに読み込まれ、ディスクアクセスが遅いシステムでの計算が大幅に高速化されます。ただし、使用可能なRAMの量には注意が必要です。粒子は倍精度で読み込まれるため、N個の粒子をRAMに読み込むには(N *box size*box size* 8 /(1024 * 1024 * 1024))ギガバイトかかることになります。並列ディスクI / Oが「Yes」で選択されている場合、すべてのMPIスレーブは粒子すべて読み込みます。並列ディスクI / Oが「No」で選択されている場合、マスタのみがすべての粒子をRAMに読み込み、精密化の反復中にMPIスレーブにそれらの粒子を送信します。)
●Use GPU acceleration? Yes
(これらに合ったGPUをお持ちの場合、このjobは高速化されます。)
●Which GPUs to use: 0
(これを空のままにすると、プログラムは使用するGPUを特定しようと試みます。0または1を使ってGPU IDを入力してください。複数のMPIプロセッサを使用している場合、指定したGPUで各MPIプロセスを実行できます。異なるMPIプロセスのGPU IDは、コロンで区切られます。 0:1:0:1はGPU 0でMPIプロセス0と2を実行し、MPIプロセス1と3はGPU 1で実行されます。)
【Running】タブで、Number of MPI processors(MPIプロセスの数)とNumber of threads(スレッドの数)を指定します。要求されたCPUまたはコアの総数は、2つの値の積になります。 スレッドは、より効率的なRAM使用に関して利点がありますが、MPIの並列化はスレッドよりも優れています。多くの場合、【3D classification】と【3D auto-refine】jobでは、各(マルチコアの)計算ノードで使用可能なRAMを共有するために、多くのスレッドを使用することが必要になります。【2D claasification】(2D分類)ではメモリを消費しないので、スレッドがあまり必要ない場合があります。しかし、MPIプロセス間通信(拡張のボトルネック)が、多くのスレッドを実行する場合に比べて、速度を制限する場合もあります。さまざまなクラスタによって異なりますので、これらのパラメータを使用して、それぞれのコンピュータのセットアップによって最適なパフォーマンスを得る必要があります。
多くの【2D classification】jobを実行するので、例えばmanualpickのような意味のあるエイリアスを使用することが賢いやり方です。GPUアクセラレータまたは(合理的なサイズの)クラスタを使用している場合、このジョブは数分で終了することでしょう。そうではなくて、コア数が少ないデスクトップの場合には、15〜20分かかります。
計算が終了した後は、[Display:]ボタン上でrun_it025_model.starを選択し、得られたクラス平均を見ることができます。ポップアップウィンドウでは、rlnClassDistribution(クラス粒子の個数)(【reverse order】をチェックして、昇順の代わりに降順の並び替え)、またはrlnAccuracyRotationsに基づくなど特定の順序でクラス平均を調べ、選択してみることができます。
この結果を実際に確かめたものを以下に示します。いままでの処理が正しく出来ているかを確認してください。
3.7 Selecting templates for auto-picking (自動検出のためのテンプレート選択)
RELION-1.4と違い、RELION-2.0では【Display:】ボタンで適切なクラスを選択することができません。これは[Subset selection]jobタイプ上で行われます。Select classes from model.starと書かれた行の横にある【Browse】ボタンを利用して、【I/O】タブ上のClass2D/manualpick/run_it025_model_.starファイルを選択してください。
※ これは、パイプライン処理を全てに与えるためです。将来、この選択も自動化されることが予定されています。
【Class opitions】タブ上で下記のように入力してください。
●Re-center the class averages?: Yes
これは、relion-2.0の新しいオプションで、2次元クラス平均の自動センタリングが可能です。画像のセンタリングはその重心を基準としており、粒子の白(黒ではない)の部分をもとに計算されます。センタリングされたテンプレートが自動検出中に必要となる一方で、2D classification実行時ではクラス平均はセンタリングされないかもしれないので、再センタリングがしばしば必要となります。
●Regroup the particles? No
これは、それぞれの電子顕微鏡写真にある粒子が非常に少ない場合に便利なオプションです。そのような場合、スペクトルノイズ強度とスケールファクターの推定が不安定になることがあります。デフォルトでは、スケールファクターの推定は顕微鏡写真ごとにそれぞれ計算されます。このオプションは計算実行時、複数の顕微鏡写真の粒子を一緒に計算してグループ化することが可能です。RELIONは、クラス分類や自動精密化実行の際にグループがとても小さくなっている場合に警告を出します。
templates4autopickのようなエイリアスはとてもいいアイディアです。
rlnClassDistributionカラムに基づいたクラス平均を再び求める可能性があります。代表となりそうな、見た目が異なる粒子のクラス平均を4〜6つ選択してください。見た目が似ているものを繰り返し選択したり、よくないクラス平均を含めたりしないでください。選択するにはクラス平均上で左クリックしてください。右クリックメニューからSave selected classes オプションを使うことで、選択したクラス平均を保存できます。
3.8 Auto-picking(自動検出)
【Auto-picking】jobタイプでは、テンプレートとして選択された2次元クラス平均を使います。 しかし、全ての顕微鏡写真で自動検出を実行する前に、【autopicking】タブ上で、【Picking threshold】、【Minimum inter-particle distance】、【Maximum stddev noise】の3つの主なパラメーターを最適化する必要があります。
そのため、まずはテストとして、選択された数枚の顕微鏡写真に対して行います。そのため、Subset selection】jobタイプを利用して、Auto-Pickingを適用する数枚の顕微鏡写真を選択し、この2〜3枚の顕微鏡写真を含むSTARファイルを作成します。
【Subset selction】jobタイプで行うには2つの方法があります。
(1) Auto-Pickingテスト用顕微鏡画像選択方法その1:"OR select from picked coords:"を使う
OR select from picked coords: の行からManualPick/job004/coords_suffix_manualpick.starを選択。
パラメータを入力後、【Run now!】をクリックし、顕微鏡画像の選択ボックスを表示させます。
この状態で、Auto-Pickingのパラメータ調整テストに用いたい画像を選びます。初期状態では、すべての顕微鏡画像にチェックが入っている状態ですので、"Invert selection"を用いると、一度にすべての画像のチェックを外せるので便利です。使用する画像を選び終わったら、"Save selection"をクリックし、STARファイルを生成します。
(2)Auto-Pickingテスト用顕微鏡画像選択方法その2:"OR select from micrographs.star:"を使う
もう1つの方法は、[Subset selection]jobタイプの【I/O】タブからOR select from micrographs.star: の行からCtfFind/job003/micrographs/ctf.starを選択します。
インプット情報を入力後、【Run now!】をクリックすると、ディスプレイ設定がポップアップしますので、適当な値を入力し、表示させます。表示させた画像から、左クリックで顕微鏡写真を選択し、右クリックメニューからSave STAR with selected imagesを選択して下さい。
どちらの方法を選んでも結構ですが、Job名へ"2mics4autopick"のようなエイリアスをつけたほうがよいかもしれません。これは、前項でのSubset selectionと区別をつけるためです。
次に、上述の方法で作成した2〜3枚の電子顕微鏡画像を含む、STARファイルに対してAuto-pickingを行っていきます。 【Auto-picking】jobタイプの【I/O】タブ上で以下のように設定します。
● Input micrographs for autopick: Select/2mics4autopick/micrographs.star ● References: Select/templates4autopick/class_averages.star ● Pixel size in microgfraphs (A): -1
(入力STARファイルがCtfFind jobからピクセルサイズ情報を得ていますので無視されます)
【Refernces】タブでは以下のように設定します
● Lowpass filter references (A): 20
("Einstein-from-noise"のアーティファクトを避けるために、データから得ようとしている最終的な分解能よりも大幅に低いローパスフィルターを使用することは非常に重要です)
● Highpass filter (A): -1
(たとえば200などの正の値を渡した場合、リファレンスは顕微鏡写真はautopickingよりも高いハイパス・フィルターが適用されます。これは強いグレースケール勾配(氷の厚さなどに依存)が顕微鏡写真にかかっているときに役立ちます)
● Pixel size in references (A): -1
(負の値を渡した場合、入力した顕微鏡写真と同じスケールであると認識します。そうでない場合、例えば粒子を抜き出す上でのリファレンスを作るために使用される粒子をリスケールする必要があるので、正しいピクセルサイズの正の値をここのリファレンスに追加します。)
● Mask diameter (A): 200
(【2D classification】 jobタイプのときに使った値と同じ値が使えます)
● Angular sampling (deg): 5
(この値はどんな場合でもほとんど正常に動くと思われます)
● References have inverted contrast? Yes
(顕微鏡写真の中にある粒子は黒い(CTF補正がかかっていないため)ので、使用するリファレンスは白を利用します)
● Are References CTF corrected? Yes
(2Dクラス平均を行った際にCTF補正をしたため)
● Ignore CTFs until first peak: No
(【2D classification】jobでもリファレンス作製のために使用した場合のみこのオプションを使って下さい)
【autopicking】タブでは以下のように設定します
●Picking threshold: 0.8
(主要調整パラメータ1:これはpeak-searchアルゴリズムのためのFOMマップ中のしきい値です。この値よりも低いFOMの粒子は検出されません)
● Minimum inter-particle distance (A): 200
(主張調整パラメータ2:これは、隣接する2つの粒子間の最大許容距離です。反復的クラスタリングアルゴリズムはこの距離よりも互いに近い粒子を取り除きます。これの効果的な値は粒子直径の50%から60%の範囲であることが多いです)
● Maximum stddev noise: 1.5
(主張調整パラメータ3:これは炭素領域や非常に汚染されてる特徴が見て取れる領域のピッキングを防止するのに有効です。標準化された顕微鏡写真のバックグラウンド標準偏差がこの値より高い領域のピークは無視します。効果的な値は1.0から1.2の範囲であることが多いです。バックグラウンド標準偏差が高いため、ピークを除去するためにこの機能をオフにするには値を-1にして下さい。)
● Write FOM maps? Yes
(下の説明参照)
● Read FOM maps? No
(下の説明参照)
● Shrink factor: 0
(これを0に設定することで、自動ピッキングアルゴリズムは顕微鏡写真をリファレンスにおいてのローパスフィルターの解像度にまで縮小します。これはチュートリアルを手軽にできるように、実行をとても速く、要求メモリも軽くしてくれます。0から1の間の値は顕微鏡写真のサイズの画分の結果になります。これはshrink=1(縮小しない)にするよりもいくらか正確さの低い検出になるということに注意して下さい。これの新要素についての詳細な説明は次のサブセクションでします)
● Use GPU acceleration? Yes
(適切なGPUを備えている場合のみ使って下さい)
● Which GPUs to use:
(これを空白のままにした場合、このプログラムはどのGPUを使えばよいのか判断しようとします。しかし、もし使いたいGPUのIDが例えば0や1などと、はっきりと分かる場合は、この項目に入力して下さい。複数のMPI-processorsを使う場合(そうでない場合も)は、それぞれのMPI processを特定のGPUで動かすことができます。それぞれのMPI processesに割り振るGPU IDは0:1:0:1など、コロンで分けられています。0:1:0:1とはMPI processの0と2はGPU 0上を、MPI processの1と3はGPU 1上で動作しているという意味です。
【Helix】タブは無視して下さい。
【Running】タブでひとつのMPI processorを使って動かして下さい。おそらくoptimise_paramsのようなエイリアスが有効かもしれません。チュートリアルでは2000×2000の顕微鏡写真を使っているので、1枚の顕微鏡写真につき、計算に5分ほどかかります。より一般的な4000×4000の顕微鏡写真を使った場合は、一般的に顕微鏡写真とリファレンスごとにだいたい5分ほどかかります。GPU-accelerationを使った場合は。10秒以内に動作完了します。そのため、FOMマップの書き込みと読み込みがあまり関係なくなります。
この計算の高負荷な部分は、a probability-based figure-of-merit(それぞれの回転したリファレンスと顕微鏡写真の中の全ての場所での相互相関関数に関連した値)の計算をすることです。その後、上で設定したしきい値と距離の最小値を利用したより高速なpeak-detectionアルゴリズムが実行されます。これらのパラメータは最適化する必要があるため、プログラムは【autopicking】タブで指定されているようにいわゆるFOMマップを作成します。これらはリファレンスごとに2つの大きなサイズ(顕微鏡写真のサイズ)のファイルになります。ハードディスク動作中のI/O障害を避けるために、FOMマップに書き出すときに自動検出プログラムは順次に動作するようにできます(従って上記で示した様に、一つのMPI processorで実行するべきです。)
FOMマップをディスクに書き出せば、より高速に検出するパラメータになるように最適化するために使用されます。最初に、動作させ終えたばかりのjobで【Display:】ボタンから coords_sufix_autopick オプションを使って、現在の設定で自動検出された粒子を調べます。ディスプレイウィンドウは、最後に実行した【Manual picking】jobのパラメーター(サイズやシグマコントラスト、カラーなど)を利用することに注意しておいて下さい。実際に【Manual picking】jobタイプでこれらのパラメーターを変更でき、左上の jobs メニューから Save job settings オプションで設定を保存できます。
FOMの状態を表示するために、【Colors】タブで次のように設定したあとに、これを行って下さい。
● Blue<>red color particles? Yes ● MetaDataLabel for color: rlnAutopickFigureOfMerit
(選択出来ないため、書き込む必要があります。)
● STAR file with color label:
(この部分は空にしておいて下さい。)
● Blue value: 1 ● Red value: 0
条件を入力した後に、この条件設定を保存します。保存するには、左上の【Jobs】の【Save job settings】を選びます。
その後、【AutoPicking】のジョブへと戻ります。今まで行っていたJobを読み出すために、【Finished jobs】の中から、【AutoPick/optimise_params】を選択します。【Auto-picking】が選ばれていることを確認した後に、【Display:】部分で、【out:coords_suffix_autopick.star】を選択し、自動ピッキング結果の電子顕微鏡画像を読みだしてみます。
顕微鏡画像を表示させると、少しわかりづらいかもしれませんが、粒子を選択している円の色が異なります。これは、FOM値に応じて色分けされており、青い円になるほどFOM値が高く、赤い円になるほどFOM値が低くなります。わかりづらいときは、上述のBlue value/Red valueを狭い範囲に設定して下さい(例えば、0.8/0.5)
この結果から、粒子をより多くか、少なく検出するか(すなわち、しきい値を小さくするか大きくするか)と、近くの粒子を同一とするか異なるものとするか(すなわち、粒子間の最小距離値の設定)を決定します。次のステップに進む間に、両方の顕微鏡写真の表示ウィンドウを開いたままにすることができます。
[Finished jobs]から、AutoPick/optimise_params jobを選択して下さい。そして【autopicking】タブ上でパラメーターを変更して下さい。次のように変更して下さい
● Write FOM maps? No ● Read FOM maps? Yes
【Continue now】ボタンをクリックすることで実行した時、全てのFOMの再計算の代わりに、以前書きだされたFOMマップをディスクから再読み込みします。新しい座標のその後の計算は、数秒で実行されます。
この後に、新しい座標セットを読み込むために、顕微鏡写真の表示ウィンドウ上で右クリックメニューから Reload coordinates を選択して下さい。この動作を繰り返して、autopickingのパラメータを調節していきます。
3つのパラメータをすべて使いこなして、検出結果をどのように変更するかを確認してください。
自動ピッキングに用いるパラメータが決まった後は、すべての電子顕微鏡画像に対してこの処理を行います。そのために、Auto-Pickingの読み込み画像を変更します。パラメータチューン用のジョブとは異なるジョブを作るために、ジョブタイプの【Auto-picking】をクリックし、あらたなジョブを作成します。その後に、【I/O】タブ上で、入力した顕微鏡写真のSTARファイルをすべての画像ファイルへと変更します。これは、【CTF estimation】jobで作られたファイル、【CtfFind/job003/micrographs_ctf.star】を指定してください。
【autopicking】タブで、先ほど求めたパラメータで設定を行います。ここでは、例として下記のようなパラメータを用います。
● Picking threshold: 0.4 ● Minimum inter-particle distance (A): 110 ● Maximum stddev noise: 1.1 ● Write FOM maps? No ● Read FOM maps? No
ここで、jobは並列に(FOMマップが書き出せされてないのであれば)実行されるかもしれません。【Running】タブで、自動検出を実行するコアの数を指定します。MPIプロセッサの最大有効数は、入力したSTARファイルの顕微鏡写真の数となります。
FOMマップを読み込み書き出しをするかどうかによって、【Continue now】ボタンの動作の仕方に重要な違いがあることに注意して下さい。FOMマップを読み込みまたは書き出しをする場合には、【Continue now】ボタンをクリックすることで、プログラムは(一般的には少数の)全ての入力した顕微鏡写真を再度検出します。しかし、FOMマップの読み込みまたは書き込みをしない場合、すなわち、自動検出を全ての顕微鏡写真で行う2回目のjobでは、【Continue now】ボタンをクリックすることで、まだ自動検出を行っていない顕微鏡写真のみ再検出します。これはスケジュール化されたjobの反復にて有効です。たとえば、顕微鏡検査セッションの間の高速データ処理などです。また、詳細は2.3のセクションを見て下さい。もし再検出を全ての顕微鏡写真について、新しい値のセットを用いてしたい場合は(仕上がっていない顕微鏡写真だけ動作させる代わりとして)、【Run now!】ボタンの代わりに、左にあるjobタイプブラウザ上で【Auto-picking】jobタイプをクリックして下さい。そのとき、新しい出力ディレクトリが作成されます。
【Display:】ボタンからcoords_suffix_autopick オプションをクリックすることで、結果を再確認することができます。手動で全ての顕微鏡写真を調べて誤検知を取り除くのを好む方もいます。たとえば、顕微鏡写真上の炭素端や高コントラストの人工物はしばしば粒子と誤認されます。【Display:】ボタンからポップアップウィンドウを使うことで、それぞれの顕微鏡写真においてこれを実行できます。マウスの中ボタンを使って粒子を取り除いて下さい。中ボタンを押しっぱなしにすることで、広い範囲の誤認粒子を複数取り除けます。忘れずに右ボタンメニューから新しい座標を保存して下さい。
ひとたび全部の結果を満たしたすと、ディスクスペースを確保するために、最初のステップで書き出したFOMマップを削除したくなるかもしれません。手軽に行うために【Job actions】ボタンから、Gentle clean オプションを使うことができます。
ひとたび座標全体セットを満たしたら、以前のデータを全て保ちつつ、【Partcle extraction】jobを再実行する必要がありますが、【Input coordinates】を自動検出で新しく生成されたものに変えて下さい。これは下記のよりよい精密化のために使われる初期単粒子データセットを作成します。おそらく、allmics_autopicked のようなエイリアスが有意義です。
3.8.1 The shrink parameter(縮小値)
より速い処理を可能にするために、RELIOM-2.0では、コマンドライン引数 --shrink を通して顕微鏡写真をフィルタリングするオプションを実装しています。最もシンプルな使い方は --shrink 0 と使うことです。しかし、これはもっと制御して使うことができます。その場合は、次の方法に従って動作して下さい。
--shrink の初期値は1.0ですが、これは効果がなく、顕微鏡写真に全くフィルターをかけません。これは以前のバージョンのRELIONの自動検出と同一の動作をします
● --shrink val = 0 では、テンプレートリファレンスのローパスと同じように、--lowpass 指定されたローパスフィルタをかけた顕微鏡写真となります。これは単粒子解析(らせん状検出のためではなく)のために使うことをお勧めします。
● --shrink 0 < val ≤ 1 では、画像サイズが、size= val ·micrograph size にした結果になります。すなわち、valとは顕微鏡写真のオリジナルサイズに適用させたスケールファクターです。
● --shrink val > 1 では、size = val − (val mod 2)、すなわち、valより小さい最小の偶数の整数値になります。これは顕微鏡写真に特定の(偶数の)整数サイズ与える方法です。
新しいサイズが--lowpassよりも小さい場合、これは要求された(あるいは初期設定の)分解能を超えて、分解能を制限することになるので、致命的ではありませんが警告をならす結果となります。RELIONの自動検出は多くのFFTを実行するため、顕微鏡写真のサイズもFFT計算の大きな落とし穴を避けるために自動的に調整されるようになりました。これらに関する注釈とどのようにパラメータが上書きされるかは、ユーザーが実行する各自動検出の最初の出力に表示されます。
3.9 Particle sorting(粒子ソート)
RELIONには、各粒子とそれらの調節されたCTF補正リファレンスとの間の差異に基づいて粒子を素早く並べ替える機能もあります。ソートプログラムは【Particle sorting】jobタイプからアクセスすることができ、それぞれの粒子でリファレンスが利用できるときであれば、いつでも動かせます。たとえば、【Auto-picking】、【2D classification】、【3D classificationo】、【3D auto-refaine】の後などです。その使用方法を示すために、自動検出の後に実行します。
【I/O】タブでは以下のように設定してください。
● Input particles to be sorted: Extract/allmics autopicked/particles.star
(このファイルは自動検出後の抽出プログラムによって生成されます。【2D classification】、【3D classification】または、【3D auto-refine】jobの場合は、最後の反復での_data.starを入力するべきです。)
● Are these from an Extract job? Yes
(入力粒子が 【2D classification】、【3D classification】、【3D auto-refine】job由来の場合、これをNoにセットしてください。)
● Autopicking references: Select/templates4autopick/class averages.star
(抽出された粒子のための座標を与える自動検出jobに使われたリファレンスを入力してください。)
【CTF】タブ上では以下のように設定してください。
● Are References CTF corrected? Yes ● Ignore CTFs until first peak? No
このプログラムはとても速く(分単位で)動作します。複数のMPI processorsを動かす必要がないほどです。おそらくエイリアスとしてafter_sortingを使えると思います。このjobは出力ディレクトリに particles_sort.star と呼ばれる入力ファイルと同じファイルを生成しますが、rlnPartileSelectZScore と呼ばれるカラムの追加があります。【Subset selection】jobタイプの 【OR select from particles.star】 とある行で、このファイルを選択することが出来ます。ポップアップ表示ウィンドウ内にて、その新しいカラムに基づいて粒子を可視化できます。ディスプレイの上に良い粒子、下に良くない粒子をもつ強い傾向が見えてきます。下から上にスクロールして、下のすべての粒子を破棄する必要があるポイントを決定でき、右マウスメニューを使用して Select all above および、または1つずつ粒子を選択することができます。
右クリックメニューを使って、出力STARファイルに忘れずに保存しましょう
4. Reference-free 2D class averaging (参照無2次元クラス平均)
前書き
良くない粒子を取り除くために、大抵は参照なし2Dクラス平均を使います。
前項で述べた粒子抽出ステップ(例えば、自動検出結果の手動管理や抽出された粒子の分類)で良い粒子を含もうとしますが、ほとんどの場合、良くない粒子がデータの中にまだ存在しています。
良くない粒子は一緒にうまく平均化しないので、大抵は比較的小さいクラスになりやすく、不適切な2Dクラス平均を算出します。これらを取り除くことで効率的にデータをきれいにします。
4.1 Running the jobs (動作の実行)
このチュートリアルでは動作タイプを【2D classificiation(2次元分類)】にセットし、その中の【I/O】タブは次の通りにセットします。その他のオプションは、前述の自動検出のテンプレートを作成した時と同じです。
Input images STAR file: Select/after_sorting/particle.star
現在約10,000個の粒子が存在するので、もっと多くのクラスを利用することができます。従って、【Optimisation(最適化)】タブを次の通りにセットします。
Number of class: 100
この動作は以前に述べた動作のものよりはるかに負担がかかる(アルゴリズムは粒子の数とクラスの数に比例してスケーリングされます)ので、この動作を並列処理することをお勧めします。1つもしくは複数のGPUがある場合、この動作を1時間半分程度で実行できます。aftersortのようなエイリアスを使用できます。
B動作が終了したら、_model.staファイルを選択して【Subset select】を起動することができます。class2d_aftersortのようなエイリアスは意味があります。ここで、すべての見栄えの良いクラスをクリックして選択します。(もしくは右クリックメニューオプション【Select all classes above】を使用する。)この時、自動検出において低いしきい値を使用して、高解像度のノイズが蓄積している場合、低解像度のゴーストのように見える【Einstein-from-noise】クラスに気を付けてください。こうしたクラスの選択は避けて下さい。より良いクラスがすべて選択されたら、マウス右クリックメニューオプションから選択したものを保存します。
この手順は、複数回繰り返すことがあるので、覚えておきましょう。また、2D classficationのあとのデータに残っている外れ画像を識別するために、ソート(並べ替え)アルゴリズムを再実行できることに注意してください。
※ 【2D classification】、【3D classifiction】は共に、最初にランダムにクラス分けをするため、外れ画像が多く混じっているとうまく分類できない可能性があります。そのため、粒子を選び直して、改めて分類するとよりよい分類が可能になります。
【Number of class: 100】とした場合の結果
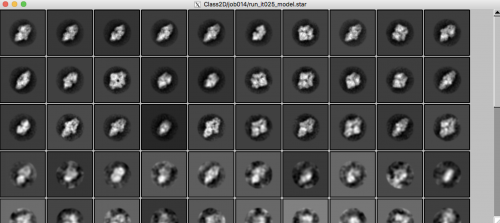
4.2 Analysing the results in more detail(より詳細な結果の分析)
ここではRELIONへの興味が深い人向けの、より詳細な情報を含んでいます。 お急ぎの方はこのセクションをスキップしても構いません。
RELION は 2D(3D)classification を繰り返すごとに、ファイルに書き出します。 2D クラス平均算出の最終反復時では、以下のファイルが作成されます。
● Class2D/allparts_run1/run_it025_classes.mrcs
これは、クラス平均結果をMRCスタックとして保存しています。これらはメインGUIのDisplay: ボタンから _model.star を選択したときに表示される画像です。RELION は GUIから選択した場合、完全なCTF補正がかかることに注意して下さい。そのためクラス平均はおそらく黒い背景に対して白いものになります。もしデータが良ければ、その画像はローパスフィルターをかけた原子モデルにとてもそっくりな投影になります。 2D クラス平均の質は3Dマップがうまくいくかどうかの指標になります。我々はタンパク質ドメインの投影を用いた内部構造と、理想的には平らになっているであろう粒子周りの溶媒領域を期待したいところです。
溶媒領域の放射状に伸びた筋は典型的な過剰適合の兆候です。もしこれがおきたときは、2D classification アルゴリズムのE-step内で分解能に制限をかけてみて下さい。
●Class2D/allparts_run1/run_it025_model.star
これは実際のクラス平均に沿って精密されたモデルパラメーター(クラス間の画像の分布、再構成された構造中のシグナル対ノイズ比の球面平均、全てのグループのノイズスペクトラム、など)を含んでいます。
less コマンドを使って、このファイルの中身を見てください。特に、data_model_classesテーブルのそれぞれのクラスの粒子分布を確認して下さい。これとクラス平均を比較すると、低分解能のクラスにある粒子はほとんどない一方で、高分解能のクラスに存在する粒子は多いと分かる。
平均的に少ない粒子を通しての平均化は自然と低いシグナル対ノイズ比になりますので、このときにベイズ推論的な考えを持つことが重要になります。それぞれのクラスにおいて概算されたスペクトラル、シグナル対ノイズ比はdata_model_class_N テーブルに保存されます。同様に、それぞれのグループにおいて概算されたノイズスペクトラムはdata_model_group_Nテーブルに保存されます。data_model_groupsテーブルはそれぞれのグループにて精密されたscale-factor強度を保存します。(平均よりも高い値をもつグループはより強力なシグナルを持っている、平均よりも低い値をもつグループは相対的に低いシグナルである)
これらの値はデフォーカスと相互関係があることが多いだけでなく、汚れのの蓄積や氷の厚さに影響される。
●Class2D/allparts_run1/run_it025_data.star
これはそれぞれの粒子に関わる全てのメタデータを含んでいます。そのメタデータとは、particles.star fileに入力した情報の他に、最適な方向、最適なクラス同定、対数尤度への寄与、などに関する追加情報です。
このファイルは再度新しく洗練されたデータをstarファイル同様の形式のでの入力に使用される。
●Class2D/allparts_run1/run_it025_optimiser.star
これは終了しなかった場合の再起動に必要な精密化過程についての全体的な情報が含まれています。たとえば、もし25反復後でもまだ収束しなかった過程を考える場合(評価のために反復24と25から出したクラス平均を比較できる)、Finished jobs パネルの中で以下の機能を選択できる。I/O タブ上で、Continue from hereから上記のファイルを選択することが出来る。そのときに、Optimisationタブ上で
Number of iterations:40
と設定して下さい。また、Sampling タブで緻密な角度や並列処理をするサンプリング比率を選ぶかもしれません。optimiser.starファイルのもう1つの有用な特徴として、1行目に、これを動かすために与えられた正確なコマンドライン引数がコメントとして含まれています。
●Class2D/allparts_run1/run_it025_sampling.star
これはサンプリング間隔の採用についての情報が含まれています。このファイルもまた再起動に必要なファイルです。
4.3 Making groups
ここではRELIONへの興味が深い人向けの、より詳細な情報を含んでいます。 お急ぎの方はこのセクションをスキップしても構いません。
RELIONは、粒子をグループ化する際に、以下のふたつのことを実行します。ひとつは、平均のノイズのスペクトラム強度(σij^2)の推定と単一数としての強度スケールファクター(τi^2)の推定です。後者は、そのデータの異なる部分の間での、全領域シグナル対ノイズ比(SNRi)の違いを表現しており、例えば、氷の厚さ、デフォーカスまたは汚染等によるものです。
通常、顕微鏡写真上の全ての粒子は別々のグループとして扱っています。この動作は顕微鏡写真ごとに多くの粒子が存在する場合には良いです。しかし、高倍率で扱っている場合、サンプルは非常に希釈されるか、最終的に選択されたものは顕微鏡写真ごとには少数しか粒子が含まれておらず、強度スケールファクター(またノイズスペクトラ)強度の推定は不安定なものとなります。一般的に各グループに少なくとも10〜20の粒子がある方が良いとしていますが、2D,3Dクラス化後、各グループの粒子の初期数はより少なくなるだろうということに注意しておいて下さい。
顕微鏡写真ごとに粒子が殆ど無い場合、複数の顕微鏡写真からの粒子をグループ化することをお薦めします。このことから、RELION 2.0での新しいGUIとして【Subset selection】の中に便利な機能が追加されています。それは【I/O】タブ上で _model.starファイルを選択した時、【Regroup particle?】と表示された時【Yes】と入力し、【Approximate nr of groups : 5】と【Class option】タブで入力することで、すべての粒子を5つのグループに分類できます。(実際の数は入力値によって幾分変化するため、入力上での近似値となる)この方法は以前の最終バージョンのRELIONでは避けられた方法でした。このチュートリアルの顕微鏡写真には十分な粒子が含まれているので、ここではこの手順を使用しません。
RELIONは、他のプログラムで時々使用されるデフォーカスグループとは非常に異なることに注意しておいて下さい。RELIONは常に各粒子に使用されたグループとは無関係に(異方性の)CTF補正がされます。
5. Unsupervised 3D classification (教師無3次元クラス分類)
前書き
全てのデータセットは異成分からなっています。問題は どのくらい寛容でいられるかです。RELIONの3Dマルチリファレンス精密化手順は、強力な教師無し3次元分類へのアプローチを提供しています。しかし、まずは低解像度の単一の合意が得られるモデルが必要です。初期の結晶構造(PDB-ID 3I3E)から生成された低分解能のベータガラクトシダーゼモデルを使用します。これはすでに projectディレクトリの中に 3i3e_lp50A.mrcという名前で入っています。典型的なプロジェクトでは、良い初期モデルがないかもしれません。RELIONは、まだ初期モデル生成を行わないので、別のプログラムで生成する必要があります。
※例えば、chimeraで、PDBを読みだ後、コマンドラインを立ち上げて、50Aのマップをつくるなら
molmap #0 50
※例えば、Eosでは、pdb2mrcを利用して作成する事ができます。
$ pdb2mrc -i pdb -o mrc ....
すでにリファレンスモデルを正しいピクセルとボックスサイズで作っています。もしこの事例に当てはまらないモデルを使う場合は、コマンドラインからrelion_image_handlerというプログラムを使って、ピクセルサイズ(--anpixelと--rescale_angpixというオプションを使用)とボックスサイズ(--new_box)を変更してもかまいません。 この場合、まず、【Import】jobタイプを使用し、直接プロジェクトに初期モデルをインポート出来ます。ファイルを入力したら、3i3a_lp50A.mrc を選択して、the Node typeで3D referenceを選択して下さい。同じ 【Import】jobを区別するために、initial-3drefのようなエイリアスを使います。
5.1 Running the job (ジョブを開始)
教師無し3次元分類は、【3D classification】jobタイプから実行可能です。
【I/O】タブの設定:
● Input images STAR file: Select/class2d_aftersort/particles.star
(選択された良い粒子が記述されたstarファイルを設定します。)
● Reference map: Import/initial_3dref/3i3e_lp50A.mrc
(比較するための参照構造を設定します)
● Reference mask (optional):
(これは空白のままにしておきます。これは、例えば、注目しているリボソーム精密化のために大/小サブユニットマスクを提供するところです。この項目を空のままにしておくと、[Optimisation]タブで指定された粒径の球形マスクが使用されます。 これは分類へのバイアスを最小限にする働きがあります。)
【Reference】タブの設定:
● Ref. map is on absolute greyscale: No
(RELION内の同じデータから再構築されていないマップは、おそらく、正しいグレースケール上にないと考慮しておきましょう。)
● Initial low-pass filter (A): 50
(精密化プロセスにバイアスが導入する可能性があるため、高解像度の初期参照モデルは使用してはいけません。[9]で説明したように、初期マップをできる限りフィルタリングする必要があります。リボソームでは大抵、70Åを使用し、小さい粒子では通常40〜60Åの値を使用します。)
● Symmetry: C1
(このサンプルはD2の対称性があることはわかっていますが、対称性を持たない初期分類を実行することをお勧めします。良くない粒子は適切なものから分離することができ、対称性は再構成されたマップで確認することができます。)
【CTF】タブの設定:
● Do CTF corection? Yes
● Has reference been CTF-correced? Yes
(このモデルはPDBファイルで作成されているため、振幅はCTFの影響を受けていません。これは"CTF修正済み"とみなすことができます。ほとんどの他のEMパッケージから作られたモデルは、適切な低周波振幅補正を実行するパッケージが多くないため、ほとんどの場合CTF補正されていないことが多いです。(いくつかは補正されているかもしれません。その場合、モデルを通してスライスをチェックすることができます。もし黒の背景に対して白く見えるならば、モデルはおそらく適切にCTF修正されたものでしょう。一方で、黒いハローを伴って灰色に見えるのであればそのモデルはおそらく正しくCTF補正されていないでしょう。)これまでRELIONでCTF補正を行ったモデルは、適切に補正されています。)
● Have data been phase flipped? No
(位相補正だけを実施しているかどうかです。)
● Ignore CTFs untill first peak? No
(リファレンスを作成するために使用した[2D classification]jobでもこのオプションを使用した場合にのみ、このオプションを使用します。)
【Optimisation】タブの設定:
● Number of class: 4
(より多くのクラスを使用すると、多様な情報が含まれているデータセットがより多くのサブセットに分割されます。計算コストは、CPU時間と必要なコンピュータメモリの両方の観点から、クラス数に比例して変化します。)
● Number of iterations: 25
(これは一般的には変更する必要はありません。)
● Regularisation parameter T: 4
(Tの定義については[10]を参照してください。クライオEMの、2次元分類では、通常、Tは1~2を設定し、3次元分類では2~4を設定します。ネガティブ染色の場合、少し低い値が効果的な時があります。クラス平均にノイズが見える場合はTの値を低くし、逆にクラス平均の分解能が低すぎる場合はTの値を高くします。主に気をつけなければならない点は、高解像度のノイズの過学習です。あらかじめ計算された結果に2という値を使用しました。おそらく4を設定しても同じようにうまくいくでしょう。)
※ Tの値は、tiと呼ばれるシグナルが大きいと値が大きくなる値を定数倍して使うことになります。つまり、SNRを過大評価するようになり、SRN低い領域まで利用する様になります。実際には、フーリエ空間のボクセル毎に決まりますが、大抵の場合、高分解能の情報まで用いる事を示しています。)
● Mask diameter (A): 200
(前回の[2D classification]のjobタイプと同じ値を使用してください。)
● Mask individual particles with zeros? Yes
(Yes: 0 / No: 乱数)
● Limit resolution E-step to (A): -1
(正の値を指定すると、この値を超える周波数はアラインメントに含まれません。これは過学習を防ぐのにも役立ちます。実際に必要にではありませんが、10~15Aに設定されている可能性があります。)
【Sampling】タブでは、通常何かを変更する必要はありません(正20面体ウイルスのような大型で高い対称性をもつ粒子の場合のみ、典型的には、この時点で3.7度の角度サンプリングを使用します)。
【Helix】タブは無視して下さい。
以前の[2D-classification]の場合と同様に【Compute】タブに記入してください。
ここでも、【Running】タブで、使用するNumber of MPI processors(MPIプロセッサの数)とthreads(スレッド数)を指定できます。[2D classification]のjobタイプで説明したように、3次元分類は2次元分類よりも多くのメモリを必要とするため、より多くのスレッドが使用されることがあります。しかし、この場合、画像はかなり小さく、RAM不足はそれほど大きな問題ではないかもしれません。おそらく、最初の3次元分類を表示するためにfirst_exhaustiveのようなエイリアスを使用でき、徹底的な角度検索を使用しているのではないでしょうか。
精密化された再構成を分析する際には、UCSFキメラなどのしきい値マップだけでなく、スライスでそれらを見ることも非常に便利です。スライスビューでは、未解決の異質性のより良い印象を受けるでしょう。これは、スライスに不鮮明な領域や縞状の領域として表示されます。スライスはまた、溶媒領域の平坦性の良好な印象を確認できます。【Display:】ボタンを使用して、最後の反復からの再構成のいずれかを選択して、RELIONのスライスビューを開きます。
UCSFキメラなどを使用し、表示されたマップを3Dで見ると、マップが精密化の間にわずかに回転する可能性があるので、それらをすべて最良のものに合わせることはしばしば良い考えです。キメラでは、【Tools】 - > 【Volume Data】 - > 【Fit in Map】ツールを使用します。お互いに並んでいる複数のマップを見るには、【Tools】 - > 【Structure Comparison】 - >【Title Structures】ツールと、【Viewing】ウィンドウの【independent】中心回転方法を組み合わせたものが好ましいです。
2次元分類の場合と同様に、【Subset selection】を使用して、1つ以上のクラスに割り当てられた粒子のサブセットを選択することができます。【I/O】タブで、_model.starファイルのサブセットを最後の反復から選択します。表示ウィンドウには、4つの精密化されたモデルの中央スライスが表示されます。最適なクラスを選択し、右クリックメニューを使用して対応する粒子を保存します。class3d_first_exhaustiveのようなエイリアスを使用してください。
4つのクラスの内の1つ
5.2 Analysing the results in more detail (詳細な結果の解析)
ここではRELIONへの興味が深い人向けの、より詳細な情報を含んでいます。 お急ぎの方はこのセクションをスキップしても構いません。
出力ファイルは基本的に2次元分類の実行時と同じです。(実際に2次元,3次元精密時に同じ記法を利用しています)唯一の違いは、以前に出力された2次元クラス平均に関するシングルMRCスタックとは対照的に、各クラスのマップが別々のMRCマップとして保存されることです。例えば、run_it025_class00?.mrcというMRCマップです。
今まで通り、小さいクラスは大きなクラスより精密にローパスフィルターにかけられ、スペクトルのシグナル対ノイズ比(SNR)が_model.starファイルのdata_model_class_Nテーブル(N = 1,,,,K)に保存されます。おそらく今がSTARファイルから任意のタイプのデータを抽出するのに便利な2つの手軽なスクリプトを導入するいい機会です。以下の通りに入力して下さい。
relion_star_printtable Class3D/first_exhaustive/run_it025_model.star data_model_class_1 rlnResolution rlnSsnrMap
上記はdata_model_class_1テーブルから解像度に関する(rlnResolution)コラムとスペクトルのシグナル対ノイズ比に関する(rlnSsnrMap)コラムを画面に出力します。ここでそのファイルを参照して、お好みの言語でプロットできます。もし、使用者のコンピューターにgnplotがインストールされているのであれば、以下の通りに入力して下さい。
relion_star_plottable Class3D/first_exhaustive/run_it025_model.star data_model_class_1 rlnResolution rlnSsnrMap
実行が収束したかどうかを確認するには(上記のように)、以下でも確認できます。
grep _rlnChangesOptimalClasses Class3D/first_exhaustive/run_it???_optimiser.star
これまででお分かりのように、STARファイルはさまざまなタイプの入出力データを扱うのに非常に便利なツールです。得られた結果の分析するための柔軟で強力なツールとなるrelion_star_printtableのようなスクリプトは、だいたいgrepやawkのようなlinuxのシェルコマンドと併用して実行できます。
6 High-resolution 3D refinement(三次元精密化)
前書き
十分に均質なサブセットは一旦選択されると、このサブセットを自動的に高い解像度に精密化するために[3D auto-refine]を使用することができます。この手順は分解能を推定するために、gold-standard Fourier Shell Correlation(FSC)(二つの群に分け、互いに独立して再構成した場合に、どの分解能まで相関があるかにより判断)と言われる判断基準を用います。これにより、過学習を避けることができます[13]。角度の割当の正確さを推定する新しい手順([11])と組み合わせ、精密化が収束するときに自動的に決定されます。そのため、この手順は使用者の入力をほとんど必要としません。すなわち、客観性をのこしたまま、多くのデータセットのための優れたマップが生成がされていると述べられています。もう一つの長所は、一般的にこの手順は必要な実行回数が一回のみであり、最適化のための引数はほとんどありません。
6.1 Running the job (動作の実行)
RELIONで、【3D auto-refine】jobタイプを選択してください。RELIONでの入力引数の大部分は【3D classification】jobタイプと同様のものが残っていますが、一部は利用できません。【Sampling】タブでの配向性のサンプリング間隔は反復の最初の数回のみ使用されており、ここでのアルゴリズムは収束するまで、角度サンプリング間隔が自動的に細かくなっていきます。したがって、8面体または正20面体対称よりも小さい角度で、全ての精密化仮定でで、一般的に大域探索では、初期値の角度である7.5度のサンプルを使用し、局所探索では1.8度のサンプルから行います。より正確な対称の精密化を行う場合のみ、3.7度でのサンプリングを行い、局所探索では0.9度ごとにサンプリングを行います。
RELION-2.0は、【Optimisation】タブでUse solvent-flattend FSCsと呼ばれる新しいオプションがあります。このオプションの設定はRELION-1.4のようにNoで実行します。もしYesで設定した場合、自動精密化は、[Post-processing]jobタイプで使用するものと似ている代表的なFSC曲線で溶媒平滑化補正が使われます。その結果、この動作では【I/O】タブでマスクの提供も必要です。タンパク質が粒子ボックスの中に比較的小さい体積を占有している時、このオプションは非常に役に立ちます。他にも、伸長された分子や、マスクを使用して小さい部分の精密化に注目した時などに役立ちます。このような場合、マスクが使用されていない代表的なFSCは、精密化の分解能が低く見積もられます。今回の精密化では、このオプションはNoのままにしてください。(マスクを提供していないからです)
入力STARファイルとして、Select/class3d_first_exhaustive/particles.starを選択し、【3D classification】実行(例えば、Class3D/first_exhaustive/run_it025_class004.mrc)から初期モデルとして、精密化されたマップのひとつを使用します。あるいは、以前に使用したものと同じ、3i3e_lp50A.mrcモデルを使用します。どちらでもおそらく違いはほとんどありません。さらに、マップ内の高周波成分へ偏るのを防ぐためと、初期マップよりも高い解像度で完全に独立した精密な”gold-standard”を保持するために、初期マップにローパスフィルターを強くかけることが最も良いです。再び50Åを使いましょう。分子が対称に見えるときは、精密化したものに2D symmentryをかけましょう。
MPIのノードは1つのマスター(他のスレーブを管理するだけのもの)と2つのスレーブ(2つの半分のセットに作用するもの)のセットで分かれているので、奇数のMPI処理装置を使用するのが最も効率が良いです。ナイキスト周波数までのすべての周波数が考慮されているため、必要メモリは最終反復でかなり増加します。従って、使っているクラスターノードのコアと同じ数のスレッドで動かしたいデータセットのボックスより大きいサイズで作られたボックスが必要です。もしかするとafter_first_class3dのようなエイリアスが良いかもしれません。
6.2 Analysing the results (結果の分析)
出力ファイルは3D classificationを実行したときのものと大部分が同じです。しかし、全てにおけるプログラムの反復では、2つのrun_it0??_half?_model.starと2つのrun_it0??_half?_class001.mrcファイルを書き出します。それぞれデータの半分が精密化されます。収束した場合にのみ、ひとつのrun_model.starとrun_class001.mrcファイルを書き出します(名前に_it0??がありません)。最終反復では、2つの独立した半分の復元データが一緒に結合されているので、一般的に最終反復で解像度がかなり改良されます。プログラムはすべてのデータでナイキスト周波数を使用しているので、この反復では更に多くのメモリとCPUが必要です。
角度サンプリングでの自動増加が自動精密化手順の様子が重要な側面であることに注意してください。[11]で説明されているシグナル対ノイズの考察が基礎となっており、角度の精度と併進運動の割り当てを評価します。プログラムは必要だと判断したものよりも細かい角度や並進運動サンプリング比は使用しません(結果がよくならないためです)。評価された精度と採用したサンプリング比は現在の解像度評価と一緒にすべて_optimiser.starと_model.starファイルに保存されますが、stdoutファイルからも抽出されるかもしれません。
例えば、以下のように試してみてください。
grep Auto Refine3D/after_first_class3d/run.out
7 Mask creation & Postprocessing(マスク作成&後処理)
前書き
3D自動精密化の実行後、マップを先鋭化する必要があります。その場合も、自動精密化の内部で生み出されるgold standard FSCカーブはマスク化されていないマップのみ使います(Use solvent-flattened FSCsオプションを使っていない場合に限ります)。これは実際の精密化中には、実際の分解能は下回っている(under-estimated)ということを意味しています。なぜなら、溶媒領域のノイズがFSCカーブを低くするからです。RELIONにおけるBファクターでの先鋭化と、マスク化したFSCカーブ[2]を計算するための手順は、"post-processing(後処理)"と呼ばれています。しかし最初には、タンパク質の末端と溶媒領域の開始点を精密化するためのマスクを作る必要があります。これは【Mask Creation】ジョブタイプを使って行います。
7.1 Making a mask (マスク作成)
【I/O】タブ上で3D自動精密化が終わった出力マップ、Refine3D/after_first_class3d/run_class001.mrcを選択してください。【Mask】タブ上で以下のように設定してください。
● Lowpass filter map (A): 15
(15Åのローパスフィルターが滑らかな溶媒マスクのために良い選択と思われます)
● Pixel size (A): 3.54
(この値はローパスフィルターの計算にのみ使われます)
● Initial binarisation threshold: 0.02
(これは例としてタンパク質領域の外側にノイズスポットが完全になく表示するキメラのように、ローパスフィルターのかかったマップの描写でのしきい値となります。適切なスポットを見つけるために、しきい値を上げたり下げたりしてみて下さい。入力マップのローパスフィルターがかけられたバージョンを得るために --lowpass 15 --angpix 3.54オプションを付与させてrelion_image_handlerと呼ばれるコマンドラインプログラムを使用できることを覚えておいてください。初期しきい値としてよい値はだいたい、0.01から0.04のあたりです)
● Extend binary map this many pixels: 2
(上記のしきい値は白黒画像マスクを生成するのに使われます。このマップ中のホワイト値は全方向の多くのピクセルで増加します。これを使用して、最初のバイナリマスクのサイズを小さくします。)
● Add a soft-edge of this many pixels: 3
(これはマスクに余弦形状のソフトエッジを配置します。これは、とても鋭いマスクにかなり繊細である可能性があるFSCカーブに、マスクが及ぼす影響を計測する補正手順として重要です。マスクの生成は相対的に速いので、しばしば分解能の最も適した概算を得るためのマスクパラメーターと共に使います。)
【Helix】タブを無視して下さい。
例えば、first3dref_th002_ext2_edg3のようなエイリアスを使って下さい。【Display:】ボタンを使うことで、マスクの結果を通した切片を見たり、UCSFキメラにマスクを読み込ませることができます。後者は、マスクが構造全体をカプセル化するが、マスク内に多くの溶媒を残さないことを確認するために、自動精密手順のマップと一緒に良いアイデアになるかもしれません。
7.2 Postprocessing (後処理)
[Post-processing]jobタイプを選択して、【I/O】タブで以下のように設定して下さい。
● One of the 2 unfiltered half-maps: Refine3D/after first class3d/run half1 class001 unfil.mrc ● Solvent mask: MaskCreate/first3dref th002 ext2 edg3/mask.mrc ● Calibrated pixel size (A): 3.54
(ときどき、正確なピクセルサイズと思って作り始めたモデルが、実は数%異なっていたということがわかることがあります。RELION内では、この時までになしたもの全ては、まだ一貫しています。そのためマップの再精密化や、データのクラスの再分類をする必要はありません。正確なマップと最終解像度の推定のために必要なことが、ここに正しいピクセルサイズを提供することです。)
【Sharpen】タブで以下のように設定してください。
● MTF of the detector (STAR file): mtf falcon2 300kV.star
このメーカーは、自身のディテクターのために、プロジェクトディレクトリの中に保存されている mtf_falcon_300kV_down4.starファイルと同じ形式で保存することになる適切なMTFカーブを提供します。RELION wikiに記録した解析結果から、2,3個の一般的なカーブをダウンロードすることもできます。
● Estimate B-factor automatically: Yes
この手順は本誌[8]に基づき、最終解像度が確実に10Åを超えるように伸ばす必要があります。もしマップの解像度が10Åに満たない場合、独自の “ad-hoc” Bファクターを代わりに使う必要があるかもしれません。
● Lowest resolution for auto-B fit (A): 10
これは通常変える必要はありません。
● Use your own B-factor? No
【Filter】タブでは以下のように設定してください。
● Skip FSC-weighting? No
このオプションはときに、全体のマップ分解能を超えるような解像度へと拡張するマップ範囲を解析するのに便利です。今回は当てはまらないので、Noにします。
jobを実行(クラスターは必要ないほど、このjobはすぐ終わります)し、first3dref のようなエイリアスを使用して下さい。【Display】ボタンを使って、後処理されるマップと、この構造のFSCカーブとGuinierプロットを含んだPDFを通して、スライスを表示します。ChimeraでPostProcess/first3dref/postprocess.mrcマップを開くこともできます。そこでは、3D自動精密化の手順となる収束したマップでの全てのα-へリックスの場所を簡単に見ることが出来ます。分解能の推定は、以前に発表された[2]のフェイズランダムの手順に基づいてます。フェイズランダムマップのFSC(赤いカーブ)が、多かれ少なかれ、推定していた後処理マップの解像度のゼロ点であることを確認してください。
そうでない場合は、マスクが鮮明すぎるか、細部の描写が多すぎます。この場合は。強いローパスフィルターや、以前の[Mask creation]ステップでの、より広く、より薄いマスクを使用し、後処理を繰り返して下さい。実際に少し薄いマスクを作りたくなるかもしれないので、その場合はエッジがだいたい5ピクセルになるように試してみてください。このために【Continue now】ボタンを使った場合、以前の結果が上書きされます。左側にあるjobタイプブラウザをクリックし、【Run now!】ボタンで新しい出力ディレクトリが出来ます。
※マスクをきつく掛けるとマスクの影響による分解能を見ることになります。それを避けるためにマスク内が乱数であった場合(つまり、構造が無い場合)のFSCカーブと比較します。
8 Local-resolution estimate(ローカル解像度推定)
後処理プログラム【Post-processing】からの推定される分解能は広域的な推定となります。しかしながら、たった一つの数では、高分子複合体の再構築でしばしば観測される分解能が場所毎で異なることを描写できません。Alp Kucukelbir and Hemant Tagareはマップ全体の解像度の変化を推定する効果的なプログラムを記述しています[4]。RELION は、【Local resolution】 jobタイプを通してプログラムのラッパーを実装しています。【I/O】タブ上で以下のように設定してください。
● One of the two unfiltered half-maps: Refine3D/after first class3d/run half1 class001 unfil.mrc ● User-provided solvent mask: MaskCreate/first3dref th002 ext2 edg3/mask.mrc ● Calibrated pixel size: 3.54
(正しいピクセルサイズであると考えたモデルを構築し始めたときに、実際には数パーセントずれていたことがわかることがあります。RELION内では、この時点までの全てが一貫しています。そのため、再度、マップの精密化とデータの分類をする必要はありません。正確なマップと最終的な解像度推定のために正しいピクセルサイズをここで指定するだけで良いです。)
【ResMap】タブ上で以下のように設定してください。
● ResMap executable: /where/ever/it/is/resmap.exe
(RESMAP実行可能ファイルへのパスを指定します。シェル上で環境変数$RELION_RESMAP_EXECUTABLEを定義することで、どのディレクトリ上でもコマンドを打てることに注意して下さい。)
● P-value: 0.05
(これを変更することはほとんどありません)
● Highest resolution (A): 7
(ナイキスト周波数は7Åです)
● Lowest resolution (A): 10
(計算上での最低の解像度を指定します)
● Resolution step size (A): 0.5
(ステップ数を増やせば、より正確なローカル解像度マップの結果が導き出せますが、更に実行時間は長くなります。)
first3refのようなエイリアスを使用して下さい。RASMAPプログラムからポップアップ画面上に表示されたら、緑色の線が幾分水平であるかどうかを確認してください。それを変更するためには、RESMAPのGUIからフィルターボタンを利用して下さい。しかしながら、RELION上で生成されたマップに変更を加える必要がないことにしばしば気がつくでしょう。RESMAPプログラムが完了するまでに時間がかかることがあります。
Resmap/first3dref/half1_resmap.mrcの結果は、UCSF chimera上でTools -> Volume data -> Surface colorと選択した後、volume data valueで選択したローカル解像度によってPostprocess/first3dref/postprocess.mrcのマップに色をつけ、rasmapファイルをブラウズ(閲覧)するために使われます。特別な場合では、データがダウンサンプリングされ、ベータガラクトシダーゼはあまり柔軟な粒子ではないために、局所分解能の程度は見るためにあまり興味深いものではありません。しかし、とにかくそのことを知るのはいいことです。
9 Moving-processing & particle polishing
9.1 Getting organized(準備)
3.2で説明したように、RELIONパイプラインを通して、電子線に誘導されたモーションの補正を実行しているなら、全ての設定が既に終わっている状態です。RELONでの作業以外で、モーション補正を行った場合は、最初にインポートした顕微鏡写真と同じディレクトリ内(このチュートリアルではMicrographs)に全ての(補正に利用した)動画を置く必要があります。このディレクトリ内で、以前粒子を選択して平均化した顕微鏡写真と同じrootnameで動画が呼び出されているかを確認してください。例えば今回はrootnameが"movie"なので、mic001.mrcのムービーはmic001_movie.mrcsとして呼び出されます。[Import]jobを使って動画を取り込んでください。また、2D micrograph moviesオプションを選択してください。
また後ほど、動画-粒子の標準化は動画となった元の顕微鏡写真ごとの平均だと仮定されています。これがあなた自身のデータではそうではない場合、全体の画像処理を開始する前に、relion_image_handlerプログラムを使用して、任意の数の動画フレームから顕微鏡写真の平均を計算することができます。
9.2 Movie refinement(動画修正)
【Movie refinement】jobは、動画フレームから粒子を抽出し、それらを3次元モデルに対応させる、この二つの処理を組み合わせて行います。
【I/O】タブの設定:
• Input movies STAR file: MotionCorr/job002/corrected_micrograph movies.star
• Rootname of movies files: movie
(これらの動画のファイル名は_movie.mrcsで終わるためです。)
• Continue 3D auto-refine from: Refine3D/after_first_class3d/run it020 optimiser.star
([3D auto-refine]を実行後、含まれていた粒子のみ動画から抽出されます。これは、relion-1.4でのmovie-particle抽出とは大きく異なっています。
• Process micrographs in batches of: -1
(relion-2.0には、大きな動画データセットを処理する際に計算が不安定になることがあることが分かっているため、顕微鏡動画をより小さなバッチで処理するオプションがあります。このテストデータセットには15個のムービーのみが含まれており、バッチ処理は不要であるため、-1の値が使用されます。大きなデータセットの場合、50-100のバッチで顕微鏡写真を処理するのが通例です。)
(データセット:コンピューターで処理されるデータのまとまり。一般的に属性によって分類されたり、ある目的で集められたりしたデータが記録されたファイル群)
【extract】タブの設定:
• First movie frame to extract: 1 • Last movie frame to extract: 16 • Average every so many frames: 1
(少なくとも平均化したフレームで、約1 electron/Ų線量を持っているためこの値を調節します。各ムービーフレームで0.5-1 electron/Ų線量未満であれば1よりも高い値を使用します。)
• Maximum number of MPI nodes: 8
(このjobの第一ステップでは、動画粒子の抽出はI/Oディスク上で非常に集中します。 したがって、動作のこの部分で多数のMPI processes使用するのは効率的ではない可能性があります。【Running】タブ上で、与えられたMPI processes数にかかわらず、ここでは与えられた数より多くのProcessesを使うことはないでしょう。我々のクラスタでは、8がいいでしょう。)
• Particle box suze (pix): 100
(【Particle extraction】jobで以前使用したサイズと同じか確認してください。)
• Invert contrast? Yes
(【Particle extraction】jobの時と同じものを使用してください)
• Rescale particles? No
(【Particle extraction】jobの時と同じものを使用してください)
【normalise】タブの設定:
• Normalize movie-particles? Yes • Diameter background circle (pix): 60
(【Particle extraction】jobの時と同じものを使用してください)
• Stddev for white dust removal: -1 • Stddev for black dust removal: -1
【refine】タブの設定:
• Running average window: 7
(最適値は、粒子のサイズや線量率などによって決まります。基本的に、この粒子をどの程度正確に累積線量に合わせることができるかということが主な問題となるでしょう。リボソームのような大きな分子では、実行するためには3-5 electron/Ų線量程あれば十分でしょう。それよりも小さい粒子、例えば、β-ガラクシダーゼなどでは、これを7-9 electrons/Ųまで増やしてもよい)
• Stddev on the translations (pix): 1
(これにより、動画フレームが平均位置からどれだけ遠くに移動できるかが制限されます。)
• Also include rotational searches? No
(回転を含めると計算上、コストがかかってしまいます。そして、粒子の研磨手順はビーム誘導の回転を無視してしまいます。ビーム誘起回転を含む(かつ、研磨段階をスキップできる)非常に大きなオブジェクトにのみ有用です。それでも、リボソームでさえ、以下に概説する研磨手順が好ましいです。)
上で述べたように、この動作では最初に動画粒子を抽出し、3次元レファレンスとの配置合わせを行います。最初の段階では、制限されたMPI processesで実行されますが、2番目のパートは更に多くのMPI processesで実行する必要があります。そしてこれを【Runnning】タブ上で選択しないといけません。必要なメモリは、3次元自動レファレンスのときとほぼ同じです。したがって、スレッド数に応じて調整してください。おそらく、first3drefのようなエイリアスを再度使うことができるのではないでしょうか?
このjobから出力された唯一のファイルは、全ての動画粒子を精密化した動画フレーム粒子のSTARファイルとなります。これはMovieRefine / first3dref / run_avg7_off1_data.starという名前で呼び出されます。実行中の平均幅と変換の標準偏差の値が出力名に反映されるのは、これらのパラメータの値が異なる場合が多く、各設定に対してすべての動画粒子を再抽出しないようにするためです。 毎回、同じ動画抽出ステップを繰り返さないためにするためには、【Continue now】ボタンを使用して、同じ出力ディレクトリに対応するファイル名を持つ別のファイルを選択してください。
9.3 Particle polishing (粒子の洗練(研磨))
粒子のpolishingアルゴリズムは、MovieRefine/first3dref/run_avg7_off1_data.star から(おそらく非常にノイズの多い)移動軌跡(movement tracks)を取得し、それを利用して直線になるように適合させます。これらのトラックのノイズを更に減らすために、顕微鏡写真上の隣接する粒子がしばしば同様の方向に移動するという観測も利用します。これは、複数の隣接する粒子のトラックを一緒に評価することによって行われる。ここで、各粒子のその隣接トラックへの寄与は、粒子間距離のガウス関数によって決定される。このガウス分布の(ピクセルの)標準偏差は、ユーザによって決まります。第2のステップでは、polishingアルゴリズムは、Bファクターおよび線形強度ファクタを推定して、個々のムービーフレームにおけるシグナルの解像度依存パワーを記述する。このように、分解能に依存する電子線ダメージは、各粒子のすべての位置合わせしたムービーフレームの加重平均を行うことによりモデル化することができます。得られる粒子は、シグナル対ノイズ比(SNR)が増加するので、"polished"または"shiny"だと言われます。
[Particle polishing]jobタイプの【I/O】タブにて以下のようにして下さい
●Input STAR file with aligned movies: MovieRefine/first3dref/run avg7 off1 data.star
●Mask for the reconstruction: MaskCreate/first3dref th002 ext2 edg3/mask.mrc
(後処理で良い結果を出したものと同じものを使用してください。)
【Movement】タブの設定:
●Linear fit particle movements? Yes
●Stddev on particle distance (pix): 300
(粒子がより大きければ、ムービーの精密化からの移動軌跡の騒音は少なくなります。したがって、ここでは小さな値を使用できます。 リボソームについては、100ピクセルくらいをよく使用します。 値が小さいほど、複雑な動きのパターンが記述できます。)
【Damage】タブの設定:
●Perform B-factor weighting? Yes
●Highres-limit per-frame maps (A): 6
(このオプションは、単にコンピューターリソースを節約するためのものです。 個々のフレームの再構成は、ナイキストにまで拡張されないため、この値をここに指定して、シングルフレームの再構成を実行する際に考慮する周波数を制限することができます。)
●Lowres-limit B-factor estimation (A): 20
(このアルゴリズムは、個々のフレーム再構成と、一緒に平均化されたすべてのムービーフレームからの再構成との間の相対的なギニアプロットを推定します。 これらのプロットは直線にフィットするために使用されることがあり、[8]で提案された元のギニアプロットよりも頻繁に低い周波数がこれらのプロットに含まれることがあります。)
●Average frames B-factor estimation: 1
(常に最初に1の値を使用するようにしてください。 Bファクターの形状を信頼して推定するために適切な高解像度を得られない再構成があれば、このオプションを使用して3、5ムービーフレーム7つを平均することができます。 奇数の値のみが許可されます。)
●• Symmetry: D2
【normalise】タブの設定:
●Diameter background circle (pix): 60
(前と同じように、[Particle extraction]jobタイプで使用します。)
●Stddev for white dust removal: -1
●Stddev for black dust removal: -1
【Helix】タブを無視し、first3drefのようなエイリアスを使用して、jobを実行します。 粒子の研磨はGPU加速されていないので、おそらくこれをクラスタ上で実行したいと考えるでしょう。このjobの最大使用MPIプロセッサ数は、データのムービーフレーム数の二倍です。粒子ボックスが比較的大きい場合、メモリの要求は重要ですが、通常は3D自動精密化最後の反復実行時よりは大きくありません。
プログラムは、モーショントラックの線形フィッティング、電子線ダメージの計量のBファクタおよびスケールファクタを推定するための単一フレーム再構成を実行し、新しい光沢のある粒子を作成します。推定されたBファクタとスケールファクタは、これらのファクタを計算するために使用されたGuinierプロット(および直線とのフィット)と共に、そして動作軌跡と線形フィットはすべて、【Display】ボタンから開けるPolish/first3dref/logfile.pdf という大きなPDFファイルにあります。
私たちはサンプルが大きく動く最初の1-2フレームではしばしば比較的大きな負のBファクタを観測し、その後に電子線ダメージが入る後のフレームでも観測します。このチュートリアルのムービーはムービーフレームあたり1電子/Å2で記録されているため、16電子/Å2の合計線量はそれほど高くなく、電子線損傷のみが重要な役割を果たす訳ではありません。多くの場合、最初の数フレームは比較的悪い可能性があるが、GuinierプロットのY軸(すなわち、線形強度スケールファクタ)を備えたフィッティングされたラインの切片は線量とともに減少する。
何らかの形で推定されたBファクタまたはスケールファクタが奇妙に見える場合、Guinierプロットを通じて線形フィットを調べることができます。時には、Guinierプロットは、【Damage】タブに入力された全周波数領域にわたって直線でよくフィットすることができません。その後、異なるパラメータでjobを再実行する価値があります。また、シングルフレームの再構成が上記の20Åの周波数を超えて拡張されない場合、ラインにフィットせず、(推定されたB因子についてNaNを与える)プログラムは動作しません。そのような場合、Bファクタの計量をスキップするか、または同じタブの Average frames B-factor estimation: 1 を使用して複数のフレームを平均することを選択します。【Display】ボタンから出力された研磨粒子(shiny.star)を使ってSTARファイルを視覚化して、すべてが正しく行われていることを確認することは、おそらく悪い考えではありません。
何らかの形で研磨結果に満足していない場合は、【Continue run】ボタンを使用して計算の一部を繰り返すことができます。 それをクリックすると、プログラムは次のファイルを探し、そこにあればその計算ステップをスキップして次のステップに移ります:
●frame0??_half?_class001_unfil.mrc (これらの地図のいずれかが欠落している場合は、再計算されます。それらが既に存在する場合、それらの再構成計算はスキップされる。Average frames B-factor estimation オプションを変更した場合は、すべての再構成を再計算すべきです。その場合は、古いものを続けるのではなく、新しいjobを開始する方が良いでしょう。あるいは、既存のフレームの再構築をすべて取り除く必要があります。)
●bfactors.star (このファイルが見つからない場合、プログラムは既存のすべてのフレーム再構成を読み込み、BファクタとスケールファクタをGuinierプロットを通して線形フィットするかどうか決定します。 したがって、たとえば【Damage】タブでフィットした解像度範囲を変更したが、Average frames B-factor estimation を変更しなかった場合、既存のbfactors.starファイルを削除すると非常に便利なので、新しいBファクタとスケールファクタが計算されますが、すべてのマップをもう再構築する必要はありません。 これはとても高速に進むでしょう。)
●shiny.star (このファイルがない場合、既存の bfactors.starファイルが読み込まれ、磨き上げられた粒子(polished particles)はこれらのパラメータに従って再計量されます。 たとえば、以前に決定されたBファクタとスケールファクタのプロットに滑らかな曲線をフィットさせることを決定し、既存の bfactors.starファイルを新しくフィットしたものに置き換えた場合など、これは非常に便利です。 この場合、プログラムは磨き上げられた粒子(polished particles)のみを再作成します。)
9.4 Re-refine the polished particles(磨き上げられた(洗練された)粒子の再精密化)
shiny.starの磨かれた粒子は、元のものと比べてシグナル対ノイズ比が向上しています。 これは、さらに分類分けするときに役立ちます。 この時点での異なる分類分けの戦略の詳細な説明については、Methods in Enzymology [12]の最新の章を参照してください。 この章はまだオンラインでは入手できないため、以下コマンドで初期の資料をダウンロードできます。
wget ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relionreview2016.pdf
私達はいつでもpolished particlesを使って最終的な再構成【3D auto-refine】を実行します。より高い解像度マップをもたらすように、「確率加重 angular assignment(角度の割り当て) 」を実行する替わりに、最も可能性の高い方向として各粒子の角度を決定します。したがって、shiny.star を入力として3D auto-refineを繰り返してください。 この精密化の後、後処理と局所分解の推定のやり直しをすることを忘れないでください。
このテストケースでは、【movie-refinement】 と 【particle polishing】 の前に、再構成は、既に(ダウンサンプリングされた)データのナイキスト周波数に達しました。 したがって、後者は三次元画像にほとんど影響しませんでした。 このデモンストレーションは、動画処理の仕方を教える上でまだ役立つことを願っています。
10 Wrapping up(まとめ)
どうやって最終的な再構築をするのか気になるでしょう。[Finished jobs]リストから実行する最後の動作を選択し、【Job actions】ボタンからMake flowchartオプションを試して下さい。これを実行するには、【LATEX】や【TikZ】をコンピューターにインストールする必要があります。最初のフローチャートはどうやってここまで終わらせるかについて示します。10ステップ以上からなるフローチャートは複数の塊に分けられることに注意してください。ワークフローにブランチ(分岐)があるかもしれません。そのため、最後のjobのフローチャートに従った後にも、またそれぞれのブランチでフローチャートがあるでしょう。ブルーボックスをクリックすることで、PDBファイル内の位置に相当するデータを得られます。最後のページには、正確なjob名のないフローチャートが表示されます。これは、出版目的に役立ちます。
以上です!このチュートリアルを楽しんで、それが役に立つことを願います。もし他にRELIONについて質問があるならば、最初にRELION WikiのFAQとCCPEMメーリングリストを確認してみてください。もし望んだ情報が得られなければ、CCPEMリストを利用して質問してみてください。決してSjorsに直接メールをしないで下さい。質問すべてに返答できるわけではありませんのでご了承下さい。
もしRELIONが利用者の研究に有益であると判断できましたら、私たちの論文を引用したり、周りに伝えてみてください。
References
[1] Xiao-chen Bai, Eeson Rajendra, Guanghui Yang, Yigong Shi, and Sjors HW Scheres. Sampling the conformational space of the catalytic subunit of human -secretase. eLife, 4, December 2015.
[2] Shaoxia Chen, Greg McMullan, Abdul R. Faruqi, Garib N. Murshudov, Judith M. Short, Sjors H W Scheres, and Richard Henderson. High-resolution noise substitution to measure overfitting and validate resolution in 3d structure determination by single particle electron cryomicroscopy. Ultramicroscopy, 135:24–35, December 2013.
[3] Timothy Grant and Nikolaus Grigorieff. Measuring the optimal exposure for single particle cryo-EM using a 2.6 reconstruction of rotavirus VP6. eLife, 4:e06980, 2015.
[4] Alp Kucukelbir, Fred J Sigworth, and Hemant D Tagare. Quantifying the local resolution of cryo-EM density maps. Nature methods, 11(1):63–65, January 2014.
[5] Xueming Li, Paul Mooney, Shawn Zheng, Christopher R Booth, Michael B Braunfeld, Sander Gubbens, David A Agard, and Yifan Cheng. Electron counting and beam-induced motion correction enable near-atomicresolution single-particle cryo-EM. Nature methods, 10(6):584–590, June 2013.
[6] Joseph A. Mindell and Nikolaus Grigorieff. Accurate determination of local defocus and specimen tilt in electron microscopy. Journal of Structural Biology, 142(3):334–347, June 2003.
[7] Alexis Rohou and Nikolaus Grigorieff. CTFFIND4: Fast and accurate defocus estimation from electron micrographs. Journal of Structural Biology, 192(2):216–221, November 2015.
[8] Peter B Rosenthal and Richard Henderson. Optimal determination of particle orientation, absolute hand, and contrast loss in single-particle electron cryomicroscopy. Journal of Molecular Biology, 333(4):721–745, October 2003.
[9] Sjors H W Scheres. Classification of Structural Heterogeneity by MaximumLikelihood Methods. In Cryo-EM, Part B: 3-D Reconstruction, volume 482 of Methods in Enzymology, pages 295–320. Academic Press, 2010.
[10] Sjors H W Scheres. A Bayesian view on cryo-EM structure determination. Journal of Molecular Biology, 415(2):406–418, January 2012.
[11] Sjors H W Scheres. RELION: Implementation of a Bayesian approach to cryo-EM structure determination. Journal of Structural Biology, 180(3):519–530, December 2012.
[12] Sjors H W Scheres. Processing of structurally heterogeneous cryo-em data in relion. Methods in Enzymology, in press, 2016.
[13] Sjors H W Scheres and Shaoxia Chen. Prevention of overfitting in cryo-EM structure determination. Nature methods, 9(9):853–854, September 2012.
[14] Sjors H W Scheres, R. Nunez-Ramirez, C. O. S Sorzano, J. M Carazo, and R. Marabini. Image processing for electron microscopy single-particle analysis using XMIPP. Nature Protocols, 3(6):977–90, 2008.
[15] J M Smith. Ximdisp–A visualization tool to aid structure determination from electron microscope images. Journal of structural biology, 125(2-3):223–228, May 1999.
[16] Guang Tang, Liwei Peng, Philip R. Baldwin, Deepinder S. Mann, Wen Jiang, Ian Rees, and Steven J. Ludtke. EMAN2: an extensible image processing suite for electron microscopy. Journal of Structural Biology, 157(1):38–46, January 2007.
[17] Kutti R. Vinothkumar, Greg McMullan, and Richard Henderson. Molecular Mechanism of Antibody-Mediated Activation of -galactosidase. Structure, 22(4):621–627, April 2014.
[18] Kai Zhang. Gctf: Real-time CTF determination and correction. Journal of Structural Biology, 193(1):1–12, January 2016.
FAQ
Q1. ルートネームとはなんですか。 A1. Relionでは、ルートネーム+画像の意味を表す拡張+フォーマットを表現する拡張子といった形で、それぞれの画像を表現しています。同じ画像に由来するものはルートネームを共通にもつ必要があります。