Imaris
Imarisは,汎用の蛍光画像等の表示・解析ツールです。
Imaris Format
[https://imaris.oxinst.com/support/imaris-file-format]から翻訳し,一部情報を追加したものです。
Imarisファイルフォーマットは、非常に大きな画像を高速に視覚化できるように設計されています。この目的のために、オリジナルの画像データだけでなく、オリジナルデータの低解像度バージョンも保存されます。これにより、可視化ソフトは低解像度のデータだけで十分な場合に、そのデータを読み込むことができます。また、高速に可視化するために、Imarisファイルフォーマットは画像データを連続した3Dチャンク(3Dブロックのhdf用語)で保存し、可視化ソフトウェアが視野内にあるデータのみをロードすることを可能にします。多解像度構造とチャンク単位での保存レイアウトは、この高性能ファイルフォーマットの基礎となっています。
概要 このファイルフォーマットは、イリノイ大学アーバナ・シャンペーン校の国立スーパーコンピューティング・アプリケーションズセンターが開発した標準的なHDF5(Hierarchical Data Format 5)をベースにしています。これにより、広く利用可能なHDF5ライブラリやツールを使用することができます。 Imaris 5.5ファイルは、主に3種類の画像のグループ(またはフォルダ)で構成されています。 データセット(DataSet)。データセット:画像自身(すべてのスライス、チャンネル、タイムポイント、解像度レベルを含む) DataSetInfo:画像のメタデータ情報(ボクセルサイズなど) Thumbnail: 画像の2Dサムネイル。
追加のグループには、「Imaris Scene」が含まれる場合があります。 SceneまたはScene8:サーパスオブジェクト、アノテーションなど。
このドキュメントでは、グループ、アトリビュート、データセットという用語は、HDF5のドキュメントで与えられている概念を参照しています。パスが与えられている場合(/DataSet/ResolutionLevel 12)、対応するグループとサブグループを開く必要があります。
以前の "Imaris Classic "と "Imaris3 "ファイルフォーマットは、HDF5の仕様に基づいていないため、このドキュメントでは説明されていません。
ツールおよびリソース
Bitplaneは、HDF5ファイルを読み書きするためのバックエンドとして、HDF GroupのHDF5ライブラリを使用します。HDF5ファイルフォーマットについて読むには、HDF5グループのウェブサイト([http://www.hdfgroup.org/HDF5/ ])が良い出発点となります。HDFグループは、そのライブラリをオープンソースとして無償で提供しています。HDF5リーダー/ライターを自作するよりも、このライブラリーを利用することをお勧めします。
また、HDF Groupはライブラリとともに有用なツールも提供している。注目すべきは、JavaベースのHDFファイルインスペクターであるHDFViewです。これは、ここから無料でダウンロードできます。
HDFViewを使って、IMSファイルのサンプルを見て、ファイル形式を理解することを強くお勧めします。
構造
Imaris 5.5のファイル構造は、ルートの「フォルダ」と、DataSet、DataSetInfo、Thumbnailの3つの主要なグループから構成されています。
右のスクリーンショットは、HDFViewでIMSファイルを表示したもので、HDFファイル構造を示しています。この例では、1チャンネル、5レベルの解像度を持つ画像です。
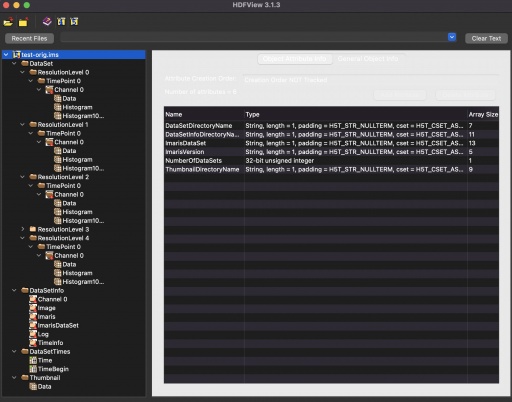
DataSet フォルダには実際の画像データが格納されています。DataSetInfo フォルダには、説明用のパラメータが格納されています。Thumbnailフォルダには、画像の2Dサムネイルが格納されています。
| Path | Attribute | Value | Description |
| / | ImarisDataSet | ImarisDataSet | Specific to format |
| FormatVersion | 5.5.0 | Format version | |
| DataSetInfo | DataSetの情報を含む | ||
| Thumbnail | Thumbnailのデータを含む |
データセット
Imaris 5.5のDataSetは次に示す構造になっています。 データセットは1つ以上の3次元画像から構成されます(2次元画像の場合、1次元のサイズは1に等しい)。 データセットが複数のチャンネルまたは複数のタイムポイントを持つ場合、各チャンネルおよび各タイムポイントの3D画像を格納します。 データセットはチャンクを使ってファイルに保存され(HDF5のドキュメントを参照)、オプションでGNU GZIPを使って圧縮することができます。読み出しのパフォーマンスを良くするために、チャンクはImarisソフトウェアが視覚化のために要求するサイズで保存する必要があります(下記のImaris Datablocksの章を参照)。
HDF5のチャンクメカニズムは、データセットの有効サイズがオリジナルの3D画像のサイズより大きくなる可能性があることを意味します。したがって、ImageSizeZ、ImageSizeY、ImageSizeXの属性も各Datasetと一緒に保存されます。
データセットのタイプは、保存されるデータのタイプに類似している必要があります。有効な型は以下の通りです。
| HDF5 Data type | Imaris Image Data type |
| H5T_NATIVE_UCHAR | 8 bit unsigned integer (char) |
| H5T_NATIVE_USHORT | 16 bit unsigned integer (short) |
| H5T_NATIVE_UINT32 | 32 bit unsigned integer |
| H5T_NATIVE_FLOAT | H5T_NATIVE_FLOAT |
データセットの作成属性は、chunked、storage allocation time incremental、fill value none、compression none、GZIPのいずれかとする。GZIP圧縮が有効な場合、有効なGZIPパラメータを使用できます(例:圧縮率0~9は許容範囲、3は推奨)。
各3D画像と一緒に、対応するヒストグラムも保存されます(下の表参照)。ヒストグラムはHDF5の1次元データセットで,型は64bitの符号なし整数です.その長さは,HDF5ライブラリを使って読み出すことができます.最初の値は,画像中の値HistogramMinのカウントに相当します.最後の値は、画像中の値HistogramMaxのカウントです。
| Path | Attribute | Description |
| /DataSet/ResolutionLevel 0/TimePoint 0/Channel 0 | Informations concerning resolution 0, time point 0 and channel 0 | |
| ImageSizeX = 285 | The size in X in pixel for Resolution Level 0 | |
| ImageSizeY = 218 | The size in Y in pixel | |
| ImageSizeZ = 64 | The size in Z in pixel | |
| HistogramMin = 0 | The minimal value of the histogram corresponding to this image | |
| HistogramMax = 255 | The maximal value of the histogram | |
| /DataSet/Resolution Level 0/TimePoint 0/Channel 0/Data | THE image, with size ImageSizeX*ImageSizeY*ImageSizeZ | |
| /DataSet/Resolution Level 0/TimePoint 0/Channel 0/Histogram | The histogram of the image. The first bin corresponds to HistogramMin and the last bin to HistogramMax | |
| /DataSet/ResolutionLevel 0/TimePoint 0/Channel 1 | Informations concerning resolution 0, time point 0 and channel 1 | |
| /DataSet/ResolutionLevel 0/TimePoint 0/Channel 0/Data | Image data for resolution 0, time point 0 and channel 0 | |
| /DataSet/ResolutionLevel 0/TimePoint 0/Channel 0/Histogram | Image histogram for resolution 0, time point 0 and channel 0 |
コード例
mFileId = H5Fopen(mFileName.c_str(), H5F_ACC_RDONLY, H5P_DEFAULT);
hid_t vDataSetId = H5Gopen(mFileId, “DataSet”);
hid_t vLevelId = H5Gopen(vDataSetId, “Resolution Level 0”);
hid_t vTimePointId = H5Gopen(vLevelId, "TimePoint 0");
hid_t vChannelId = H5Gopen(vTimePointId, "Channel 0");
hid_t vDataId = H5Dopen(vChannelId, “Data”);
// read the attributes ImageSizeX,Y,Z hsize_t vFileDim[3] = {ImageSizeZ, ImageSizeY, ImagesizeX};
hid_t vFileSpaceId = H5Screate_simple(3, vFileDim, NULL); char* vBuffer = new vBuffer[ImageSizeZ*ImageSizeY*ImageSizeX];
H5Dread(vDataId, H5T_NATIVE_CHAR, H5S_ALL, vFileSpaceId, H5P_DEFAULT, vBuffer);
上記の例では、解像度0、時間点0、チャンネル0に対応する3D画像全体が読み込まれます。HDF5 ライブラリを使用すると、メモリやその他の要件に応じて、画像の一部だけを読み込むことも可能です。
データセット情報
DataSetInfoグループには、Imarisからのすべての画像パラメータが含まれています。セクションはHDF5グループとして、パラメータはHDF5アトリビュートとして格納されています。HDF5属性はH5Aopen_nameで開き、H5Areadで読み込むことができます。属性はすべて文字列型として保存されます。つまり、数値は文字列に変換する必要があります。属性のHDF5型はH5T_C_S1(C String)の配列である。
以下、LSMはLaser Scanning Microscopeの略称として使用する。
| Group / Section Name | Attribute / Parameter Name | Description | |
| ImarisDataSet | This section holds information about the data organization in the file. (always present) | ||
| Creator = Imaris | |||
| NumberOfImages = 1 | At the moment there is only one multi-channel-time image per file | ||
| Version = 5.5 | The version of Imaris which created this file | ||
| Imaris | Information about the thumbnail and Imaris. (always present) | ||
| Filename = retina.ims | The name of the original file | ||
| ManufactorString = Generic Imaris 3.x | Manufactor information | ||
| ManufactorType = LSM | Manufactor type information | ||
| ThumbnailMode = thumbnailMIP | The type of data representation of the thumbnail. Valid values are "thumbnailNone", "thumbnailMiddleSection", "thumbnailMIP" or "thumbnailNone". | ||
| Version = 5.5 | The version of Imaris which created this file | ||
| Image | Information about the DataSet. (always present) | ||
| Description = nucleus | Detailed description of the image in plain text (can be multiple lines) | ||
| ExtMax0 = 46.7464 | Data max. extension X (in given units: um, nm, …) | ||
| ExtMax1 = 35.7182 | Data max. extension Y | ||
| ExtMax2 = 12.6 | Data max. extension Z | ||
| ExtMin0 = -10.3 | Data origin X | ||
| ExtMin1 = -1.5 | Data origin Y | ||
| ExtMin2 = 3.4 | Data origin Z | ||
| LensPower = 63x | Deconvolution parameter | ||
| Name = m1193.pic | Short description of the image (some characters) | ||
| Noc = 2 | Number of channels | ||
| RecordingDate = 1991-10-01 16:45:45 | “YYYY-MM-DD HH:MM:SS” | ||
| Unit = um | "m“, "mm“, "um“ or "nm“ | ||
| X = 285 | Image Size X (in voxels) | ||
| Y = 218 | Image Size Y (in voxels) | ||
| Z = 64 | Image Size Z (in voxels) | ||
| Information about channel X (there is one section per channel) (always present) | |||
| Color = 1 0 0 | The base color (r,g,b float values from 0.0 to 1.0) | ||
| ColorMode = BaseColor | “BaseColor”, “TableColor” see example below for the color table mode. | ||
| ColorOpacity = 0.168 | The opacity with which the volume rendering displays the channel (float value from 0.0 to 1.0) | ||
| ColorRange = 0 194.921 | The display “contrast” | ||
| Description = Cy5 | Detailed description of the channel | ||
| Gain = 0 | Deconvolution parameter | ||
| LSMEmissionWavelength = | Emission Wavelength | ||
| LSMExcitationWavelength = | Excitation Wavelength | ||
| LSMPhotons = | Deconvolution parameter | ||
| LSMPinhole = | Pinhole diameter | ||
| Max = 255 | The data maximum value of the channel image | ||
| MicroscopeMode = | Deconvolution parameter | ||
| Min = 0 | The data minimum value of the channel image | ||
| Name = CollagenIV (TxRed) | Short description of the channel (some chars) | ||
| NumericalAperture = Numerical Aperture | |||
| Offset = 0 | Deconvolution parameter | ||
| Pinhole = 0 | Deconvolution parameter | ||
| RefractionIndexEmbedding = | Deconvolution parameter | ||
| RefractionIndexImmersion = | Deconvolution parameter | ||
| TimeInfo | Information about time for all channels. (always present) | ||
| DataSetTimePoints = 1 | The number of time points in the DataSet | ||
| FileTimePoints = 1 | The number of time points in the file (currently the same as DataSetTimePoints) | ||
| TimePoint1 = 1991-10-01 16:45:45.000 | Time for time point 1. Time must be strictly increasing between time points. | ||
| Log | Any information about the history of the image (especially the data manipulation). | ||
| Entries = 3 | Number of entries (number of image data modifications). Can be zero. | ||
| Entry0 = | First modification | ||
| Entry1 = | Second modification | ||
| Entry2 = | Third modification |