Difference between revisions of "Electron Tomography"
(→3D Reconstruction) |
(→Example1 (Reconstruction of 1 axis tilt)) |
||
| Line 304: | Line 304: | ||
===[[mrc2Dto3D]]=== | ===[[mrc2Dto3D]]=== | ||
====Example1 (Reconstruction of 1 axis tilt)==== | ====Example1 (Reconstruction of 1 axis tilt)==== | ||
| − | <div> | + | <div> If the tilt axis don't deviate, set the angle information directly to input file list.</div> |
<br> | <br> | ||
| Line 311: | Line 311: | ||
<tr> | <tr> | ||
<td><p align="Center">[[File:1WDC-Tom-Fit-2d.png]]<br> | <td><p align="Center">[[File:1WDC-Tom-Fit-2d.png]]<br> | ||
| − | + | 10° interval<br></p> | |
</td> | </td> | ||
</tr> | </tr> | ||
| Line 317: | Line 317: | ||
<br> | <br> | ||
| − | <div> | + | <div>Command: [[mrc2Dto3D]] -i Input.3dlst -o Input.3d -m 1</div> |
<br> | <br> | ||
| − | <div>[[Media:Makefile-Tomogram.zip| | + | <div>By using [[Media:Makefile-Tomogram.zip|This Makefile]], perform the following command for reconstruction.</div> |
| − | <div> | + | <div>Setting data</div> |
<pre> | <pre> | ||
# For Reconstruction | # For Reconstruction | ||
Revision as of 06:50, 26 September 2014
Electron Tomography is one of 3D Reconstruction using electron microscope. It is the method that reconstruct 3D image by electron micrographs which is projected the same photo field for several angle, then it creates Tomogram by using computer.
In the following, describe its method in order.
Contents
take tilt-series images
 1 Axis Tilt(Center is 0°'s image) |
 2 Axis Tilt(Center is 0°'s image)(as 90° rotation image for 1 Axis Tilt's image) |
Image Correction
Tilt-series image's defocus is larger than normal electron photograph's one, because it is taken as whole field is underfocus. In this case, CTF Correction is not required, because outside data of 1st thon ring don't have almost information if you use electron gun as LaB6. But, in the case of using field-emission electron gun (FEG), note that you might get a incorrect data.
Rough Alignment
Align roughly each tilt image by using the correlation among images around center.
Preprocess(Windowing)
# For Windowing WIN_X=0.2 WIN_X_MAX=0.2 WIN_Y=0.1 WIN_Y_MAX=0.1 WIN_MODE=18
make Windowing
Alignment
e.g. case of images whose interval is 2 degree
Align 2° image to 0° image.
Align 4° image to Aligned 2° image.
Align 6° image to Aligned 4° image.
.
.
case of using mrcImageCorrelation
Example 1
make CorFit1
Fine Alignment
Example 1(Alignment of tilt axis)
# RotMode ROTMODE=ZOYS # Rot1 ROT1MIN=10 ROT1MAX=10 ROT1D=10 # Rot2 ROT2MIN=-60 ROT2MAX=60 ROT2D=2 # Rot3 ROT3MIN=0 ROT3MAX=0 ROT3D=30 ### For mrcImageMove SHIFT2MAX=10 SHIFT3MAX=0 ### For mrcImageTiltAxisSearch TILTMIN=0 TILTMAX=20 TILTN=10 TILTITER=1 TILTSCALE=5
make TiltFit
9.895
| Before correction | ||
 |
 |
 |
| After correction | ||
 |
 |
 |
| xy-plane | yz-plane | zx-plane |
Example2(Repeatedly Alignment of tilt axis)
# For mrcImageTiltAxisSearch IN_TILT_EXT=roi # RotMode ROTMODE=ZEYR # Rot1 ROT1MIN=1 ROT1MAX=1 ROT1D=1 # Rot2 ROT2MIN=-60 ROT2MAX=60 ROT2D=10 # Rot3 ROT3MIN=0 ROT3MAX=0 ROT3D=10 ### For mrcImageTiltAxisSearch TILTMIN=-10 TILTMAX=10 TILTN=10 TILTITER=100 TILTSCALE=5
make TiltFit
0.084 0.161 0.230 0.294 0.345 ... 0.701 0.700 0.685 0.706 0.701
3D Reconstruction
Min Max |
0 (0, 0, 0) 4 (31, 26, 26) |
mrc2Dto3D
Example1 (Reconstruction of 1 axis tilt)
# For Reconstruction IN_3D_EXT=fit # RotMode ROTMODE=YOYS # Rot1 ROT1MIN=-60 ROT1MAX=60 ROT1D=2 # Rot2 ROT2MIN=0 ROT2MAX=0 # Rot3 ROT3MIN=0 ROT3MAX=0
make 3DList make Input.3d
Min Max |
-0.00437076 (39, 34, 36) 0.00799233 (37, 34, 36) |
実行例2(2軸傾斜での再構成)
make all
今回OutputしたInformationfile
DataA_006.mrcsmth-0000.roi Rect 0.000000 0.000000 63.000000 0.000000 63.000000 63.000000 0.000000 63.000000
Min Max |
-9.32199 (29, 37, 34) 21.995 (40, 35, 32) |
実行例3(Doubleを使用した2軸傾斜での再構成)
.roilst.mrc3d: # mrc2Dto3D -I $*.roilst -o $*.mrc3d -single 0 -InterpolationMode 2 -m 1 mrc2Dto3D -I $*.roilst -o $*.mrc3d -Double -InterpolationMode 2 -m 1
Min Max |
-10360.9 (41, 24, 0) 8132.05 (40, 35, 31) |
ラドン変換を使った3次元再構成
位置合わせ済みの2Dリスト
↓mrcImageSinogramCreate
Sinogramリスト
↓mrcRadon2Dto3D
3Dラドンfile
↓mrcImageInverseRadonTransform
実行例1
make Radon3D
| xy-plane | yz-plane | zx-plane |
 |
 |
 |
Min Max |
1.84923e+06 (0, 59, 28) 1.17991e+07 (34, 31, 30) |
実行例2
### RadonTransform RBP_MODE=4 RBP_OPTION=-Rmin 0.05 -Rmax 0.1
make Radon3D
| xy-plane | yz-plane | zx-plane |
 |
 |
 |
Min Max |
-344789 (25, 25, 0) 631546 (37, 34, 28) |
Problems of Electron Tomography image
ミッシングエリア
Min Max |
0 (0, 0, 0) 2 (29, 29, 14) |
TILTAXISNUMBER=1 # Single: 1 Double: 2
make Target.ini2d make TestData2DSet make all
Min Max |
-0.00330818 (52, 36, 32) 0.00425516 (13, 37, 32) |
TILTAXISNUMBER=2 # Single: 1 Double: 2
make Target.ini2d1 make TestData2DSetDouble make all
Min Max |
-0.00248567 (12, 38, 32) 0.00344027 (13, 37, 32) |
画像の位置,フォーカスの問題
parallel transformの補正
軸の決定
トモグラフに使われる画像処理
Smoothing
実行例1
Min Max |
-0.00268221 (76, 92, 66) 0.00642324 (89, 42, 71) |
Min Max |
-0.00136636 (75, 42, 133) 0.00292404 (94, 65, 68) |
セグメンテーション
3次元画像の切り出し
実行例1
Min Max |
-0.00136636 (75, 42, 133) 0.00292404 (94, 65, 68) |
Min Max |
-0.0012017 (5, 9, 13) 0.00263957 (13, 6, 15) |
2次元画像の切り出し
傾斜画像(2D画像複数)(前処理済) ↓ 切り出し範囲のSetting(Display2: 0°'s imageのみ) 0°の切り出しInformationfile(ROIInformation) ↓ 各傾斜画像の切り出し範囲を計算する(現在はMakefile内で処理) 各傾斜画像の切り出しInformation ↓ 画像の切り出し(mrcImageROIs) 切り出された複数's imagefile(切り出し数 × 傾斜数分) ↓ 位置合わせ(mrcImageCorrelarion + Makefileの処理) 位置合わせ済みの切り出しInformation(傾斜数分) ↓ 再度画像の切り出し(mrcImageROIs) (※ 切り出し画像をシフトするのではなく,切り出し位置を再Settingして切り出します) 位置合わせ済み's image(ROI)file(切り出し数 × 傾斜数分) ↓ 角度InformationfileのCreate(Makefile内の処理) 角度Informationfile(切り出し数分) ↓ 3次元再構成(mrc2Dto3D) 3次元画像(切り出し数分)
実行例1(1軸傾斜のサブトモグラム)
make all
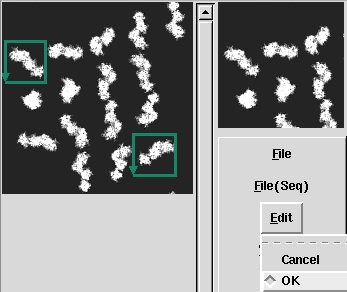
切り出し部分決めてEdit->OKで決定します. |
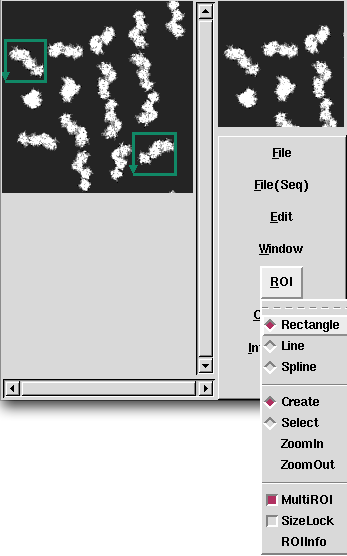
複数切り出したいときはROI->MultiROIを選択します. |
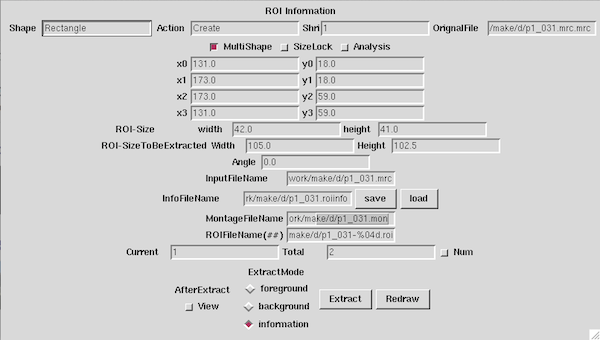
| 今回はInformationだけCreateすれば,切り出しは自動で行われます. ROI InformationウィンドウにてInfoFileName右側のsaveボタンを押すとfileがCreateされます. |
p1_031-0000.roi Rect 20 30 60 30 60 70 20 70 p1_031-0001.roi Rect 15 75 55 75 55 115 15 115 p1_031-0002.roi Rect 25 110 65 110 65 150 25 150 p1_031-0003.roi Rect 45 0 85 0 85 40 45 40 p1_031-0004.roi Rect 60 50 100 50 100 90 60 90 p1_031-0005.roi Rect 55 80 95 80 95 120 55 120 p1_031-0006.roi Rect 55 120 95 120 95 160 55 160 p1_031-0007.roi Rect 85 15 125 15 125 55 85 55 p1_031-0008.roi Rect 85 51 125 51 125 91 85 91 p1_031-0009.roi Rect 85 91 125 91 125 131 85 131 p1_031-0010.roi Rect 93 130 133 130 133 170 93 170 p1_031-0011.roi Rect 115 25 155 25 155 65 115 65 p1_031-0012.roi Rect 120 60 160 60 160 100 120 100 p1_031-0013.roi Rect 123 100 163 100 163 140 123 140 p1_031-0014.roi Rect 115 140 155 140 155 180 115 180 p1_031-0015.roi Rect 152 152 192 152 192 192 152 192
 |
||
| -60° | 0° |
60° |
| xy-plane | |
| yz-plane | |
| zx-plane |
実行例2(2軸傾斜のサブトモグラム)
| x-axis回転 | |||
| y-axis回転 |
make all
DataA_006-0000.roi Rect 0.722314 137.147614 33.000000 106.000000 49.053597 122.636041 16.775911 153.783654 DataA_006-0001.roi Rect 15.000000 79.000000 42.000000 79.000000 42.000000 107.000000 15.000000 107.000000 DataA_006-0002.roi Rect 13.000000 39.000000 58.000000 39.000000 58.000000 64.000000 13.000000 64.000000 DataA_006-0003.roi Rect 41.000000 129.000000 84.000000 129.000000 84.000000 154.000000 41.000000 154.000000 DataA_006-0004.roi Rect 51.000000 85.000000 82.000000 85.000000 82.000000 119.000000 51.000000 119.000000 DataA_006-0005.roi Rect 47.269722 69.494676 78.455043 40.316766 95.000000 58.000000 63.814679 87.177909 DataA_006-0006.roi Rect 66.000000 4.000000 92.000000 4.000000 92.000000 44.000000 66.000000 44.000000 DataA_006-0007.roi Rect 77.597890 150.922358 95.484138 132.052220 120.886248 156.129861 103.000000 175.000000 DataA_006-0008.roi Rect 93.000000 92.000000 115.000000 92.000000 115.000000 132.000000 93.000000 132.000000 DataA_006-0009.roi Rect 100.000000 51.000000 121.000000 51.000000 121.000000 93.000000 100.000000 93.000000 DataA_006-0010.roi Rect 98.589377 19.087800 119.760822 10.100593 135.000000 46.000000 113.828555 54.987208 DataA_006-0011.roi Rect 122.000000 141.000000 146.000000 141.000000 146.000000 184.000000 122.000000 184.000000 DataA_006-0012.roi Rect 136.883039 95.213292 160.000000 104.000000 144.116961 145.786708 121.000000 137.000000 DataA_006-0013.roi Rect 127.000000 73.000000 146.061183 60.635990 169.000000 96.000000 149.938817 108.364010 DataA_006-0014.roi Rect 140.000000 22.000000 177.361289 43.286947 166.470292 62.402025 129.109004 41.115078 DataA_006-0015.roi Rect 163.000000 152.000000 183.000000 152.000000 183.000000 190.000000 163.000000 190.000000
| xy-plane | |
| yz-plane | |
| zx-plane |
Interpretation of 3D Reconstruction
また,多様な構造を含むため,3次元画像から興味あるセグメントを切り出すなどの作業が必要です.そのための支援ソフトウェアが必要です.
Averaging of Subtomograms
 |
 |
 |
 |
 |
 |
 |
 |
 |
| xy-plane | yz-plane | zx-plane |
実行例1
| xy-plane | |
| yz-plane | |
| zx-plane |
Min Max |
-0.0012017 (5, 9, 13) 0.00263957 (13, 6, 15) |
| xy-plane | |
| yz-plane | |
| zx-plane |
Min Max |
-0.000536652 (6, 13, 12) 0.00177258 (13, 17, 13) |
実行例2
| xy-plane | |
| yz-plane | |
| zx-plane |
Min Max |
-5.47817 (34, 19, 20) 16.0715 (31, 22, 19) |
| xy-plane | |
| yz-plane | |
| zx-plane |
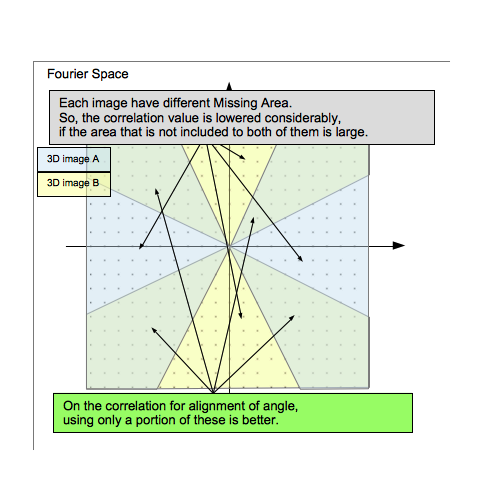
これはそれぞれのサブトモグラムの3次元像にてミッシングエリアが異なるので,どちらかにdataがない角度では相関値が大Widthに下がってしまうためです.
Min Max |
-2.32263 (22, 29, 20) 5.20816 (19, 24, 22) |