「単粒子解析」の版間の差分
| 行264: | 行264: | ||
<br> | <br> | ||
| − | <div>もしくは[[参照付投影角度決定#演習に利用したMakefile| | + | <div>もしくは[[参照付投影角度決定#演習に利用したMakefile|こちらのMakefile]]から[[pdb2mrc]]を実行することもできます。<br> |
設定項目(今回は変更の必要はありません)</div> | 設定項目(今回は変更の必要はありません)</div> | ||
<pre> | <pre> | ||
| 行308: | 行308: | ||
=====3次元の概形が分からない場合===== | =====3次元の概形が分からない場合===== | ||
| − | <div> 概形が分からない場合は[[mrcImageModelCreate]] | + | <div> 概形が分からない場合は[[mrcImageModelCreate]]にて仮の3次元モデルデータを作成し、参照画像とします。今回は粒子を対象としているので、楕円体として参照画像を作成します。</div> |
<br> | <br> | ||
| − | |||
| − | |||
<div>コマンド [[mrcImageModelCreate]] -o Model-ellipsoidal.ref3d -m 7 -nx 20 -ny 20 -nz 20 -lx 1 -ly 1 -lz 1</div> | <div>コマンド [[mrcImageModelCreate]] -o Model-ellipsoidal.ref3d -m 7 -nx 20 -ny 20 -nz 20 -lx 1 -ly 1 -lz 1</div> | ||
<br> | <br> | ||
| 行342: | 行340: | ||
=== 3次元再構成=== | === 3次元再構成=== | ||
<div> 3次元再構成を行うためには多くの参照投影像が必要になりますので、[[Makefile]]を使用することをお薦めします。<br> | <div> 3次元再構成を行うためには多くの参照投影像が必要になりますので、[[Makefile]]を使用することをお薦めします。<br> | ||
| − | + | [[ROI]]ファイル(.roi)および参照画像(.ref3d)がある場合は[[参照付投影角度決定#演習に利用したMakefile|こちらのMakefile]]にて、'''make All'''と入力すれば、3次元再構成を1ターン分行い、3次元再構成されたファイル(.3d)が作成されます。<br> | |
以降の項目では3次元再構成に至るまでの各処理を[[Eos]]の[[コマンド]]や[[Makefile]]のコマンドと対応させながら、実行例を記述していきます。</div> | 以降の項目では3次元再構成に至るまでの各処理を[[Eos]]の[[コマンド]]や[[Makefile]]のコマンドと対応させながら、実行例を記述していきます。</div> | ||
<br> | <br> | ||
| 行445: | 行443: | ||
==== 最も類似度(相関値)の高い参照投影像の角度を粒子画像の投影角として決定==== | ==== 最も類似度(相関値)の高い参照投影像の角度を粒子画像の投影角として決定==== | ||
<div> [[mrcImageAutoRotationCorrelation]]を使用して粒子画像の投影角を決定します。</div> | <div> [[mrcImageAutoRotationCorrelation]]を使用して粒子画像の投影角を決定します。</div> | ||
| + | <br> | ||
| + | <div>[[Media:Input-121p-shiftr.2d-0000.roi|-i に使用するROIファイル]]の画像</div> | ||
| + | <table> | ||
| + | <tr> | ||
| + | <td>[[画像:Input-121p-shiftr-roi.png]]</td> | ||
| + | <td><p align="left">最小<br> | ||
| + | 最大<br> | ||
| + | 平均値<br> | ||
| + | 標準偏差<br> | ||
| + | 標準誤差<br></p> | ||
| + | </td> | ||
| + | <td><p align="left">-461.072 (17, 21, 0)<br> | ||
| + | 1098.8 (10, 13, 0)<br> | ||
| + | 28.9941<br> | ||
| + | 317.641<br> | ||
| + | 13.5938<br></p> | ||
| + | </td> | ||
| + | </tr> | ||
| + | </table> | ||
| + | <br> | ||
| + | |||
| + | <div>[[Media:121p-shift.ref2d|-r に使用する参照投影像]]</div> | ||
| + | <table> | ||
| + | <tr> | ||
| + | <td><p align="Center">[[画像:121p-shift2d.png]]<br> | ||
| + | z=0<br></p> | ||
| + | </td> | ||
| + | <td><p align="Center">[[画像:121p-shift2d1.png]]<br> | ||
| + | z=1<br></p> | ||
| + | </td> | ||
| + | <td><p align="Center">[[画像:121p-shift2d2.png]]<br> | ||
| + | z=2<br></p> | ||
| + | </td> | ||
| + | <td><p align="Center">[[画像:121p-shift2d3.png]]<br> | ||
| + | z=168<br></p> | ||
| + | </td> | ||
| + | <td><p align="left">サイズ<br> | ||
| + | 最小<br> | ||
| + | 最大<br> | ||
| + | 平均値<br> | ||
| + | 標準偏差<br> | ||
| + | 標準誤差<br></p> | ||
| + | </td> | ||
| + | <td><p align="left">( 64, 64, 169)<br> | ||
| + | -76.7146 (32, 41, 138)<br> | ||
| + | 2557.54 (35, 32, 131)<br> | ||
| + | 42.1574<br> | ||
| + | 207.727<br> | ||
| + | 0.249672<br></p> | ||
| + | </td> | ||
| + | </tr> | ||
| + | </table> | ||
| + | <br> | ||
| + | |||
| + | <div>コマンド [[mrcImageAutoRotationCorrelation]] -i Input-121p-shiftr.2d-0000.roi -r 121p-shift.ref2d </div> | ||
| + | <br> | ||
| + | |||
| + | <div></div> | ||
<br> | <br> | ||
2014年4月3日 (木) 08:25時点における版
単粒子解析法とは、Single Particle Analysis(SPA)の日本語訳である。単粒子とは、画像中の粒子(対象)が元来、2次元的にもしくは3次元的に同じ形をしていることを仮定して、その2次元、3次元構造解析を行う画像処理法のひとつである。
2次元単粒子解析
2次元単粒子解析とは、2次元で得られている電子顕微鏡投影像を分類し、それぞれを平均し、構造の違いを論じるための解析方法を指します。
3次元単粒子解析
元々の粒子の構造が3次元的に単一であることを仮定して、2次元の粒子画像群から3次元構造を再構成する画像処理法を指します。その手順は、下記のようになります。
ROI(粒子画像の抽出)
電子顕微鏡画像の前処理
以下の流れでCTF補正を行います。
電子顕微鏡画像(2D) ↓ A: mrcImageFFT 電子顕微鏡画像(2DFFT) ↓ B: ctfDisplay 電子顕微鏡画像(2DFFT) + CTF情報(ASCII) ↓ C: mrcImageCTFCompensation CTF補正済み画像(2D)
 |
最小 最大 |
-1066.31 (69, 47, 0) 489.111 (58, 48, 0) |
|
最小 最大 |
0.129048 (255, 128, 0) 772.6 (142, 252, 0) |
 実行直後 |
> |  lmax=400でトーンリングが見える |
> |  Rmax=0.08で拡大表示する |
> |  谷に線を合わせる(Defoucus=27000) |
最後にInfoのSaveボタンでCTF情報ファイルを保存します。(保存した出力データ)
 |
最小 最大 |
-461.072 (75, 56, 0) 1206.48 (16, 58, 0) |
電子顕微鏡画像から粒子画像の抽出
 |
最小 最大 |
-461.072 (75, 56, 0) 1206.48 (16, 58, 0) |
 実行直後 |
> |  mrcImage InfoのMax, Minの値を、 |
> |  切り出したい範囲を囲む |
 複数囲む場合はROI->MultiROIにチェック |
最小 最大 |
-461.072 (17, 21, 0) 1098.8 (10, 13, 0) |
最小 最大 |
-394.013 (18, 10, 0) 1206.48 (14, 12, 0) |
最小 最大 |
-418.302 (17, 6, 0) 1107.53 (14, 10, 0) |
参照画像の作成
参照画像の準備(mrcImageModelCreate, pdb2mrc etc.)
3次元の概形が分かっている場合
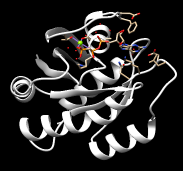 |
重心 最大半径 |
3.012034e-03 -4.102425e-03 7.530045e-04 3.286664e+01 |
###
### Initial model from PDB
###
REFSOURCE=121p-shift
DELTA=2.5
SIZE_X=64
SIZE_Y=64
SIZE_Z=64
START_X=`awk 'BEGIN { print -1*$(DELTA)*$(SIZE_X)/2}'`
START_Y=`awk 'BEGIN { print -1*$(DELTA)*$(SIZE_Y)/2}'`
START_Z=`awk 'BEGIN { print -1*$(DELTA)*$(SIZE_Z)/2}'`
最小 最大 |
0 (0, 0, 0) 22320.6 (34, 39, 32) |
3次元の概形が分からない場合
最小 最大 |
0 (0, 0, 0) 2 (10, 10, 1) |
3次元再構成
ROIファイル(.roi)および参照画像(.ref3d)がある場合はこちらのMakefileにて、make Allと入力すれば、3次元再構成を1ターン分行い、3次元再構成されたファイル(.3d)が作成されます。
参照画像から2次元の参照投影像のセットを生成
最小 最大 |
0 (0, 0, 0) 22320.6 (34, 39, 32) |
今回はファイル名のみ変更しています。
# Ref File Name INITIAL=121p-shift # # Search Area for 3D # ROTMODE=YOYS # # Search Area for 3D # ROTMODE=YOYS # Rot1 ROT1MIN=0 ROT1MAX=359 ROT1D=30 # Rot2 ROT2MIN=0 ROT2MAX=359 ROT2D=30 # Rot3 ROT3MIN=0 ROT3MAX=0 ROT3D=30
サイズ 最小 |
( 64, 64, 169) -76.7146 (32, 41, 138) |
最も類似度(相関値)の高い参照投影像の角度を粒子画像の投影角として決定
最小 最大 |
-461.072 (17, 21, 0) 1098.8 (10, 13, 0) |
サイズ 最小 |
( 64, 64, 169) -76.7146 (32, 41, 138) |
三次元像を再構成する
三次元像の分解能・質の確認
繰り返し(精密化)






